修回日期: 2012-10-12
接受日期: 2012-10-29
在线出版日期: 2012-11-08
目的: 基于磁珠分离的生物质谱技术, 建立食管癌诊断的血清学检测方法.
方法: 收集血清样本123例(其中食管癌63例, 正常人61例), 随机分为建模组和验证组. 运用弱阳离子纳米磁珠(magneticbead-weak cation exchange, MB-WCX)联合基质辅助激光解吸离子飞行质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry, MALDI-TOF-MS), 建立食管癌与正常人血清蛋白质谱. 用Flex Analysis 2.4软件收集数据, 应用Clinpro Tools 2.2软件对建模组32例食管癌患者和30例正常人血清差异蛋白质谱进行定量分析, 应用Support Vector Machine算法建立食管癌诊断模型, 应用所获取的诊断模型对验证组样本(31例食管癌和31例正常人)进行分类诊断, 以评价诊断模型的诊断价值.
结果: 通过比较分析食管癌与正常人血清蛋白质谱, 发现共有21个差异蛋白峰(P<0.05), 其中在食管癌中表达上调12个, 表达下调9个, 利用其中3个差异峰(Mr分别为654.74、1 451.48、1 866.67 Dr)建立诊断模型, 获得了93.75%(30/32)的敏感性和90.00%(27/30)的特异性, 经独立样本双盲验证, 其灵敏度为93.55%(29/31), 特异度为90.32%(28/31).
结论: 基于磁珠分离和MALDI-TOF-MS技术能直接检测出食管癌患者血清差异表达蛋白, 建立的诊断模型具有较高的敏感性和特异性, 对提高食管癌的诊断具有一定的临床意义.
引文著录: 刘清银, 孙良起, 涂斌. 基于磁珠分离的生物质谱技术用于食管癌临床诊断. 世界华人消化杂志 2012; 20(31): 3021-3026
Revised: October 12, 2012
Accepted: October 29, 2012
Published online: November 8, 2012
AIM: To detect specific protein biomarkers for esophageal cancer by comparing serum proteomic spectra between patients with esophageal cancer and healthy individuals.
METHODS: Serum samples were collected from 63 patients with esophageal cancer and 62 healthy individuals, and randomized into model construction group and validation group. WCX kit and matrix-assisted laser desorption/ ionization time-of-flight mass spectrometry (MALDI-TOF-MS) technology were used to detect serum samples from patients with esophageal cancer and healthy individuals. Flex Analysis software was used to analyze protein peaks for significant difference. Mass spectrometry data obtained were analyzed using ClinPro Tools software. The model was then built using ClinproTools2.2 software and evaluated in a blind test for reliability.
RESULTS: Twenty-four differentially expressed proteins in serum were screened by comparing serum protemic spectra between esophageal cancer patients and healthy individuals, including 12 up-regulated proteins and 9 down-regulated ones. Three proteins (654.74, 1 451.48 and 1 866.67 Dr) were obtained for developing a ClinProt model which was able to classify esophageal cancer patients and healthy individuals with a sensitivity of 93.75% (30/32) and a specificity of 90.00% (27/30). In a double blind validation, the ClinProt model yielded a sensitivity of 93.55% (29/31) and a specificity of 90.32% (28/31).
CONCLUSION: MALDI-TOF-MS combined with WCX kit technology allows directly screening differentially expressed serum protein in esophageal cancer, and these protein markers can be used for diagnosis of early esophageal cancer.
- Citation: Liu QY, Sun LQ, Tu B. Magnetic beads-based biological mass spectrometry technology for diagnosis of esophageal carcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(31): 3021-3026
- URL: https://www.wjgnet.com/1009-3079/full/v20/i31/3021.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i31.3021
食管癌是常见恶性肿瘤之一[1]. 在我国, 食管癌的发病率呈上升趋势, 在我国北方具有很高的发病率和死亡率, 与肺癌、胃癌、肝癌等各种恶性肿瘤一起, 成为中国人口的主要死因[2]. 早期患者及时手术治疗后5年生存率可到90%以上, 但中晚期患者即使行以手术为主的综合治疗, 仍有半数以上患者死于局部复发或转移[3]. 目前, 食管癌诊断技术虽然大有提高, 但仍有半数以上患者在首次就诊时已属中晚期, 因此同其他恶性肿瘤一样, 早期诊断是提高患者生存率和生活质量的关键[3]. 本研究利用WCX纳米磁珠联合基质辅助激光解析离子化飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry, MALDI-TOF-MS)技术对食管癌患者及健康志愿者血清蛋白质谱的变化进行分析对比, 旨在发现可用于食管癌早期临床诊断的新方法, 以期提高早期诊断水平, 改善预后.
本项目研究得到我院伦理委员会认证, 所纳入病例及健康志愿者知情同意后行标本采集和相关检查. 本组食管癌患者63例, 术后经病理诊断为食管癌, 其中TNMⅠ期16例, TNMⅡ期47例, 平均年龄为(48.0±3.6)岁, 术前经B超等影像学检查, 除外肝、骨转移. 同时, 收集健康志愿者血清61例, 年龄与食管癌组差异无统计学意义, 经体格检查、胃镜检查等除外食管占位性病变. 以上血清标本均由解放军150中心医院提供. 按照完全随机化的方法, 将食管癌患者与健康志愿者分为建模组与验证组. 其中, 建模组包括食管癌32例(其中TNMⅠ期8例, TNMⅡ期24例)与健康志愿者30例, 验证组包括健康志愿者与食管癌各31例(其中TNMⅠ期8例, TNMⅡ期23例).
采集志愿者清晨空腹静脉全血5 mL(不加抗凝剂), 室温静置30 min后, 以2 000 r/min离心15 min, 将上层血清取出并分装, 冻存于-80 ℃冰箱. 实验时, 样品常温解冻, 待用. 食管癌患者于术前当日清晨采集.
基质辅助激光解析离子化飞行时间质谱仪(matrix-assisted laser desorption ionization time-of-flight mass spectrum, MALDI-TOF-MS, Autoflex-Ⅲ)、弱阳离子纳米磁珠(magneticbead-weak cation exchange, MB-WCX)及其缓冲液体系、α-氰基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic acid, HCCA)和混合标肽均购自Bruker公司. 乙腈(acetonitrile, ACN)、三氟乙酸(trifluoroacetic acid, TFA)、SPA(sinapinic acid)、尿素、DTT、CHAPS、Tris-HCl、NaAC等购自美国Sigma公司.
1.2.1 WCX纯化多肽: (1)4 ℃冰箱取出磁珠试剂盒, 取出弱阳离子磁珠悬浮液一管, 手动上下摇动, 完全混匀磁珠悬浮液, 1 min; (2)取出10 μL磁珠结合缓冲液(binding solution, BS)加入200 μL样品管中, 再加入10 μL磁珠及5 μL血清, 用加样枪上下吸打混匀, 避免产生气泡; (3)室温静置5 min, 将样品管放入磁珠分离器, 使磁珠贴壁1 min, 磁珠与悬浮的液体分离, 用加样枪吸去悬浮的液体; (4)加入100 μL磁珠清洗缓冲液(washing solution, WS), 在磁珠分离器前后相邻两孔间反复移动样品管10次, 使样品管在磁珠分离器上静置, 磁珠贴壁, 用加样枪吸去悬浮的液体, 枪头应避免接触到磁珠, 避免吸走磁珠, 重复本步骤2次; (5)从磁珠分离器上取下样品管, 并向样品管中加入5 μL磁珠洗脱缓冲液(eluting solution, ES), 混匀贴壁的磁珠, 反复吸打10次, 将样品管放入磁珠分离器, 磁珠贴壁2 min, 磁珠与悬浮的液体充分分离后, 将上清液移入干净的0.5 mL样品管(先在管内加入5 μL稳定缓冲液(SS), 用加样枪小心吸打混匀); (6)先点1 μL洗脱样品, 室温放干后, 再点1 μL基质(3 mg/mL CHCA, 50%ACN, 2%TFA), 室温放干, 准备质谱分析.
1.2.2 质谱检测与数据采集: 采用Autoflex-Ⅲ MALDI-TOF-MS仪器, 应用线性模式, 采集范围为相对分子量范围600-18 000. 应用Flex Analysis 2.4软件收集数据, ClinproTools 2.2软件进行分析, 包括质谱图的平滑、衰减及标峰处理, 并输出含有质荷比值和峰强度的Excel格式数据, 软件自动对数据进行统计学处理, 以P<0.05为差异有统计学意义. ClinproTools 2.2分析建模组样本质谱图, 采用支持向量机(Support Vector Machine, SVM), 计算灵敏度与特异度. 用诊断模板对验证组样本进行分组, 以评价诊断模板的诊断价值.
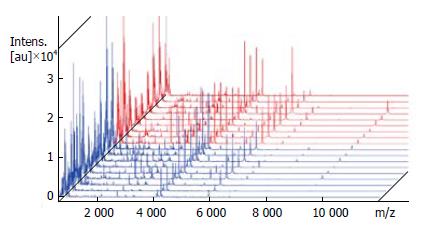
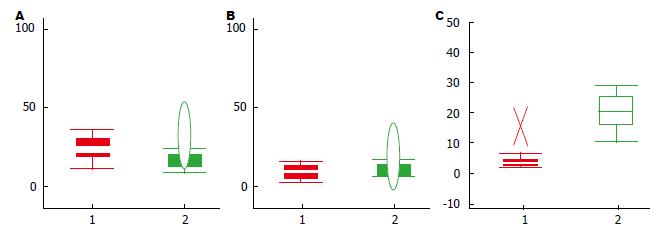
建立建模组样本(食管癌患者32例, 正常人30例)血清蛋白质谱, 在分子质量600-18 000内共检测到70个蛋白峰, 代表性蛋白质谱见图1. 比较分析食管癌与健康志愿者之间差异蛋白质谱, 共发现21个蛋白峰有显著性差异(P<0.05). 其中在食管癌中表达上调峰12个, 表达下调峰9个(表1). 以m/z 654.74, 1 451.48及1 866.67 Dr 3个差异蛋白峰为例, 从图2可见, 这3个质谱峰分别呈明显上调和下调趋势.
| 差异蛋白质荷比(m/z) | 峰曲线下面积(mean±SD) | P值 | 在食管癌组的表达变化 | |
| 食管癌组 | 健康志愿者 | |||
| 698.68 | 104.08±27.41 | 72.52±25.72 | 0.00371000 | ↑ |
| 622.97 | 53.17±26.88 | 29.98±16.06 | 0.00603000 | ↑ |
| 622.40 | 52.81±27.77 | 33.17±17.59 | 0.02160000 | ↑ |
| 676.68 | 37.73±23.13 | 13.70±7.97 | 0.00000173 | ↑ |
| 909.79 | 25.42±12.59 | 37.24±13.12 | 0.01160000 | ↓ |
| 654.74 | 25.38±7.21 | 16.61±6.10 | 0.00125000 | ↑ |
| 1505.35 | 16.62±11.93 | 30.86±15.98 | 0.01160000 | ↓ |
| 720.86 | 14.31±5.39 | 21.96±7.22 | 0.00323000 | ↓ |
| 894.11 | 12.02±6.61 | 21.40±7.48 | 0.00121000 | ↓ |
| 1618.31 | 11.35±8.26 | 19.21±14.36 | 0.03160000 | ↓ |
| 1451.48 | 9.17±3.25 | 11.85±3.68 | 0.02170000 | ↓ |
| 3193.32 | 7.86±5.49 | 4.02±2.47 | 0.02340000 | ↑ |
| 1546.39 | 7.18±4.85 | 10.39±3.07 | 0.02250000 | ↓ |
| 1246.07 | 6.93±3.71 | 10.62±4.90 | 0.03940000 | ↓ |
| 1866.67 | 6.57±4.42 | 13.23±7.36 | 0.00121000 | ↓ |
| 2770.63 | 6.43±4.65 | 2.36±1.54 | 0.00125000 | ↑ |
| 2933.95 | 5.01±3.97 | 1.98±0.99 | 0.00518000 | ↑ |
| 5340.24 | 2.12±2.45 | 0.54±0.75 | 0.04870000 | ↑ |
| 7776.13 | 0.84±1.00 | 0.35±0.52 | 0.02300000 | ↑ |
| 5963.74 | 0.76±0.77 | 0.23±0.35 | 0.01720000 | ↑ |
| 4645.47 | 0.46±0.38 | 0.23±0.23 | 0.02250000 | ↑ |
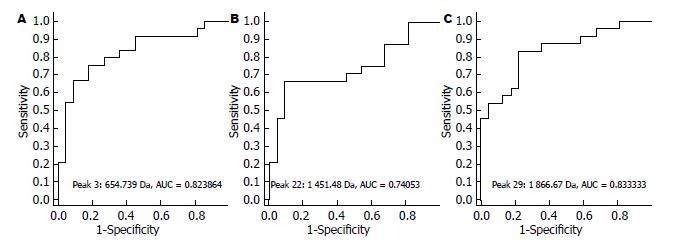
利用ClinproTools 2.2中支持向量机算法建立了由上述3个差异表达蛋白(Mr分别为654.74, 1 451.48及1 866.67 Dr)组成的诊断模型, 获得了93.75%(30/32)的敏感性和90.00%(27/30)的特异性, 正确率为91.94%. 分子量为654.74, 1 451.48及1 866.67的3个蛋白诊断食管癌的ROC曲线下面积分别为0.82、0.74及0.83(图3).
为进一步评价本研究中所建立诊断模板的诊断价值, 我们将利用诊断模板对验证组样本进行双盲验证, 结果显示其灵敏度为93.55%(29/31), 特异度为90.32%(28/31), 约登指数为0.84. 其中, 对验证组中8例早期患者(TNMⅠ期)诊断的灵敏度为87.50%(7/8).
食管癌为常见的恶性肿瘤之一, 据统计我国食管癌发病率有升高趋势, 严重威胁着人们的身心健康. 患者如获得早期诊断和治疗, 5年生存率将达到90%, 遗憾的是, 大部分患者在确诊时已处于进展期或已有转移, 错过了极佳的治疗窗口期. 其原因是无特异性高的早期诊断方法, 因此从血清中寻求特异生物标志物用于食管癌的诊断是临床急需解决的难题. 食管癌早期诊断方法如食管拉网细胞学检查等均不同程度地存在敏感性与特异性不高的问题. 标志物检测由于具有侵入性小、易于检测等优点成为当前研究的热点, 但目前临床应用较多的食管癌相关性肿瘤标志物有CEA、 CYFRA21-1、SCC-Ag、CA19-9、CA125、CA724等, 在疾病临床诊断中应用价值有限, 从血清中寻找能用于食管癌早期诊断的敏感性和特异性高的肿瘤标志物已是亟待解决的问题[4].
食管癌的发生、发展是一个多步骤、多因素参与的复杂过程, 这个过程是多通路级联反应, 其中包含了无数蛋白质的变化, 很多变化同时也在外周血中有所体现. 因而通过分析血清蛋白组可以发现许多有潜在价值的肿瘤标志物, 分析这些蛋白质的变化, 可以帮助我们发现特异性肿瘤标志物, 来诊断食管癌及监测病情的变化, 甚至可能找到治疗的靶点.
蛋白质组最早由澳大利亚Wiliams和Wilkins于1994年提出, 指的是"一个细胞或组织所表达的全部蛋白质". 蛋白质组学是以蛋白质组为研究对象, 从整体水平分析细胞内动态变化的蛋白质组成成分、表达水平与修饰状态, 以及蛋白质之间的相互作用与联系, 揭示蛋白质功能, 从蛋白质水平进一步认识生命活动的机理和疾病发生机制[5,6]. 目前已广泛用于疾病标志物的筛查[5,7-11]. MALDI-TOF-MS是目前蛋白质组学研究的核心技术, 具有高灵敏度、高通量、重复性好等优点, 纳米磁珠可以根据蛋白质组分的理化性质不同对蛋白质进行分离提纯, 具有较好的重复性, 两者结合为疾病蛋白组研究提供了有力的工具, 目前以用于肾移植、风湿类疾病、酒精中毒、乳腺癌等标志物的研究[12-15].
本研究采用纳米磁珠联合MALDI-TOF-MS技术对32例食管癌患者和30例健康志愿者进行了血清蛋白质谱对比分析研究, 结果发现了有68个蛋白质峰具有判断性差别, 其中有21个蛋白质峰具有显著性差异. 将两组血清蛋白质谱建立数据库, 利用ClinProt Tool统计软件对数据库进行分析, 确立了以m/z为654.74、1 451.48、1 866.67 Dr等3个蛋白质所组成的模板, 能正确地将食管癌患者与正常人分组, 正确分组率达91.94%. 为进一步评价诊断模板的诊断价值, 我们将收集的另外一组样本作为验证组, 由建立的诊断模板对验证组样本在双盲条件下进行分类, 结果显示, 该模板可以将31例食管癌中的29例正确分组, 31例正常人中的28例进行正确分组, 其灵敏度和特异度分别为93.55%(29/31), 特异度为90.32%(28/31), 约登指数为0.84, 具有较高的诊断价值. 有趣的是, 该诊断模版可以将87.50%(7/8)的早期患者(TNMⅠ期)正确诊断, 说明在早期诊断中具有较好的应用价值. 尽管如此, 但由于样本量较小, 尤其是早期患者的样本量较小, 因此有待于扩大样本之后进一步验证其临床应用价值.
本研究中, 发现了一组对食管癌早期诊断有价值的蛋白质/多肽, 对其进行鉴定和功能分析将有助于阐明食管癌发生、发展机制, 但由于技术上的局限性, 此部分研究尚未取得理想结果. 因此, 不断地采用新技术、新方法对差异蛋白进行鉴定和功能描述也是我们今后的研究方向.
总之, 我们采用WCX磁珠结合MALDI质谱对食管癌血清蛋白质谱进行了研究, 发现了一组差异蛋白质, 并建立了具有较高灵敏度和特异度的诊断模板. 所发现的差异蛋白为食管癌发生、发展研究提供了新的线索, 所建立的诊断模板为食管癌的诊断提供了新的有效的方法, 对提高诊断水平具有重要意义.
食管癌是我国常见的恶性肿瘤之一, 其预后差的主要原因在于缺乏高灵敏度、特异度的早期诊断方法. 蛋白质组学具有高通量、高灵敏度等技术优势, 为肿瘤早期诊断的研究开拓了新的视野. 开展食管癌早期诊断的蛋白质组学研究具有重要的临床意义.
吕宾, 教授, 浙江中医药大学附属医院(浙江省中医院)
应用磁珠分离结合基质辅助激光解吸离子飞行质谱(MALDI-TOF-MS)检测进行比较蛋白质组学研究, 可以发现差异蛋白, 通过软件分析, 建立诊断模板, 对疾病的筛查有重要价值, 是疾病蛋白质组学研究的热点, 但由于所发现的有诊断价值的多肽多是低丰度、小分子量蛋白, 其鉴定是一项具有挑战性的工作.
磁珠结合MALDI-TOF-MS技术已应用于结直肠癌、乳腺癌、类风湿等疾病诊断研究, 通过软件分析, 建立了具有较高诊断价值的诊断模型.
利用磁珠结合MALDI-TOF-MS技术进行食管癌的比较蛋白质组学研究, 发现了多个有意义的蛋白, 通过软件分析建立了诊断模板, 通过双盲验证, 评价了诊断模型的临床应用价值. 科研设计严密, 结果符合统计学要求.
可用于食管癌的初步筛查, 为血清标志物的发现提供了目标蛋白.
MALDI-TOF-MS: 激光解析离子化飞行时间质谱, 与蛋白质分离纯化技术(磁珠)相结合, 经配套软件对结果定量分析, 发现疾病差异蛋白, 建立诊断模板, 并可利用此模板对疾病进行筛查诊断, 具有高通量、高灵敏度等技术优势.
本研究设计合理, 对临床医师有一定的参考价值.
编辑: 李军亮 电编:鲁亚静
| 1. | Jemal A, Bray F, Center MM, Ferlay J, Ward E, Forman D. Global cancer statistics. CA Cancer J Clin. 2011;61:69-90. [PubMed] [DOI] |
| 3. | Smith RA, Cokkinides V, Brooks D, Saslow D, Shah M, Brawley OW. Cancer screening in the United States, 2011: A review of current American Cancer Society guidelines and issues in cancer screening. CA Cancer J Clin. 2011;61:8-30. [PubMed] [DOI] |
| 4. | Bird-Lieberman EL, Fitzgerald RC. Early diagnosis of oesophageal cancer. Br J Cancer. 2009;101:1-6. [PubMed] [DOI] |
| 5. | Issaq HJ, Xiao Z, Veenstra TD. Serum and plasma proteomics. Chem Rev. 2007;107:3601-3620. [PubMed] [DOI] |
| 6. | Phizicky E, Bastiaens PI, Zhu H, Snyder M, Fields S. Protein analysis on a proteomic scale. Nature. 2003;422:208-215. [PubMed] [DOI] |
| 7. | Fan NJ, Gao CF, Wang CS, Zhao G, Lv JJ, Wang XL, Chu GH, Yin J, Li DH, Chen X. Identification of the up-regulation of TP-alpha, collagen alpha-1(VI) chain, and S100A9 in esophageal squamous cell carcinoma by a proteomic method. J Proteomics. 2012;75:3977-3986. [PubMed] [DOI] |
| 8. | Fan NJ, Gao CF, Wang CS, Lv JJ, Zhao G, Sheng XH, Wang XL, Li DH, Liu QY, Yin J. Discovery and verification of gelsolin as a potential biomarker of colorectal adenocarcinoma in the Chinese population: Examining differential protein expression using an iTRAQ labelling-based proteomics approach. Can J Gastroenterol. 2012;26:41-47. [PubMed] |
| 9. | Ray S, Reddy PJ, Choudhary S, Raghu D, Srivastava S. Emerging nanoproteomics approaches for disease biomarker detection: a current perspective. J Proteomics. 2011;74:2660-2681. [PubMed] [DOI] |
| 10. | Lehtiö J, De Petris L. Lung cancer proteomics, clinical and technological considerations. J Proteomics. 2010;73:1851-1863. [PubMed] [DOI] |
| 11. | McDonnell LA, Corthals GL, Willems SM, van Remoortere A, van Zeijl RJ, Deelder AM. Peptide and protein imaging mass spectrometry in cancer research. J Proteomics. 2010;73:1921-1944. [PubMed] [DOI] |
| 12. | Sui W, Huang L, Dai Y, Chen J, Yan Q, Huang H. Proteomic profiling of renal allograft rejection in serum using magnetic bead-based sample fractionation and MALDI-TOF MS. Clin Exp Med. 2010;10:259-268. [PubMed] [DOI] |
| 13. | Dai Y, Hu C, Wang L, Huang Y, Zhang L, Xiao X, Tan Y. Serum peptidome patterns of human systemic lupus erythematosus based on magnetic bead separation and MALDI-TOF mass spectrometry analysis. Scand J Rheumatol. 2010;39:240-246. [PubMed] [DOI] |
| 14. | Sogawa K, Satoh M, Kodera Y, Tomonaga T, Iyo M, Nomura F. A search for novel markers of alcohol abuse using magnetic beads and MALDI-TOF/TOF mass spectrometry. Proteomics Clin Appl. 2009;3:821-828. [PubMed] [DOI] |
| 15. | Fan NJ, Gao CF, Zhao G, Wang XL, Liu QY. Serum peptidome patterns of breast cancer based on magnetic bead separation and mass spectrometry analysis. Diagn Pathol. 2012;7:45. [PubMed] [DOI] |