修回日期: 2012-05-26
接受日期: 2012-08-11
在线出版日期: 2012-09-08
目的: 分析胃癌组织中miR-191的表达及预测其靶基因, 并检测靶基因在胃癌组织中的表达.
方法: 胃癌及配对癌旁正常组织标本50例, 应用基于SYBR GreenⅠ的实时荧光定量PCR(qRT-PCR)检测miR-191的表达情况, 后用2-∆∆CT方法进行相对定量分析. 利用生物信息学软件对miR-191进行靶基因预测. 用免疫组织化学检测靶基因在胃癌组织中的表达情况, 并分析其与miR-191表达情况的关系.
结果: miR-191在胃癌组织中的表达显著高于癌旁正常组织[0.0314(0.0037-0.4924) vs 0.0240(0.0037-0.1593), P<0.05], miR-191与临床病理特征(性别、年龄、病理组织分型、淋巴结转移和TNM分期)无显著相关性. 对miR-191预测得到磷脂酶C-delta 1(phospholipase C-delta 1, PLCD1)、SOX4等9个靶基因, 其中SOX4和NDST1被证明是miR-191的靶基因. PLCD1在胃癌组织中的表达显著低于癌旁正常组织(56.7% vs 96.7%, P<0.01), 且其与miR-191在胃癌中的表达呈负相关(90% vs 30%, r = -0.639, P<0.01).
结论: miR-191在胃癌组织中呈高表达, 可能通过调控PLCD1对胃癌的发病起重要作用.
引文著录: 郭鹏辉, 杜燕蕾, 聂玉强. miR-191在胃癌组织中的表达及其靶基因的预测. 世界华人消化杂志 2012; 20(25): 2347-2352
Revised: May 26, 2012
Accepted: August 11, 2012
Published online: September 8, 2012
AIM: To investigate the expression of miR-191 and its predicted target genes in gastric carcinoma.
METHODS: The relative expression of miR-191 in gastric carcinoma and tumor-adjacent tissue was quantified by qRT-PCR. The potential target genes of miR-191 were predicted using bioinformatic software. Expression of target genes in gastric carcinoma and tumor-adjacent tissue was detected by immunohistochemistry. The association between miR-191 expression and target gene expression was analyzed.
RESULTS: The expression of miR-191 was significantly higher in gastric carcinoma than in tumor-adjacent tissue [0.0314 (0.0037-0.4924) vs 0.0240 (0.0037-0.1593), P < 0.05]. MiR-191 expression was not related with age, gender, histological type, lymph node metastasis or clinical stage. Nine target genes were predicted, including SOX4 and NDST1, both of which had been confirmed as direct targets of miR-191. The expression of phospholipase C-delta 1 (PLCD1) was significantly up-regulated in gastric carcinoma compared to tumor-adjacent tissue (56.7% vs 96.7%, P < 0.01). MiR-191 expression was significantly inversely correlated with PLCD1 expression in gastric carcinoma (90% vs 30%, r = -0.639, P < 0.01).
CONCLUSION: The expression of miR-191 is up-regulated in gastric carcinoma. MiR-191 may play a pivotal role in the pathogenesis of gastric carcinoma by regulating PLCD1 expression.
- Citation: Guo PH, Du YL, Nie YQ. Expression of miR-191 and its potential target genes in gastric carcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(25): 2347-2352
- URL: https://www.wjgnet.com/1009-3079/full/v20/i25/2347.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i25.2347
胃癌是最常见的恶性肿瘤之一, 在我国每年因胃癌死亡的患者占死于消化系肿瘤患者的首位[1], 但其发病机制尚未清楚. 随后, 近年研究表明, miRNAs在肿瘤的发生发展中起类似于癌基因或抑癌基因的作用[2]. 研究者利用基因芯片技术检测出miR-191在多种肿瘤中呈异常表达[3], 但其在胃癌中的表达及其与胃癌临床病理的关系所知尚少, 而且, miR-191在胃癌中是通过哪个靶基因发挥作用更是尚未有相关报道. 本实验应用qRT-PCR技术对miR-191在胃癌及配对癌旁正常组织中的表达情况进行检测, 并进行靶基因预测, 分析miR-191与靶基因的表达相关性, 初步探讨miR-191在胃癌发病中的作用, 并为进一步的功能实验提供依据.
选取2009-04/2010-12在广州市第一人民医院外科切除的胃癌患者50例, 其中男32例, 女18例, 年龄范围33-80岁, 平均年龄59岁±11.9岁. 所有患者术前均未进行过放疗及化疗. 取胃癌及距癌肿5 cm以上的正常组织标本置于液氮速冻后储存于-80 ℃冰箱. 所有癌及癌旁正常组织均经过术后病理证实, 癌组织中高分化腺癌4例、中分化腺癌21例、低分化腺癌21例、黏液腺癌4例, 其中39例有淋巴结转移. 根据Lauren分型[4], 其中肠型27例、弥散型23例. TNM分期根据美国癌症联合委员会(AJCC)方案[5]: Ⅰ期5例, Ⅱ期17例, Ⅲ期25例, Ⅳ期3例.
1.2.1 总RNA的提取: 应用TRIzol试剂对50例胃癌及癌旁正常组织进行总RNA抽提(具体方法根据试剂说明书). 用紫外分光光度计(Amersham Ultrospec 2001)检测总RNA的浓度和纯度, 所有标本RNA吸光度A260/280均为1.8-2.1, 表明总RNA纯度好. 用1.2%琼脂糖电泳检测总RNA的完整性, 28S条带亮度约为18S两倍则表明总RNA完整性好.
1.2.2 总RNA的逆转录: 应用逆转录试剂盒miR CURY LNATM Universal cDNA Synthesis Kit(Exiqon公司, 丹麦)对总RNA进行逆转录. 根据试剂盒说明书, 将所有标本的总RNA均稀释成终浓度为1.42-1.79 μg/L, 然后配成20 μL反应液: 5×Reaction buffer 4 μL、Enzyme mix 2 μL、模板总RNA(稀释后)14 μL. 反应条件: 42 ℃ 60 min, 95 ℃ 5 min.
1.2.3 miR-191的实时荧光定量PCR检测: 应用实时定量PCR试剂盒miRCURY LNATM SYBR® Green master mix Universal RT(Exiqon公司, 丹麦)和miR-191及内参U6引物(LNATM PCR primer set, Exiqon公司, 丹麦), 在MJ Research DNA Engine Opticon 2荧光定量PCR仪进行检测. 根据试剂说明书配成20 μL反应液: 模板cDNA 8 μL、SYBR® Green master mix 10 μL、miR-191/内参U6引物2 μL. 反应条件: 预变性95 ℃ 10 min; 95 ℃ 10 s、60 ℃ 1 min, 40个循环. 每个标本设3个复孔. 实验得到miR-191及U6在各标本中的CT值, 以U6内参校正, 用2-∆∆CT相对定量法比较miR-191在胃癌及配对正常组织的表达量, 2-∆∆CT = 2-(CT癌miR-191-CT癌U6)-(CT癌旁miR-191-CT癌旁U6), 若2-∆∆CT值>1.0认为表达增高, <1.0认为表达降低, = 1.0认为表达不变.
1.2.4 靶基因预测: 应用MiRanda[6]、TargetScan[7]及PicTar[8]3个生物信息学软件对miR-191进行靶基因预测, 取3个软件均预测到的基因作为靶基因.
1.2.5 靶基因的免疫组织化学: 在上述50例病例中取30例进行免疫组织化学实验. 兔抗人磷脂酶C-delta 1(phospholipase C-delta 1, PLCD1)多克隆抗体(购自美国Santa Cruz Biontechnology公司)按1:50进行稀释, 应用二抗试剂盒(Dako REALTM EnVisionTM Detection Systems Peroxidase/DAB, Rabbit/Mouse购自丹麦Dako公司)进行EnVision二步法检测. 用PBS代替一抗作为阴性对照. 每张切片随机选取10个高倍视野(×400)进行观察. 评分标准为[9], 依据着色强度评分: 无着色为0分, 淡黄色为1分, 棕黄色为2分, 褐黄色为3分; 依据显示细胞率评分: 无着色为0分, ≤25%为1分, 26%-50%为2分, 51%-75%为3分, >75%为4分. 两者相加得综合评分: 0分为(-), 2-3分为(±), 4-5分为(+), 6-7分为(++). 最后(-)和(±)判为阴性, (+)和(++)判为阳性.
统计学处理 数据统计学分析利用SPSS17.0进行, 胃癌及癌旁正常组织的相对表达量采用配对样本Wilcoxon秩和检验(双侧)分析, 并应用χ2检验(双侧)分析不同的临床病理特征与miR-191表达情况的关系. 用配对样本Wilcoxon秩和检验(双侧)分析胃癌组织与配对癌旁正常组织PLCD1表达水平的差异. 用Spearman相关分析方法分析miR-191和PLCD1在胃癌组织中表达水平(与癌旁正常组织相比)的相关性. 所有的统计分析中, P<0.05表示有统计学意义.
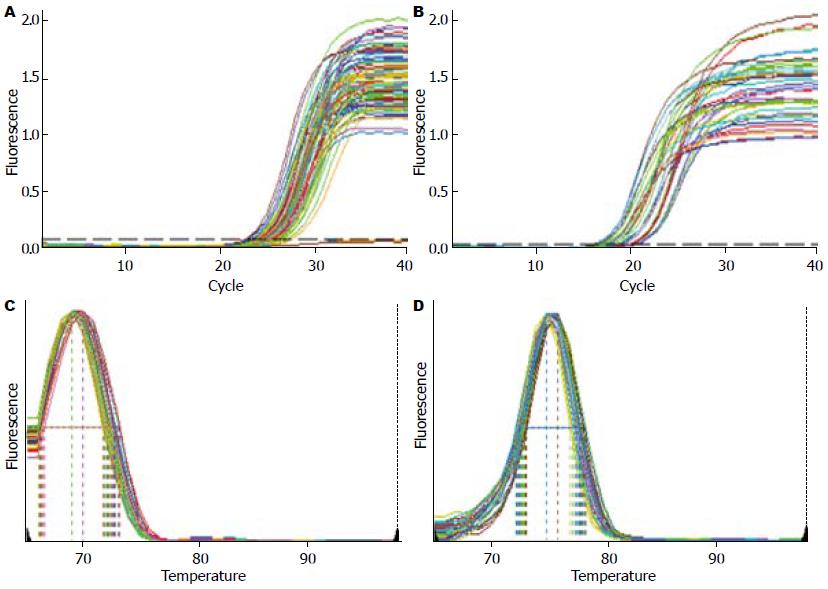
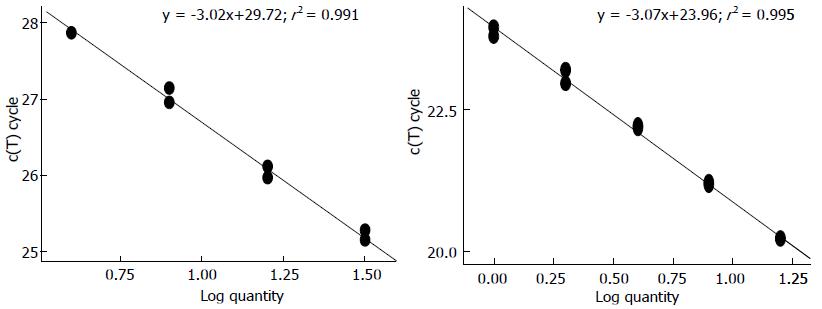
在本实验中, miR-191及U6的扩增曲线均呈典型S型曲线(图1A, B); 融解曲线呈单峰且融解温度均一(图1C, D); qRT-PCR产物电泳图可见单一条带, 无杂带. 实验以U6为内参做校正, 与配对的正常组织相比, 68%(34/50)胃癌组织的miR-191相对表达量呈高表达. miR-191及U6的标准曲线的扩增效率(E)分别为114.4%和111.7%, 两扩增效率相近并接近100%(图2A, B), 应用2-∆∆CT进行相对定量分析, miR-191在胃癌组织中的表达量是配对正常组织的2.01倍, 有显著性差异(P<0.05).
应用χ2检验(双侧)分析不同临床病理学参数与miR-191表达间的关系, 结果显示均无显著差异性(P>0.05, 表1).
| 临床病理学参数 | n | 胃癌组织中miR-191表达 | P值 | |
| 高表达(n = 34) | 低表达(n = 16) | |||
| 性别 | 0.434 | |||
| 男 | 32 | 23 | 9 | |
| 女 | 18 | 11 | 7 | |
| 年龄(岁) | 0.121 | |||
| <60 | 28 | 16 | 12 | |
| ≥60 | 22 | 18 | 4 | |
| 组织学分型 | 0.603 | |||
| 高分化腺癌 | 4 | 2 | 2 | |
| 中分化腺癌 | 21 | 14 | 7 | |
| 低分化腺癌 | 21 | 16 | 5 | |
| 黏液腺癌 | 4 | 2 | 2 | |
| Lauren分型 | 0.408 | |||
| 肠型 | 27 | 17 | 10 | |
| 弥散型 | 23 | 17 | 6 | |
| 淋巴结转移 | 0.725 | |||
| 有 | 39 | 27 | 12 | |
| 无 | 11 | 7 | 4 | |
| TNM分期 | 0.525 | |||
| Ⅰ期-Ⅱ期 | 22 | 16 | 6 | |
| Ⅲ期-Ⅳ期 | 28 | 18 | 10 | |
应用生物信息学软件进行miR-191的靶基因预测, 有9个基因在3个软件中均预测到, 包括PLCD1、SOX4、RNF139、FZD5、GAP43、TJP1、TMOD2、NDST1和AMMECR1.
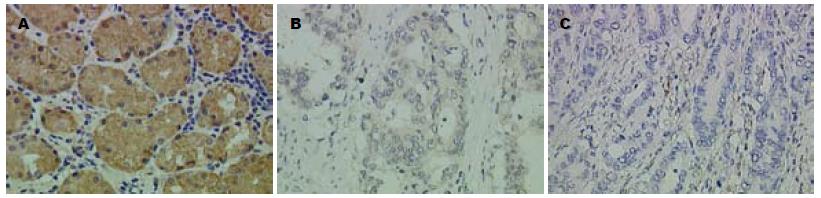
对30例胃癌组织及配对癌旁正常组织进行免疫组织化学检测, 发现PLCD1均在上皮细胞的胞浆染色表达, 阳性细胞的胞浆DAB染色呈棕黄色(图3A). 96.7%(29/30)癌旁正常组织中的PLCD1表达呈(+)或(++), 而胃癌组织中仅56.7%(17/30)表达(+)或(++), 差异有统计学意义(P<0.01, 表2). 与配对正常组织相比, 70%(21/30)胃癌组织中的PLCD1表达强度下降或阴性(图3B, C); 有90%(18/20)miR-191表达增高的胃癌组织中的PLCD1表达强度下降, 而只有30%(3/10)miR-191表达降低的胃癌组织中的PLCD1表达强度下降. 再对miR-191与PLCD1在胃癌组织中表达水平进行相关性分析, 结果显示两者呈显著负相关(r = -0.639, P<0.01).
| 分组 | PLCD1表达强度 | P值 | |||
| - | ± | + | ++ | ||
| 胃癌组织 | 8 | 5 | 15 | 2 | 0.000 |
| 癌旁正常组织 | 0 | 1 | 9 | 20 | |
miRNAs是一类内源性的保守的非编码的小分子双链RNA, 是目前研究较多的ncRNA[10], 其大小长约20-25个核苷酸, 成熟的miRNAs主要与靶基因mRNA的3'非翻译区完全或不完全互补使mRNA降解或翻译的抑制[11]. 自1993年, Lee等[12]在线虫中发现了第一个miRNA: Lin-4, 人类基因组内发现的miRNAs己经超过1 000个, 并推测其可能调控基因组内1/3左右基因的表达[13,14]. miRNA与肿瘤有着密切关系, 参与了肿瘤的生成、发展和转归过程[15]. 目前已有不少研究表明, 大量miRNAs在胃癌中呈现差异性表达[16-18]. 本研究发现: 与配对癌旁正常组织相比, miR-191在胃癌组织中的表达显著升高, 但其表达与性别、年龄、病理组织学分型、淋巴结转移和TNM分期无相关性, 利用生物信息学软件对miR-191进行靶基因预测筛选出PLCD1、SOX4(sex determining region Y-box 4)等9个靶基因.
Volinia等[3]利用基因芯片技术对363个实体癌标本(乳腺癌、结肠癌、肺癌、胰腺癌、前列腺癌、胃癌)及177个正常组织标本进行分析, 其中miR-191在5种实体癌中呈高表达. Ueda等[19]同样应用基因芯片技术对184例胃癌组织及169例非癌胃黏膜组织进行分析, 发现包括miR-191等22个miRNAs在胃癌中高表达, 13个miRNAs低表达; 而且在2种不同组织类型(肠型和弥散型)的胃癌中miRNA的异常表达是不一样的. 国内西京医院也发现miR-191在胃癌中高表达[20]. miR-191在肝细胞癌中高表达, 抑制miR-191可以减缓细胞增殖和诱导细胞凋亡, 在老鼠模型中有减小癌肿体积的作用, miR-191参与了MAPK/ERK、TGF-β及MAPK/JNK 3条通路, SOX4、IL1A(interleukin-1a)、TMC7(transmembrane channel-like 7)这3个基因是miR-191的靶基因[21]. 另外, 在结肠癌、急性粒细胞白血病、恶性黑色素瘤、乳癌和卵巢癌等有miR-191高表达[3,22]. 所以, miR-191可能与恶性肿瘤的发生发展有着密切关系. 本研究通过qRT-PCR检测发现miR-191在胃癌组织中高表达, 与上述研究者的基因芯片结果一致, 提示miR-191可能参与胃癌的发病机制, 但尚需进一步的功能实验进行验证. 在多种肿瘤中, miR-191高表达与预后不佳有关[22]. 本实验显示miR-191表达与性别、年龄、病理组织分型、淋巴结转移和TNM分期无相关性, 这可能与样本量有关, 需扩大样本量进行验证, 也需对本组病人进行追踪随访以观察miR-191的表达与胃癌预后的关系.
预测miR-191的9个靶基因中的SOX4[21]和NDST1[23]为已证实的miR-191的靶基因. SOX4是与胚胎细胞发育、调节细胞分化增殖有关的转录因子, 其在多种肿瘤中高表达[21]. 胃癌细胞株MGC803中证实NDST1(N-deacetylase/N-sulfotransferase 1)是miR-191的靶基因, NDST1 抑制胃癌细胞增殖[23]. 其他已证实为miR-191的靶基因有肝细胞癌中的IL1A、TMC7[21]和TIMP3(tissue metallopeptidase inhibitor 3)[22], 小鼠红细胞发育成熟有关的Riock3和Mxil[24]. PLCD1是磷脂酶C家族中的一员, 参与激素分泌、神经递质传导、胞膜运输及细胞骨架调节等多种重要功能. PLCD1在胃癌中的表达降低并与其CpG岛的甲基化相关, 在胃癌发生中可能起着抑癌基因的作用[25]. 另外, PLCD1也被证明在乳腺癌和食管鳞状细胞癌中呈低表达并发挥抑癌的功能[26,27]. miR-191可能通过抑制这些基因的表达, 从而促使胃黏膜细胞的癌变. PLCD1可能是包括胃癌等多种癌症的抑癌基因, 并且为miR-191预测靶基因, 本实验选择PLCD1进行免疫组织化学实验, 初步分析其表达与miR-191的表达是否有相关性. 本实验结果显示, 相对于癌旁正常组织, PLCD1在胃癌中的表达降低, 而且miR-191与PLCD1在胃癌组织中表达水平呈显著负相关. 所以, 我们认为PLCD1可能是miR-191的靶基因, 但需要更进一步进行靶基因的鉴定.
生物信息学软件对miRNA靶基因的预测是功能研究的最初步骤, 由于miRNA与靶序列可以不完全配对结合, 一个miRNA可以有多个靶基因[28,29], 所以需要联合3-5个软件均预测到的基因作为靶基因从而降低假阳性率[30], 能够避免在功能实验中对靶基因选择的盲目性.
本实验应用了含有锁核苷酸(1ocked nucleic acid, LNA)技术的qRT-PCR试剂盒. miRNAs的以下特点将会给普通qRT-PCR研究带来困难: (1)成熟miRNAs序列短(20-22 nt); (2)哺乳动物中的miRNAs家族成员间往往只相差1个核苷酸; (3)引物结合目标序列除了在成熟miRNAs中也存在于pri-miRNA或pre-miRNA. 而LNA技术可以有效解决上述问题. LNA分子中部分核糖上的2'-O与4'-C连结在一起, 与靶核酸分子具有很强的杂交能力, 与DNA/RNA形成的双链的Tm值分别可升高1 ℃-8 ℃和2 ℃-10 ℃, 可以使整个qRT-PCR更具稳定性、特异性及敏感性[31].
总之, 本实验对miR-191在胃癌中表达情况的研究及其靶基因的预测为后续功能研究提供了重要依据.
在我国, 每年因胃癌死亡的患者占死于消化系肿瘤患者的首位, miRNAs与多种肿瘤相关, miR-191在多种肿瘤中呈差异性表达.
樊晓明, 教授, 主任医师, 复旦大学附属金山医院消化科
胃癌死亡率高, 早期诊断的手段少, 发现时一般已属中晚期, 寻找新的诊断标志物和治疗靶标是一个亟待解决的问题.
Ueda等应用基因芯片技术, 发现包括miR-191等22个miRNAs在胃癌中高表达, 13个miRNAs低表达; Hu等发现磷脂酶C-delta 1(PLCD1)在胃癌中呈低表达,与其启动子的CpG岛的甲基化有关,这表明PLCD1可能是胃癌抑癌基因.
运用qPCR技术验证前人用基因芯片技术发现miR-191在胃癌中高表达的结果, 并用生物信息学软件对其进行靶基因预测, 免疫组织化学检测靶基因的表达, 分析miR-191与靶基因的相关性.
本文检测了miR-191在胃癌组织中的表达及其与临床病理之间的关系, 并利用免疫组织化学半定量方法检测PLCD1, 间接验证了其可能是miR-191的靶基因. 该文对于进一步研究miR-191及其靶基因PLCD1在胃癌发生中的作用提供了初步依据.
编辑: 张姗姗 电编:鲁亚静
| 2. | Zhang B, Pan X, Cobb GP, Anderson TA. microRNAs as oncogenes and tumor suppressors. Dev Biol. 2007;302:1-12. [PubMed] [DOI] |
| 3. | Volinia S, Calin GA, Liu CG, Ambs S, Cimmino A, Petrocca F, Visone R, Iorio M, Roldo C, Ferracin M. A microRNA expression signature of human solid tumors defines cancer gene targets. Proc Natl Acad Sci U S A. 2006;103:2257-2261. [PubMed] [DOI] |
| 4. | Sakamoto H, Yoshimura K, Saeki N, Katai H, Shimoda T, Matsuno Y, Saito D, Sugimura H, Tanioka F, Kato S. Genetic variation in PSCA is associated with susceptibility to diffuse-type gastric cancer. Nat Genet. 2008;40:730-740. [PubMed] [DOI] |
| 5. | Rice TW, Blackstone EH, Rusch VW. 7th edition of the AJCC Cancer Staging Manual: esophagus and esophagogastric junction. Ann Surg Oncol. 2010;17:1721-1724. [PubMed] |
| 6. | Enright AJ, John B, Gaul U, Tuschl T, Sander C, Marks DS. MicroRNA targets in Drosophila. Genome Biol. 2003;5:R1. [PubMed] [DOI] |
| 7. | Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell. 2003;115:787-798. [PubMed] [DOI] |
| 8. | Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M. Combinatorial microRNA target predictions. Nat Genet. 2005;37:495-500. [PubMed] [DOI] |
| 10. | Taft RJ, Pang KC, Mercer TR, Dinger M, Mattick JS. Non-coding RNAs: regulators of disease. J Pathol. 2010;220:126-139. [PubMed] [DOI] |
| 11. | Hwang HW, Mendell JT. MicroRNAs in cell proliferation, cell death, and tumorigenesis. Br J Cancer. 2006;94:776-780. [PubMed] [DOI] |
| 12. | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-854. [PubMed] [DOI] |
| 13. | Sontheimer EJ, Carthew RW. Silence from within: endogenous siRNAs and miRNAs. Cell. 2005;122:9-12. [PubMed] [DOI] |
| 14. | Zamore PD, Haley B. Ribo-gnome: the big world of small RNAs. Science. 2005;309:1519-1524. [PubMed] [DOI] |
| 15. | Garzon R, Marcucci G, Croce CM. Targeting microRNAs in cancer: rationale, strategies and challenges. Nat Rev Drug Discov. 2010;9:775-778. [PubMed] [DOI] |
| 16. | Petrocca F, Visone R, Onelli MR, Shah MH, Nicoloso MS, de Martino I, Iliopoulos D, Pilozzi E, Liu CG, Negrini M. E2F1-regulated microRNAs impair TGFbeta-dependent cell-cycle arrest and apoptosis in gastric cancer. Cancer Cell. 2008;13:272-286. [PubMed] [DOI] |
| 17. | Kim YK, Yu J, Han TS, Park SY, Namkoong B, Kim DH, Hur K, Yoo MW, Lee HJ, Yang HK. Functional links between clustered microRNAs: suppression of cell-cycle inhibitors by microRNA clusters in gastric cancer. Nucleic Acids Res. 2009;37:1672-1681. [PubMed] [DOI] |
| 18. | Li X, Zhang Y, Zhang Y, Ding J, Wu K, Fan D. Survival prediction of gastric cancer by a seven-microRNA signature. Gut. 2010;59:579-585. [PubMed] [DOI] |
| 19. | Ueda T, Volinia S, Okumura H, Shimizu M, Taccioli C, Rossi S, Alder H, Liu CG, Oue N, Yasui W. Relation between microRNA expression and progression and prognosis of gastric cancer: a microRNA expression analysis. Lancet Oncol. 2010;11:136-146. [PubMed] [DOI] |
| 20. | Li X, Zhang Y, Zhang H, Liu X, Gong T, Li M, Sun L, Ji G, Shi Y, Han Z. miRNA-223 promotes gastric cancer invasion and metastasis by targeting tumor suppressor EPB41L3. Mol Cancer Res. 2011;9:824-833. [PubMed] [DOI] |
| 21. | Elyakim E, Sitbon E, Faerman A, Tabak S, Montia E, Belanis L, Dov A, Marcusson EG, Bennett CF, Chajut A. hsa-miR-191 is a candidate oncogene target for hepatocellular carcinoma therapy. Cancer Res. 2010;70:8077-8087. [PubMed] [DOI] |
| 22. | He Y, Cui Y, Wang W, Gu J, Guo S, Ma K, Luo X. Hypomethylation of the hsa-miR-191 locus causes high expression of hsa-mir-191 and promotes the epithelial-to-mesenchymal transition in hepatocellular carcinoma. Neoplasia. 2011;13:841-853. [PubMed] |
| 23. | Shi X, Su S, Long J, Mei B, Chen Y. MicroRNA-191 targets N-deacetylase/N-sulfotransferase 1 and promotes cell growth in human gastric carcinoma cell line MGC803. Acta Biochim Biophys Sin (Shanghai). 2011;43:849-856. [PubMed] [DOI] |
| 24. | Zhang L, Flygare J, Wong P, Lim B, Lodish HF. miR-191 regulates mouse erythroblast enucleation by down-regulating Riok3 and Mxi1. Genes Dev. 2011;25:119-124. [PubMed] [DOI] |
| 25. | Hu XT, Zhang FB, Fan YC, Shu XS, Wong AH, Zhou W, Shi QL, Tang HM, Fu L, Guan XY. Phospholipase C delta 1 is a novel 3p22.3 tumor suppressor involved in cytoskeleton organization, with its epigenetic silencing correlated with high-stage gastric cancer. Oncogene. 2009;28:2466-2475. [PubMed] [DOI] |
| 26. | Xiang T, Li L, Fan Y, Jiang Y, Ying Y, Putti TC, Tao Q, Ren G. PLCD1 is a functional tumor suppressor inducing G(2)/M arrest and frequently methylated in breast cancer. Cancer Biol Ther. 2010;10:520-527. [PubMed] [DOI] |
| 27. | Fu L, Qin YR, Xie D, Hu L, Kwong DL, Srivastava G, Tsao SW, Guan XY. Characterization of a novel tumor-suppressor gene PLC delta 1 at 3p22 in esophageal squamous cell carcinoma. Cancer Res. 2007;67:10720-10726. [PubMed] [DOI] |
| 28. | Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP. The impact of microRNAs on protein output. Nature. 2008;455:64-71. [PubMed] [DOI] |
| 29. | Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N. Widespread changes in protein synthesis induced by microRNAs. Nature. 2008;455:58-63. [PubMed] [DOI] |
| 30. | Min H, Yoon S. Got target? Computational methods for microRNA target prediction and their extension. Exp Mol Med. 2010;42:233-244. [PubMed] [DOI] |
| 31. | Kauppinen S, Vester B, Wengel J. Locked nucleic acid: high-affinity targeting of complementary RNA for RNomics. Handb Exp Pharmacol. 2006;405-422. [PubMed] |