修回日期: 2012-05-12
接受日期: 2012-06-01
在线出版日期: 2012-07-08
目的: 检测微小RNA-155(miR-155)在肝癌组织中的表达并分析其对肝癌细胞增殖和细胞凋亡的影响.
方法: 采用TagMan MGB探针法荧光定量PCR分析42例原发性肝癌及对应的癌旁组织miR-155的表达; 利用miR-155反义寡核苷酸(ASO-miR-155)降低肝癌细胞HepG2和SMMC7721中miR-155的表达; 利用MTT比色法检测肝癌细胞增殖的变化, 并通过流式细胞技术检测肝癌细胞早期凋亡情况.
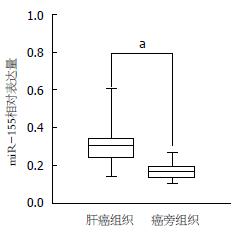
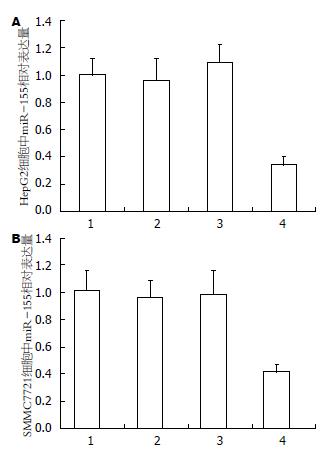
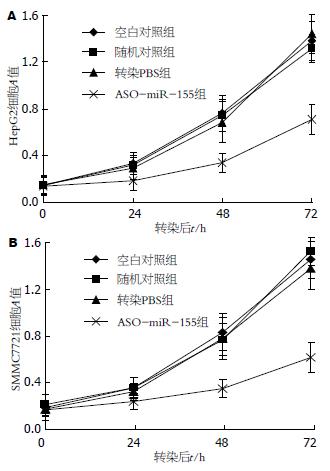
结果: 42例肝癌及癌旁组织标本中, miR-155在52%(22/42)肝癌组织中的表达明显高于癌旁组织(P<0.05); 利用脂质体将ASO-miR-155转染肝癌细胞HepG2和SMMC7721后, miR-155的表达明显降低, 肝癌细胞HepG2和SMMC7721生长受到明显抑制; 并且细胞的早期凋亡明显增加.
结论: miR-155在肝癌组织中过表达, 降低其表达能明显抑制肝癌细胞的生长并诱导细胞早期凋亡, miR-155有可能成为肝癌治疗的新靶点.
引文著录: 胡蓉环, 蔡婧, 兰琼玉. miR-155在肝细胞癌中的表达及对肝癌细胞增殖的影响. 世界华人消化杂志 2012; 20(19): 1737-1741
Revised: May 12, 2012
Accepted: June 1, 2012
Published online: July 8, 2012
AIM: To detect the expression of microRNA 155 (miR-155) in hepatocellular carcinoma and to analyze its influence on tumor cell proliferation and apoptosis.
METHODS: The expression of miR-155 in 42 cases of primary liver cancer and matched tumor-adjacent normal tissue was detected by quantitative PCR. The expression of miR-155 in hepatocellular carcinoma cell lines HepG2 and SMMC7721 was inhibited using a miR-155-specific antisense oligonucleotide (ASO-miR-155), and cell proliferation and early apoptosis were then determined by MTT assay and flow cytometry, respectively.
RESULTS: The positive rate of miR-155 expression was significantly higher in hepatocellular carcinoma (52%) than in tumor-adjacent tissue (P < 0.05). After ASO-miR-155 was transfected into HepG2 and SMMC7721 cells using Lipofectamine, the expression of miR-155 was significantly reduced. MiR155 knockdown significantly inhibited growth and promoted early apoptosis of HepG2 and SMMC7721 cells.
CONCLUSION: MiR-155 is overexpressed in hepatocellular carcinoma. Inhibition of miR-155 expression could inhibit growth and induce early apoptosis of hepatocellular carcinoma cells. MiR-155 may become a new target for the treatment of liver cancer.
- Citation: Hu RH, Cai J, Lan QY. Expression of miR-155 in primary hepatocellular carcinoma and its effect on tumor cell proliferation. Shijie Huaren Xiaohua Zazhi 2012; 20(19): 1737-1741
- URL: https://www.wjgnet.com/1009-3079/full/v20/i19/1737.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i19.1737
原发性肝细胞癌(hepatocellular carcinoma, HCC)是我国乃至世界上常见、且极具危害性的恶性肿瘤之一[1-4]. 尽管近年来对其诊断、治疗技术不断进步, 但肝癌的预后仍然较差, 对肝癌的早期诊断及早期治疗具有特别重要意义. MicroRNAs(miRNAs)是近年来发现的一类长度约为22个核苷酸的单链非编码小分子RNA, 通过与靶基因完全或不完全互补结合形式调控基因转录后的表达[5-7]. 在恶性肿瘤发生发展过程中, miRNAs经常扮演着"癌基因"和/或"抑癌基因"的重要角色[8-14]. 近年研究发现miR-155在乳腺癌、直肠癌、胰腺癌、肺癌等恶性肿瘤中表达上调, 并在其生物学行为方面起重要的调节作用[15-20]. 然而miR-155在肝癌中的表达及功能研究尚未见报道, 本研究拟检测miR-155在肝癌组织中的表达情况, 并通过反义寡核苷酸降低肝癌细胞miR-155的表达, 观察miR-155对肝癌细胞增殖和凋亡的影响, 以期为肝癌早期诊治提供新的理论基础.
收集2009-04/2011-04南昌大学第二附属医院肝胆外科手术切除的原发性肝癌组织标本42例, 每例均有对应的癌旁组织. 其中男25例, 女17例, 年龄35-72岁. 所有标本均经病理学检查确诊, 收集后立即放入液氮中保存. TaqMan miRNA分析试剂盒购自美国ABI公司; Annexin V-FITC凋亡检测试剂盒购自南京凯基公司; 脂质体LipofectamineTM 2000购自美国Invitrogen公司; 全硫代修饰的ASO-miR-155购自Gene Pharma公司; 人肝癌细胞株HepG2和SMMC7721由江西省分子医学重点实验室保存.
1.2.1 实时荧光定量PCR检测miR-155的表达: 采用TRIzol试剂提取组织总RNA, 紫外分光光度计测定浓度, -70 ℃保存备用, 用miR-155检测试剂盒检测miR-155的表达. 首先取2 μg总RNA作为模板与3 μL逆转录酶混合, 在20 μL反应体系中, 16 ℃ 30 min, 42 ℃ 30 min, 85 ℃ 5 min, 进行逆转录反应. 收集cDNA, 然后将cDNA 150倍稀释, 取1 μL稀释的cDNA与2 μL TaqMan引物混合, 20 μL反应体系: 95 ℃ 10 min; 95 ℃ 15 s, 60 ℃ 60 s, 40个循环. 相对miRNA表达水平用Ct值准确计算, U6 snRNA为内参.
1.2.2 细胞株的培养及转染: HepG2和SMMC7721细胞株接种于含100 mL/L胎牛血清的DMEM培养基, 37 ℃、50 mL/L CO2条件下培养. 收集细胞, 以1×105个细胞/孔铺6孔板, 细胞生长至90%融合率时严格按照脂质体LipofectamineTM 2000转染试剂说明书进行转染.
1.2.3 ASO-miR-155转染后对miR-155表达的影响: 转染ASO-miR-155肝癌细胞48 h后, 抽提总RNA经纯度分析, 逆转录为cDNA, 测定cDNA浓度, 同时设立空白对照组、随机对照组及转染PBS组, 检测miR-155的表达. 实验重复3次.
1.2.4 MTT检测各组细胞的增殖情况: 转染ASO-miR-155后48 h收集细胞, 在离心管内将各组细胞悬液充分打匀, 按8×103/孔接种于96孔培养板, 每孔加液量200 μL, 24 h后换液. 实验分组同1.2.3, 每组设6个复孔. 继续将培养板移入培养箱中培养24、48、72 h后分别每孔加入5 g/L试剂MTT 20 μL, 培养4 h后, 吸去上清液, 加入150 μL的DMSO液, 振荡培养板10 min, 用酶标仪检测波长为490 nm的吸光度(A)值. 每组重复3次. 按下列公式计算细胞生长抑制率(%):
Math 1
1.2.5 流式细胞术分析细胞早期凋亡: 试验分组同1.2.3. 细胞接种6孔板, 转染ASO-miR-155后48 h收集细胞, 250 μL结合缓冲液重悬细胞, 取100 μL细胞悬液于5 mL流式管中, 加入5 μL Annexin V/PI和10 mL 20 mg/L的碘化丙啶(PI终浓度1 mg/L), 室温避光孵育15 min, 加入400 μL PBS, 流式细胞仪检测, 实验重复3次.
统计学处理 采用SPSS17.0统计软件, 计量资料以mean±SD表示, 两组比较采用独立样本t检验, 多组间比较采用单因素方差分析, P<0.05为差异有统计学意义.
实时荧光定量PCR检测发现42对原发性肝癌及癌旁组织中, 52%(22/42)的肝癌组织中miR-155的表达明显高于对应的癌旁组织, 结果有统计学意义(P<0.05, 图1).
肝癌细胞HepG2和SMMC7721中转染ASO-miR-155后48 h, 实时荧光定量PCR检测发现, miR-155的表达较对照组降低(P<0.05), 而空白对照组、随机对照组及PBS组间无明显差异(P>0.05, 图2).
转染ASO-miR-155后肝癌细胞生长明显慢于对照组(P<0.05), 而空白对照组、随机对照组及转染PBS组间肝癌细胞的生长无明显差异(P>0.05, 图3).
肝癌细胞HepG2和SMMC7721转染ASO-miR-155后48 h, 结果发现转染ASO-miR-155组较空白对照组、随机对照组、转染PBS组早期凋亡明显增加(P<0.05), 而晚期凋亡无明显改变; 空白对照组、随机对照组、转染PBS组之间各组细胞未见明显变化(P>0.05, 表1, 2).
原发性肝癌的发生发展是癌基因激活和抑癌基因失活共同作用的结果, 其确切的发病机制尚未完全清楚. 研究表明, 作为多步骤、多途径肿瘤发生的典型, 发现肝癌的早期诊断标记物和治疗靶点具有重要意义[21,22]. 近年发现, miRNAs与恶性肿瘤密切相关, 主要通过与目标mRNA分子的3'端非编码区域特异结合来抑制转录后的翻译. 约52%调控miRNAs的编码基因位于肿瘤相关基因位点和染色体区域[23,24]. 因此miRNAs可能成为诊断肿瘤的新的分子标志和判断肿瘤治疗及预后的分子靶点[25,26]. miR-155定位于人染色体21q21, 由B细胞整合簇(B-cell integration cluster, BIC)基因第3个外显子高度保守的区域编码[27]. 最近研究发现, miR-155在多种人体恶性肿瘤中异常表达, 与肿瘤的发生、发展及预后密切相关, 被认为是癌性微小RNA[15]. 另外, Kong等[28]研究证实, 乳腺癌中过表达的miR-155通过抑制人叉头框蛋白03(forkhead box protein 03, FOX03)表达, 促进乳腺癌的存活及增殖, 降低乳腺癌对化疗药物的敏感性. Greither等[29]研究显示, 胰腺癌组织中miR-155的表达异常增高, 预示患者预后不良. Habbe等[30]研究显示, 当胰腺导管上皮发生上皮内瘤变时, 其细胞及胰液内miR-155水平即有所增高, 且在最容易癌变的肠型上皮内瘤变中miR-155升高最明显. 提示miR-155可作为早期筛查胰腺癌的重要指标. 但miR-155在肝癌中的表达及其作用目前还不清楚.
我们首先通过荧光定量PCR TagMan MGB探针法分析了42例肝癌患者手术标本癌组织及对应癌旁组织中miR-155的表达, 发现52%(22/42)的肝癌患者癌组织中miR-155表达显著上调(P<0.05), 表明miR-155很可能成为一个新的肝癌的癌前标记物. 李海东等[15]针对miR-122及miR-192的反义寡核苷酸能够明显抑制小鼠不同组织中对应miRNAs的表达, 表明针对miRNAs的反义寡核苷酸可以有效抑制miRNAs的表达. 随后本研究同样利用反义寡核苷酸技术成功降低肝癌细胞HepG2和SMMC7721中miR-155表达, 发现其表达明显下降. 同时我们通过MTT法检测发现HepG2和SMMC7721肝癌细胞的生长受到抑制. 最后我们应用流式细胞术对转染ASO-miR-155前后细胞凋亡情况进行分析, 结果发现降低miR-155表达后, 细胞的早期凋亡也明显增加.
总之, miR-155在肝细胞癌中过表达, 参与促进肝癌细胞的增殖并抑制凋亡, miR-155很可能成为一个新的肝癌癌前标记物, 为肝癌基因治疗提供新的靶点. 但miR-155通过哪些靶基因促进肝癌细胞的增殖及具体调控机制仍有待进一步研究.
原发性肝癌恶性程度高, 治疗效果差, miRNAs在肝癌等恶性肿瘤发生发展过程中起了癌基因或抑癌基因的作用.
王阁, 教授, 中国人民解放军第三军医大学第三附属医院
近年研究发现miR-155在乳腺癌、直肠癌、胰腺癌、肺癌等恶性肿瘤中表达上调, 并在其生物学行为方面起重要的调节作用, 但在肝癌中表达的功能尚不完全清楚.
Kong等研究证实, 乳腺癌中过表达的miR-155通过抑制人叉头框蛋白03表达, 促进乳腺癌的存活及增殖, 降低乳腺癌对化疗药物的敏感性. Greither等研究显示, 胰腺癌组织中miR-155的表达异常增高, 预示患者预后不良.
本研究分析了miR-155在肝癌及癌旁组织中的表达, 并探讨了降低miR-155表达后肝癌细胞的增殖及凋亡情况.
本文提示miR-155在肝细胞癌中过表达, 参与促进肝癌细胞的增殖并抑制凋亡. 选题起点高, 技术方法先进, 结果可靠, 结论客观, 图表清晰.
编辑: 张姗姗 电编:鲁亚静
| 1. | Jemal A, Siegel R, Ward E, Hao Y, Xu J, Thun MJ. Cancer statistics, 2009. CA Cancer J Clin. 2009;59:225-249. [PubMed] [DOI] |
| 2. | Llovet JM, Bruix J. Novel advancements in the management of hepatocellular carcinoma in 2008. J Hepatol. 2008;48 Suppl 1:S20-S37. [PubMed] [DOI] |
| 3. | Jemal A, Bray F, Center MM, Ferlay J, Ward E, Forman D. Global cancer statistics. CA Cancer J Clin. 2011;61:69-90. [PubMed] [DOI] |
| 5. | Zeng Y. Principles of micro-RNA production and maturation. Oncogene. 2006;25:6156-6162. [PubMed] [DOI] |
| 6. | Kloosterman WP, Plasterk RH. The diverse functions of microRNAs in animal development and disease. Dev Cell. 2006;11:441-450. [PubMed] [DOI] |
| 7. | Sayed D, Abdellatif M. MicroRNAs in development and disease. Physiol Rev. 2011;91:827-887. [PubMed] [DOI] |
| 8. | Croce CM. Causes and consequences of microRNA dysregulation in cancer. Nat Rev Genet. 2009;10:704-714. [PubMed] [DOI] |
| 9. | Munker R, Calin GA. MicroRNA profiling in cancer. Clin Sci (Lond). 2011;121:141-158. [PubMed] [DOI] |
| 10. | Szafranska AE, Davison TS, John J, Cannon T, Sipos B, Maghnouj A, Labourier E, Hahn SA. MicroRNA expression alterations are linked to tumorigenesis and non-neoplastic processes in pancreatic ductal adenocarcinoma. Oncogene. 2007;26:4442-4452. [PubMed] [DOI] |
| 11. | Yu DC, Li QG, Ding XW, Ding YT. Circulating MicroRNAs: Potential Biomarkers for Cancer. Int J Mol Sci. 2011;12:2055-2063. [PubMed] [DOI] |
| 12. | Tricoli JV, Jacobson JW. MicroRNA: Potential for Cancer Detection, Diagnosis, and Prognosis. Cancer Res. 2007;67:4553-4555. [PubMed] [DOI] |
| 13. | Zhang W, Dahlberg JE, Tam W. MicroRNAs in tumorigenesis: a primer. Am J Pathol. 2007;171:728-738. [PubMed] [DOI] |
| 14. | Cho WC. OncomiRs: the discovery and progress of microRNAs in cancers. Mol Cancer. 2007;6:60. [PubMed] [DOI] |
| 16. | Jiang S, Zhang HW, Lu MH, He XH, Li Y, Gu H, Liu MF, Wang ED. MicroRNA-155 functions as an OncomiR in breast cancer by targeting the suppressor of cytokine signaling 1 gene. Cancer Res. 2010;70:3119-3127. [PubMed] [DOI] |
| 17. | Nikiforova MN, Tseng GC, Steward D, Diorio D, Nikiforov YE. MicroRNA expression profiling of thyroid tumors: biological significance and diagnostic utility. J Clin Endocrinol Metab. 2008;93:1600-1608. [PubMed] [DOI] |
| 18. | Yanaihara N, Caplen N, Bowman E, Seike M, Kumamoto K, Yi M, Stephens RM, Okamoto A, Yokota J, Tanaka T. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell. 2006;9:189-198. [PubMed] [DOI] |
| 19. | Wang X, Tang S, Le SY, Lu R, Rader JS, Meyers C, Zheng ZM. Aberrant expression of oncogenic and tumor-suppressive microRNAs in cervical cancer is required for cancer cell growth. PLoS One. 2008;3:e2557. [PubMed] [DOI] |
| 20. | Bloomston M, Frankel WL, Petrocca F, Volinia S, Alder H, Hagan JP, Liu CG, Bhatt D, Taccioli C, Croce CM. MicroRNA expression patterns to differentiate pancreatic adenocarcinoma from normal pancreas and chronic pancreatitis. JAMA. 2007;297:1901-1908. [PubMed] [DOI] |
| 23. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 24. | Zhong X, Coukos G, Zhang L. miRNAs in human cancer. Methods Mol Biol. 2012;822:295-306. [PubMed] [DOI] |
| 25. | Garzon R, Fabbri M, Cimmino A, Calin GA, Croce CM. MicroRNA expression and function in cancer. Trends Mol Med. 2006;12:580-587. [PubMed] [DOI] |
| 26. | Jemal A, Siegel R, Ward E, Hao Y, Xu J, Murray T, Thun MJ. Cancer statistics, 2008. CA Cancer J Clin. 2008;58:71-96. [PubMed] [DOI] |
| 27. | Xiang X, Zhuang X, Ju S, Zhang S, Jiang H, Mu J, Zhang L, Miller D, Grizzle W, Zhang HG. miR-155 promotes macroscopic tumor formation yet inhibits tumor dissemination from mammary fat pads to the lung by preventing EMT. Oncogene. 2011;30:3440-3453. [PubMed] [DOI] |
| 28. | Kong W, He L, Coppola M, Guo J, Esposito NN, Coppola D, Cheng JQ. MicroRNA-155 regulates cell survival, growth, and chemosensitivity by targeting FOXO3a in breast cancer. J Biol Chem. 2010;285:17869-17879. [PubMed] [DOI] |
| 29. | Greither T, Grochola LF, Udelnow A, Lautenschläger C, Würl P, Taubert H. Elevated expression of microRNAs 155, 203, 210 and 222 in pancreatic tumors is associated with poorer survival. Int J Cancer. 2010;126:73-80. [PubMed] [DOI] |
| 30. | Habbe N, Koorstra JB, Mendell JT, Offerhaus GJ, Ryu JK, Feldmann G, Mullendore ME, Goggins MG, Hong SM, Maitra A. MicroRNA miR-155 is a biomarker of early pancreatic neoplasia. Cancer Biol Ther. 2009;8:340-346. [PubMed] [DOI] |