修回日期: 2012-03-13
接受日期: 2012-03-25
在线出版日期: 2012-05-08
目的: 建立肝癌血清学诊断模型, 探讨评估SELDI-TOF-MS技术在肝癌诊断和介入治疗评价中的价值.
方法: 用弱阳离子交换芯片(CM10芯片)和表面增强激光解吸电离飞行时间质谱仪(surface-enhanced laser desorption ionization time-of-flight mass spectrometry, SELDI-TOF-MS)技术, 测定60例肝癌患者和60例正常对照者的血清蛋白质指纹图谱, 应用Biomarker Wizard统计软件比较肝癌组和正常对照组血清蛋白质表达的差异性, 采用Biomarker Pattern软件分析数据建立肝癌诊断模型, 比较介入治疗前后血清蛋白质指纹图谱的差异性.
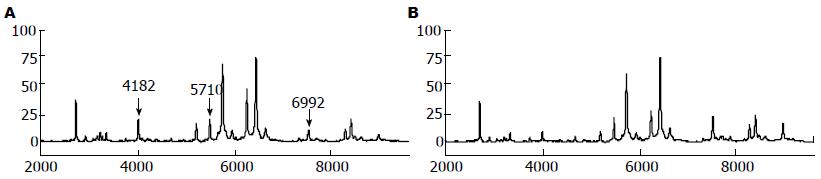
结果: 在质荷比(M/Z)为2 000-10 000范围内, 和正常血清比较, 肝癌的差异峰有3个(M/Z为4 182 Da、5 710 Da、6 992 Da; P<0.01), 4 182 Da和5 710 Da下调, 6 992 Da上调. 用这3个差异蛋白峰建立肝癌诊断模型, 诊断肝癌的灵敏度为93.3%(28/30), 特异度为90.0%(27/30), 正确率为91.7%(55/60), 约登指数为0.833. 差异蛋白峰(M/Z 4 182 Da)在介入术后1 mo明显上调(P<0.05).
结论: 应用SELDI-TOF-MS技术进行肝癌血清蛋白质指纹图谱分析, 建立肝癌诊断树模型, 对肝癌的诊断有一定的价值; 筛选出的差异蛋白峰对肝癌的介入治疗评估有一定的应用价值.
引文著录: 胡育斌, 林海澜, 郝明志, 叶韵斌, 陈慧菁, 陈起忠, 陈强. SELDI-TOF-MS技术在肝癌诊断和介入治疗评价中的价值. 世界华人消化杂志 2012; 20(13): 1107-1111
Revised: March 13, 2012
Accepted: March 25, 2012
Published online: May 8, 2012
AIM: To establish a serum protein pattern model for diagnosing hepatocellular carcinoma (HCC) by using the surface-enhanced laser desorption ionization time-of-fligh mass spectrometry (SELDI-TOF-MS), and to evaluate the value of this model in predicting the effect of interventional treatment for HCC.
METHODS: Serum samples collected from patients with HCC (n = 60) and healthy people (n = 60) were used for SELDI-TOF-MS on CM10 chips. The proteomic spectra were analyzed by using the Biomarker Wizard software. The diagnosis model was established by using the Biomarker Pattern software.
RESULTS: Three significantly different protein peaks were found in serum samples between HCC patients and healthy controls. A protein peak at 6 992 Da showed higher expression and the other two protein peaks (4 182 Da, 5 710 Da) showed lower expression in HCC patients than in healthy people. The diagnostic model containing these three candidate biomarkers could distinguish patients with HCC from healthy controls with a sensitivity of 93.3% (28/30), a specificity of 90.0% (27/30), an accuracy of 91.7% (55/60), and a Youden index value of 0.833. The protein peak at 4 182 Da was significantly decreased one month after interventional treatment in HCC patients (P < 0.05).
CONCLUSION: The diagnostic model developed by using SELDI-TOF-MS allows efficiently identifying patients with HCC and may play a valuable role in the diagnosis of HCC. The protein peak at 4 182 Da is helpful for the evaluation of interventional curative effect in HCC patients.
- Citation: Hu YB, Lin HL, Hao MZ, Ye YB, Chen HJ, Chen QZ, Chen Q. Application of surface-enhanced laser desorption ionization time-of-flight mass spectrometry to the diagnosis and evaluation of interventional effect in patients with hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(13): 1107-1111
- URL: https://www.wjgnet.com/1009-3079/full/v20/i13/1107.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i13.1107
原发性肝癌发病率和病死率高, 在我国年发病数约30.6万人, 死亡率居我国肿瘤死因的第2位[1,2]. 早期诊断和治疗是提高原发性肝癌疗效的关键. 目前表面增强激光解吸电离飞行时间质谱仪技术(surface-enhanced laser desorption ionization time-of-flight mass spectrometry, SELDI-TOF-MS)在肝癌诊断的价值已得到肯定[3-6], 而在介入治疗评价中的应用却鲜有报道. 本课题应用SELDI-TOF-MS技术建立肝癌诊断模型, 探讨其在原发性肝癌诊断和介入治疗评价中的价值.
收集2009-01/12初次在我科治疗的原发性肝癌患者60例, 其中男48例, 女12例, 年龄19-81岁, 中位年龄53岁, 诊断符合2001-09第8届全国肝癌学术会议修订的诊断标准[7], 其中Ⅰb期10例, Ⅱa期14例, Ⅱb期19例, Ⅲa期9例, Ⅲb期8例. 对照样品来自2009-01/12在我院体检的健康志愿者60例, 其中男45例, 女15例, 年龄15-78岁, 中位年龄49岁. 乙酸钠、乙腈、三氟乙酸、尿素、CHAPS缓冲液购自Sigma公司, 芥子酸、弱阳离子交换芯片(CM10)、生物处理器、PBSⅡ质谱仪均购自美国Ciphergen公司.
采集患者(治疗前)及健康对照者清晨空腹静脉血5 mL, 4 ℃静置1 h后分离血清, -80 ℃保存. 取10 μL与20 μL U9缓冲液EP管中充分混匀, 冰浴振荡30 min, 加入360 μL CM10-buffer混匀. 用CM10-buffer预处理的芯片中加入100 μL处理好的样品, 室温下震荡1 h. 洗涤芯片, 分2次加上SPA 2 μL, 干燥即可上机检测. 采用PBSⅡ-C型蛋白质芯片阅读仪读取芯片, 利用Ciphergen ProteinChip 3.1.1软件收集数据. 利用Biomarker Wizard 3.1软件筛选表达有差异的蛋白质峰.
统计学处理 用BioMarker Wizard软件对血清蛋白质谱数据进行统计学分析, 分析蛋白质峰强度与数量的差异性, 筛选出的蛋白质峰在10%以上的不同样本中同时存在, 且同一蛋白质峰在不同样本中的偏差<0.3%. 对不同分组中相同质荷比的蛋白质含量用Biomarker Wizard软件进行分析, P<0.05具有统计学意义. 用Biomarker Pattern软件用于分析数据建立分类树诊断模型.
肝癌患者与健康对照血清图谱相比较, 筛选出3个差异蛋白质峰其质荷比(M/Z)分别为4 182 Da、5 710 Da、6 992 Da(P<0.01, 表1, 图1). 其中M/Z为4 182 Da、5 710 Da的差异蛋白质峰表达下调, M/Z为6 992 Da的差异蛋白质峰表达上调.
| M/Z(Da) | 肝癌组平均值 | 肝癌组标准差 | 健康对照组平均值 | 健康对照组标准差 | P值 |
| 4182 | 3.469 | 3.591 | 10.933 | 5.560 | 3.1×10-5 |
| 5710 | 1.926 | 1.271 | 3.912 | 1.310 | 2.6×10-6 |
| 6992 | 2.360 | 1.107 | 1.730 | 0.933 | 8.0×10-4 |
采用BioMarker Patterns System软件以M/Z为4 182 Da、5 710 Da和6 992 Da的3个差异蛋白质峰建立肝癌诊断模型, 其灵敏度为95.0%, 特异度为91.7%(表2).
| 测试结果 | 正确划分(n) | 错误划分(n) | 正确率(%) |
| 肝癌组 | 57 | 3 | 95.0 |
| 健康对照组 | 55 | 5 | 91.7 |
| 合计 | 112 | 8 | 93.3 |
| 盲筛验证结果 | |||
| 肝癌组 | 28 | 2 | 93.3 |
| 健康对照组 | 27 | 3 | 90.0 |
| 合计 | 55 | 5 | 91.7 |
根据建立的肝癌诊断模型对随机抽选的30例肝癌患者和30例正常对照进行盲筛, 结果灵敏度为93.3%, 特异性为90.0%, 符合率为91.7%, 约登指数为0.833(表2).
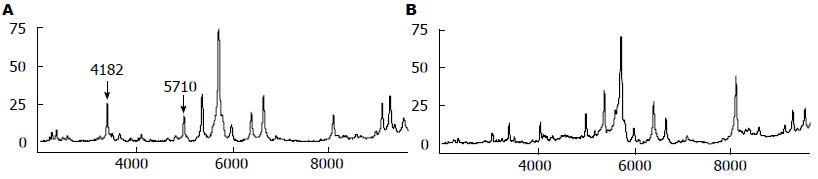
采用Biomarker Wizard软件对60例原发性肝癌患者介入治疗前与介入治疗后1 wk的血清蛋白质谱图进行比较和统计学分析(表3, 图2), 筛选有统计学意义的血清蛋白质差异峰. 结果显示: M/Z为4 182 Da、5 710 Da的2个差异蛋白质峰在介入治疗后1 wk表达均显著下调(P<0.05).
| M/Z(Da) | 介入前平均值 | 介入前标准差 | 介入后平均值 | 介入后标准差 | P值 |
| 1 wk | |||||
| 4 182 | 2.808 | 1.9013 | 1.908 | 1.0376 | 0.043 |
| 5 710 | 2.064 | 1.3469 | 1.328 | 0.6567 | 0.018 |
| 1 mo | |||||
| 4 182 | 1.908 | 1.0376 | 3.220 | 2.3427 | 0.014 |
| 5 710 | 2.408 | 1.9287 | 1.968 | 0.8683 | 0.303 |
临床上约有2/3的原发性肝癌患者初诊时已属中晚期, 失去了根治性治疗的机会, 一般生存期不到半年, 即使经过积极有效地治疗, 其5年生存率也不足10%. 而早期发现的亚临床期小肝癌经过手术等综合治疗后, 5年生存率可提高为60%-80%. 因此肝癌早期诊断至关重要. 肝癌的形成是一个多步骤、多因素、多基因参与的过程, 包括一系列基因和蛋白质表达的变化, 而有些基因或蛋白可用作肝癌预后的标记[8]. 目前肝癌的生物标记物以甲胎蛋白(AFP)最为理想[9]. 但70%的肝癌患者血清AFP浓度低于诊断肝癌的临界值(500 μg/L)[10], 约30%的肝癌患者AFP阴性, 且易受肝炎、生殖源性肿瘤等影响出现假阳性[11]. AFP的敏感性和特异性尚不理想, 多中心三期临床试验认为其灵敏度为43%, 特异度为46%[12]. 故寻找新的肝癌标志物以提高肝癌诊断水平尤为重要.
蛋白质作为功能基因的直接体现者, 是生命活动和生物结构最主要的承担者[13]. 尽管所有的细胞都拥有同样的基因组, 但在不同的细胞内会有不同的基因处在活跃状态, 从而合成不同的蛋白质; 即使同一细胞, 当处于不同机体功能状态时合成和分泌的蛋白质也会发生改变[14]. 蛋白质通常代表疾病的分子诊断的终末点[15], 人血清是一个蛋白质相当丰富并不断变化的系统, 可能含有大量未发现的可用作疾病标志物的多肽[16], 单一的血清肿瘤标记物难以真正反溯到相关的细胞组织, 而发现一组与特定组织癌变相关的血清蛋白质(多参数标记物)则是更有效的途径, 这也就是所谓的"癌症指纹"[17].
由于SELDI-TOF-MS技术具有高分辨率、高流通量的特点, 其灵敏度可达10-15 mol, 是目前最有希望的肿瘤早期检测方法[18], 他可在疾病发生的极早期检测到某些蛋白质的变化, 由此从理论上讲, 该技术亦可用于肿瘤的早期预警.
本研究结果发现, M/Z为4 182 Da、5 710 Da处的蛋白质含量在正常人组明显高于肝癌组, 推测这两处蛋白质可能含有在肝细胞癌的发生、发展时被抑制的蛋白, 他们可能在维持细胞正常功能, 促进凋亡和抑制增殖等方面发挥作用. 本研究建立的肝癌诊断模型诊断肝癌的灵敏度达93.3%, 特异度达90.0%, 均明显优于文献报道的AFP诊断肝癌的价值, 在肝癌诊断中显示出了很好的应用价值.
肝癌组M/Z为4 182 Da处蛋白质峰值在介入治疗前明显低于正常对照组, 介入治疗后1 wk明显下调, 而介入术后1 mo 4 182 Da峰值显著上调, 有向正常对照峰值变化的趋势, 提示4 182 Da表达水平可能与肿瘤负荷的大小有关. 4 182 Da在介入治疗后1 wk表达下降, 可能与介入治疗后肝功能损伤有关, 而介入治疗后1 mo其表达明显增加, 考虑为介入治疗后肿瘤细胞大量坏死致使肿瘤负荷明显减小. 此动态变化规律, 提示M/Z为4 182 Da处的蛋白质很可能含有对肝癌介入治疗极为敏感的靶分子. 随着在今后的研究中样本量的扩大, 随访时间的延长, 4 182 Da可能成为评价肝癌介入治疗的参考指标.
但由于SELDI-TOF-MS技术中每一个波峰的质荷比对应的可能是很多个相对分子质量相近的多肽, 并不能直接对复杂体液中的具体蛋白质或多肽进行鉴定[19]. 同时由于蛋白质指纹图谱的重复性较差, 不同实验室对肝癌患者进行血清蛋白分析的结果不尽相同, 目前很难得到一个统一的"金标准". 把血清蛋白质指纹图谱与AFP联合应用于肝癌的早期诊断, 可能是以后的发展方向.
目前甲胎蛋白(AFP)诊断肝癌的敏感性和特异性尚不理想, 寻找新的肝癌标志物以提高肝癌诊断水平尤为重要.
唐南洪, 教授, 福建医科大学附属协和医院省肝胆外科研究所; 张宗明, 教授, 清华大学第一附属医院消化医学中心
SELDI-TOF-MS在肝癌诊断的价值已得到肯定, 而在介入治疗评价中的应用却鲜有报道.
由于蛋白质指纹图谱的重复性较差, 把血清蛋白质指纹图谱与AFP联合应用于肝癌的早期诊断, 可能是以后的发展方向.
SELDI-TOF-MS技术用于评价肝癌介入治疗还较少见, 本文创新性强, 有一定的学术价值.
编辑: 张珊珊 电编:鲁亚静
| 1. | Parkin DM. Global cancer statistics in the year 2000. Lancet Oncol. 2001;2:533-543. [PubMed] [DOI] |
| 2. | Wang HT, Wang J, Ou QJ, Liu XP, Chen S. [Expression of growth hormone receptor mRNA in hepatocellular carcinoma and matched para-cancer cirrhotic liver tissue]. Ai Zheng. 2002;21:146-148. [PubMed] |
| 3. | Ward DG, Cheng Y, N'Kontchou G, Thar TT, Barget N, Wei W, Billingham LJ, Martin A, Beaugrand M, Johnson PJ. Changes in the serum proteome associated with the development of hepatocellular carcinoma in hepatitis C-related cirrhosis. Br J Cancer. 2006;94:287-292. [PubMed] [DOI] |
| 4. | Lee IN, Chen CH, Sheu JC, Lee HS, Huang GT, Chen DS, Yu CY, Wen CL, Lu FJ, Chow LP. Identification of complement C3a as a candidate biomarker in human chronic hepatitis C and HCV-related hepatocellular carcinoma using a proteomics approach. Proteomics. 2006;6:2865-2873. [PubMed] [DOI] |
| 8. | Zeindl-Eberhart E, Haraida S, Liebmann S, Jungblut PR, Lamer S, Mayer D, Jäger G, Chung S, Rabes HM. Detection and identification of tumor-associated protein variants in human hepatocellular carcinomas. Hepatology. 2004;39:540-549. [PubMed] [DOI] |
| 9. | Feitelson MA, Sun B, Satiroglu Tufan NL, Liu J, Pan J, Lian Z. Genetic mechanisms of hepatocarcinogenesis. Oncogene. 2002;21:2593-2604. [PubMed] [DOI] |
| 10. | Seow TK, Liang RC, Leow CK, Chung MC. Hepatocellular carcinoma: from bedside to proteomics. Proteomics. 2001;1:1249-1263. [PubMed] [DOI] |
| 11. | Yoshida S, Kurokohchi K, Arima K, Masaki T, Hosomi N, Funaki T, Murota M, Kita Y, Watanabe S, Kuriyama S. Clinical significance of lens culinaris agglutinin-reactive fraction of serum alpha-fetoprotein in patients with hepatocellular carcinoma. Int J Oncol. 2002;20:305-309. [PubMed] |
| 12. | Hayman MW, Przyborski SA. Proteomic identification of biomarkers expressed by human pluripotent stem cells. Biochem Biophys Res Commun. 2004;316:918-923. [PubMed] [DOI] |
| 14. | Wilkins MR, Pasquali C, Appel RD, Ou K, Golaz O, Sanchez JC, Yan JX, Gooley AA, Hughes G, Humphery-Smith I. From proteins to proteomes: large scale protein identification by two-dimensional electrophoresis and amino acid analysis. Biotechnology (N Y). 1996;14:61-65. [PubMed] [DOI] |
| 15. | Kumar S, Mohan A, Guleria R. Biomarkers in cancer screening, research and detection: present and future: a review. Biomarkers. 2006;11:385-405. [PubMed] [DOI] |
| 16. | Liotta LA, Ferrari M, Petricoin E. Clinical proteomics: written in blood. Nature. 2003;425:905. [PubMed] [DOI] |
| 17. | Powell K. Proteomics delivers on promise of cancer biomarkers. Nat Med. 2003;9:980. [PubMed] [DOI] |
| 18. | Paradis V, Degos F, Dargère D, Pham N, Belghiti J, Degott C, Janeau JL, Bezeaud A, Delforge D, Cubizolles M. Identification of a new marker of hepatocellular carcinoma by serum protein profiling of patients with chronic liver diseases. Hepatology. 2005;41:40-47. [PubMed] [DOI] |
| 19. | Issaq HJ, Veenstra TD, Conrads TP, Felschow D. The SELDI-TOF MS approach to proteomics: protein profiling and biomarker identification. Biochem Biophys Res Commun. 2002;292:587-592. [PubMed] [DOI] |