修回日期: 2010-03-29
接受日期: 2010-04-07
在线出版日期: 2011-02-08
目的: 构建类泛素FAT10高表达肝癌细胞株Hep3B的酵母双杂交用cDNA文库.
方法: 从肝癌细胞Hep3B中提取总RNA, 分离mRNA. 利用反转录酶M-MLV与Oligo(dT) Anchor Primer合成1st Strand cDNA, 用E.coli DNA Polymerase与E.coli DNA Ligase将RNA链置换成DNA链, 合成2nd Strand cDNA. 将双链cDNA与EcoRⅠ Adaptor连接, 然后用EcoRⅠ/XhoⅠ进行酶切. 使用Spin Column除去短链cDNA与pGADT7载体连接, 转化入E.coli DH10B, 建成原始文库. 然后对其进行扩增并随机挑取单菌落, 酶切鉴定重组子插入片段大小.
结果: 提取的总RNA降解少且分子完整; RNA纯度高, 相对分子质量为400-5000 bp; 成功合成双链cDNA, 均符合建库要求; 库容量达到1.03×106克隆, 原始文库滴度为2.50×109 cfu/L, 扩增后的文库滴度为3.60×1012 cfu/L. 插入片段大小分布为0.5-3.5 kb, 平均长度约为2.0 kb.
结论: 所构建文库的各项指标均达到要求, 为筛选FAT10作用蛋白奠定了重要基础.
引文著录: 余新, 刘天, 德洪波, 李国惠, 邵江华. 类泛素FAT10高表达肝癌细胞株Hep3B酵母双杂交cDNA文库的构建. 世界华人消化杂志 2011; 19(4): 400-403
Revised: March 29, 2010
Accepted: April 7, 2010
Published online: February 8, 2011
AIM: To construct a yeast two-hybrid cDNA library from a FAT10-overexpressing human hepatic carcinoma cell line Hep3B.
METHODS: Total RNA was prepared from Hep3B cells and used to purify poly (A) mRNA. Double-stranded cDNA was synthesized from the purified mRNA, ligated to EcoR I adaptor, digested with EcoR I/Xho I enzymes, and then cloned into the pGADT7 vector. The recombinant vector was transformed into E. coli DH10B to obtain a primary cDNA library. The primary library was amplified and used to determine the size of cDNA inserts through enzyme digestion.
RESULTS: The primary cDNA library contained 1.03 × 106 independent clones. The titer of the cDNA library was estimated to be 2.50 × 106 cfu/mL, and that of the amplified library was 3.60 × 109 cfu/mL. The size of the inserts varied from 0. 5 to 3.5 kb, with an average value of about 2.0 kb.
CONCLUSION: A yeast two-hybrid cDNA library has been successfully generated from FAT10-overexpressing Hep3B cells and can be used for future screening of proteins interacting with FAT10.
- Citation: Yu X, Liu T, De HB, Li GH, Shao JH. Construction and characterization of a yeast two-hybrid cDNA library from a FAT10-overexpressing human hepatic carcinoma cell line Hep3B. Shijie Huaren Xiaohua Zazhi 2011; 19(4): 400-403
- URL: https://www.wjgnet.com/1009-3079/full/v19/i4/400.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i4.400
原发性肝细胞癌(hepatocellular carcinoma, HCC)是严重危害人类健康的恶性肿瘤, 其发生与发展是一个多步骤、多基因的参与过程. 泛素/类泛素系统为体内细胞蛋白降解的一个重要途径, 大量的研究表明泛素/类泛素系统功能失调将导致癌基因与抑癌基因的表达异常, 其在肿瘤的发生、发展过程中起着重要作用. 类泛素基因有着同泛素类似的结构域, 作为信号使者控制着多种细胞功能, 如细胞增殖、凋亡、细胞周期及DNA修复. 其功能的异常可导致许多疾病的产生, 包括肝癌. FAT10作为类泛素家族成员, 被发现在肝癌等恶性肿瘤中表达增高, 而被认为在肝癌的形成中起着重要作用, 但具体作用机制尚不清楚. 本研究通过构建类泛素FAT10高表达肝癌细胞株Hep3B酵母双杂交cDNA文库, 为研究FAT10相互作用蛋白与FAT10基因的结构及功能奠定了重要基础.
肝癌细胞株Hep3B购自中科院上海细胞库; 大肠杆菌DH10B为本实验室保存; 胎牛血清为Hyclone产品; DMEM培养基为Gibco产品; TaKaRa MiniBEST Plasmid Purification Kit、One Shot LA PCRTM Mix、RNAisoTM Plus、cDNA Library Construction Kit、OligotexTM-dT30<Super> mRNA Purification Kit、λ-EcoT14 I Marker以及pHY Marker均为TaKaRa公司产品.
1.2.1 细胞培养: 各组细胞置于DMEM培养液(含100 mL/L胎牛血清), 在37 ℃、50 mL/L CO2条件下培养.
1.2.2 总RNA提取与鉴定: 按照RNAisoTM Plus操作手册说明书进行. 提取RNA 经1.2%琼脂糖凝胶电泳鉴定.
1.2.3 mRNA提取: 按照OligotexTM-dT30<Super >mRNA Purification Kit操作手册说明书进行. 将RNA、bind buffer及OligotexTM-dT30<Super>混合, 然后70 ℃加热3 min, 室温放置10 min, 离心. 去除上清后加入350 μL Wash Buffer, 将OligotexTM-dT30<Super>充分悬浮, 再加入到Spin Column Set的Column Cup中, 离心后加入wash buffer重悬, 重复离心, 最后用50 μL 70 ℃预热的DEPC H2O充分悬浮OligotexTM-dT30<Super>, 离心, 重复2次后回收mRNA.
1.2.4 cDNA合成与文库构建: 根据mRNA浓度确定mRNA模板量. 按TaKaRa的cDNA Library Construction Kit使用手册进行. 利用反转录酶M-MLV与Oligo(dT) Anchor Primer合成1st Strand cDNA(1st Strand cDNA合成时使用5-methyl dCTP); E.coli RNase H使mRNA-1st Strand cDNA杂合体中的RNA形成Nick, 再用E.coli DNA Polymerase和E.coli DNA Ligase将RNA链置换成DNA链, 合成2nd Strand cDNA. T4 DNA Polymerase将双链cDNA末端平滑化再与EcoRⅠ Adaptor连接, XhoⅠ酶切; Spin Column除去短链cDNA后与EcoRⅠ/XhoⅠ酶切的pGADT7载体进行连接(Directional Cloning). Oligo(dT)Anchor Primer: 5'-(GA)10ACTAGTCTCGAG(T)18V-3'(V: A or C or G); EcoRⅠ Adaptor序列: 5'-OH- AATTCGGCACGAGG-3', 3'-GCCGTGCTCCp-5'.
1.2.5 文库质量鉴定: 将原始文库分别稀释10-3与10-6倍, 称为A液与B液, 取1 μL A液加到50 μL的LB液体培养基中, 混匀; 取50 μL与100 μL的B液, 分别涂布于含100 mg/L氨苄青霉素的LB平板上, 室温吸收20 min, 37 ℃培养过夜. 单菌落计数, 确定文库滴度, 公式如下: cfu/mL = A液平板上单菌落数×103×103, cfu/mL = (B液平板上单菌落数P涂板体积)×103×103×103根据文库滴度, 估算进行文库扩增所需平板数. 从原始文库中取10 μL菌液加到15 mL LB液体培养基中, 混匀; 分别取150 μL菌液涂布于100个直径150 mm含100 mg/L氨苄青霉素的LB平板上. 室温吸收1 h, 37 ℃培养18-20 h. 收集菌落加到500 mL LB甘油混合液中, 最后分装于10个50 mL离心管中, 即扩增文库. 将原始文库做1:10 000和1:1 000 000稀释, 转化宿主菌后铺平板, 测定扩增文库的滴度, 其滴度计算, 方法与原始文库滴度计算方法相同.
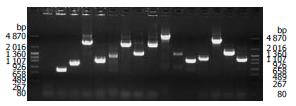
1.2.6 文库的PCR鉴定: 随机挑选了16个菌落单克隆进行扩增, 提取质粒作为模板, 以引物正义: 5'-GGAGTACCCATACGACGTACC-3', 反义: 5'-TATCTACGATTCATCTGCAGC-3'进行PCR扩增, 共30个循环. 用1.0%的琼脂糖凝胶电泳检测.
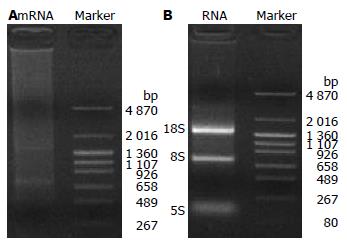
所提总RNA经变性胶电泳显示2条明显的带: 28S与18S, 说明总RNA提取较好, 降解少且分子完整. RNA A260 = 0.120, 其浓度为0.96 g/L, A280 = 0.063, A260/A280 = 1.9, 表明所提RNA纯度高; 电泳结果显示成功分离mRNA, 相对分子质量为: 400-5 000 bp, 符合建库要求(图1).
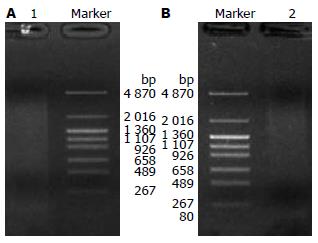
合成的双链cDNA, 5 μL PCR产物进行琼脂糖电泳分析, 结果0.3-5 kb呈弥散状条带, 中间有几条与组织特异性高丰度mRNA相对应的亮带, 短片段去除后, 弥散范围更加集中, 成功合成双链cDNA, 符合建库要求(图2).
文库容量达到1.03×106克隆, 原始文库滴度为2.50×106 cfu/mL, 扩增后的文库滴度为3.60×109 cfu/mL. 插入片段大小分布为0.5-3.5 kb, 平均长度约为2.0 kb(图3).
HCC是我国乃至世界上常见且极具危害性的恶性肿瘤之一. 研究表明, 作为多步骤、多途径肿瘤发生的典型, 肝癌的发生、发展是癌基因的激活与抑癌基因的失活共同作用结果. 泛素/类泛素系统为体内细胞蛋白降解的一个重要途径, 大量的研究表明泛素/类泛素系统功能失调将导致癌基因与抑癌基因的表达异常, 其在肿瘤的发生发展中起着重要作用[1-3].
近年来随着对泛素的研究发现, 真核细胞中也广泛存在着一些同泛素有着部分相同序列或功能相近的蛋白, 他们同样发挥着转录后的修饰作用, 这一类蛋白被称为类泛素家族, 其主要分为两类: 类泛素调节子(ubiquitin-like modifiers, UBLs)与泛素区域蛋白(ubiquitin-domain proteins, UDPs). FAT10属于泛素相关蛋白的生长集落, 参与各种基本的细胞发展, 包括信号转导、蛋白易位及细胞周期调控. Hipp等[4]在研究FAT10作用机制中发现他同泛素一样, 通过标记底物而被特异性识别, 最后被降解, 但又不同于泛素的作用通路. 有研究表明[5]FAT10能与人纺锤体聚集检测点蛋白(MAD2)非共价结合, 学者们利用这一特点, 发现FAT10的过度表达会导致MAD2功能受到抑制, 而MAD2蛋白负责在有丝分裂时保持纺锤体的完整性, 他的功能受到抑制则引起染色体不稳定[6,7], 这正是许多肿瘤发生的重要特征. Lee等[8]在肝癌、胃癌以及其他类型的肿瘤中发现FAT10的蛋白表达要远高于其癌旁和正常组织, 指出FAT10可能在肝癌等恶性肿瘤的形成中起着重要作用. Canaan等[9]通过对FAT10基因缺失小鼠的研究发现, FAT10的缺失会增加肿瘤凋亡的易感性. Zhang等[10]通过对肝癌细胞的体外研究发现, 在野生型p53(wild type p53, wtp53)缺失的Hep3B细胞中FAT10的蛋白表达量要高于含wtp53的HepG2与KB3-1细胞中的FAT10蛋白表达量, 当在Hep3B细胞中导入外源性wtp53后会导致FAT10转录表达能力和启动子活性降低; 反之, 通过RNA干扰减低wtp53的表达量, 则会增强FAT10的启动子活性和蛋白表达. 因此, FAT10在肝癌等恶性肿瘤形成中的作用引起了许多研究者的极大兴趣. 为了研究FAT10的作用通路, 利用酵母双杂交技术, 以FAT10基因作为"诱饵", 以肝癌细胞株Hep3B的cDNA文库作为"猎物", 筛选出FAT10作用通路蛋白与同FAT10作用的底物蛋白.
本研究通过构建肝癌细胞株Hep3B的cDNA文库, 为下一步酵母双杂交筛选提供基础. 在本实验中, 用RNAisoTM Plus试剂与OligotexTM-dT30<Super>磁珠法分别分离提取总RNA和mRNA, 经过电泳证实所获取的mRNA相对分子质量分布范围较广, 无明显降解. 合成的双链cDNA经电泳分析也显示所合成的cDNA是合格的, 这为成功构建cDNA文库打下了良好基础. 一个好的cDNA文库的质量标准应是: 原始文库的重组子数>5×105-5×107; 插入的cDNA片段≥300 bp, 平均>1 000 bp. 此外, 根据Clare-Carbon公式, 要从文库中以99.0%的几率选到一低丰度克隆, 所要求的文库至少应包括1.7×105个克隆数. 本研究构建的肝癌细胞株Hep3B的cDNA库容量达到1.03×106克隆, 原始文库滴度为2.50×106 cfu/mL, 扩增后的文库滴度为3.60×109 cfu/mL. 插入片段大小分布为0.5-3.5 kb, 平均长度约为2.0 kb, 完全达到了构库要求.
泛素/类泛素系统为体内细胞蛋白降解的一个重要途径, 大量的研究表明泛素/类泛素系统功能失调将导致癌基因与抑癌基因的表达异常, 其在肿瘤的发生、发展过程中起着重要作用.
王蒙, 副教授, 中国人民解放军第二军医大学附属东方肝胆外科医院肝外综合治疗一科
Hipp等在研究FAT10作用机制中发现他同泛素一样, 通过标记底物而被特异性识别, 最后被降解, 但又不同于泛素的作用通路.
本文选题较好, 设计合理, 结果可信, 具有较好的参考价值.
编辑: 李军亮 电编:何基才
| 1. | Hoeller D, Hecker CM, Dikic I. Ubiquitin and ubiquitin-like proteins in cancer pathogenesis. Nat Rev Cancer. 2006;6:776-788. [PubMed] [DOI] |
| 2. | Kerscher O, Felberbaum R, Hochstrasser M. Modification of proteins by ubiquitin and ubiquitin-like proteins. Annu Rev Cell Dev Biol. 2006;22:159-180. [PubMed] [DOI] |
| 3. | Kirkin V, Dikic I. Role of ubiquitin- and Ubl-binding proteins in cell signaling. Curr Opin Cell Biol. 2007;19:199-205. [PubMed] [DOI] |
| 4. | Hipp MS, Kalveram B, Raasi S, Groettrup M, Schmidtke G. FAT10, a ubiquitin-independent signal for proteasomal degradation. Mol Cell Biol. 2005;25:3483-3491. [PubMed] [DOI] |
| 5. | Liu YC, Pan J, Zhang C, Fan W, Collinge M, Bender JR, Weissman SM. A MHC-encoded ubiquitin-like protein (FAT10) binds noncovalently to the spindle assembly checkpoint protein MAD2. Proc Natl Acad Sci U S A. 1999;96:4313-4318. [PubMed] [DOI] |
| 6. | Ren J, Kan A, Leong SH, Ooi LL, Jeang KT, Chong SS, Kon OL, Lee CG. FAT10 plays a role in the regulation of chromosomal stability. J Biol Chem. 2006;281:11413-11421. [PubMed] [DOI] |
| 7. | Lim CB, Zhang D, Lee CG. FAT10, a gene up-regulated in various cancers, is cell-cycle regulated. Cell Div. 2006;1:20. [PubMed] [DOI] |
| 8. | Lee CG, Ren J, Cheong IS, Ban KH, Ooi LL, Yong Tan S, Kan A, Nuchprayoon I, Jin R, Lee KH. Expression of the FAT10 gene is highly upregulated in hepatocellular carcinoma and other gastrointestinal and gynecological cancers. Oncogene. 2003;22:2592-2603. [PubMed] [DOI] |
| 9. | Canaan A, Yu X, Booth CJ, Lian J, Lazar I, Gamfi SL, Castille K, Kohya N, Nakayama Y, Liu YC. FAT10/diubiquitin-like protein-deficient mice exhibit minimal phenotypic differences. Mol Cell Biol. 2006;26:5180-5189. [PubMed] [DOI] |