修回日期: 2011-06-24
接受日期: 2011-07-05
在线出版日期: 2011-07-28
目的: 研究肝癌细胞株OY-TES-1启动子的甲基化状态及甲基转移酶抑制剂对其启动子甲基化的影响.
方法: 运用生物信息学在线软件预测OY-TES-1启动子区域及转录因子结合位点; 应用亚硫酸氢盐测序法(bisulfite-sequencing PCR, BSP)分析目的基因CpG位点的甲基化状态; 用甲基转移酶抑制剂5-Aza-CdR干预肝癌细胞株BEL-7404, 比较干预前后OY-TES-1启动子甲基化状态的改变.
结果: 通过软件分析确定了OY-TES-1启动子区域位于-184-+67 bp, 该区域含多个潜在的转录因子结合位点; 在6种肝癌细胞株中OY-TES-1启动子的甲基化频率范围为51.25%-87.92%; 肝癌细胞株BEL-7404经过5-Aza-CdR干预后, 甲基化频率由干预前的87.08%下降至21.25%.
结论: 在检测的肝癌细胞株中, OY-TES-1启动子呈高甲基化状态, 甲基转移酶抑制剂能显著降低肝癌细胞株OY-TES-1启动子甲基化频率.
引文著录: 王炜炜, 范蓉, 罗彬, 郭文文, 张庆梅, 林永达, 肖绍文, 谢小薰. 肝癌细胞中OY-TES-1基因启动子的甲基化状态. 世界华人消化杂志 2011; 19(21): 2274-2278
Revised: June 24, 2011
Accepted: July 5, 2011
Published online: July 28, 2011
AIM: To detect the methylation status of CpG sites in the promoter region of the OY-TES-1 gene in six hepatoma cell lines and to investigate the influence of DNA methyltransferase inhibitor 5-Aza-CdR on methylation status of OY-TES-1 promoter in hepatoma cell line BEL-7404.
METHODS: Online bioinformatic tools were used to predict the promoter region of the OY-TES-1 gene and putative transcription factor binding sites. Bisulfite sequencing PCR (BSP) was applied to detect the methylation status of CpG sites in the promoter region of the OY-TES-1 gene. After treatment of BEL-7404 cells with 5-Aza-CdR, the methylation status of CpG sites in the promoter region of OY-TES-1 gene was detected again.
RESULTS: The promoter region of the OY-TES-1 gene was predicted to be located between -184 bp and +67 bp, which contains several potential transcription factor binding sites. The frequency of promoter methylation of the OY-TES-1 gene ranged between 51.25% and 87.92% in six hepatoma cell lines. 5-Aza-CdR treatment decreased the frequency of promoter methylation of the OY-TES-1 gene from 87.08% to 21.25% in BEL-7404 cells.
CONCLUSION: The OY-TES-1 gene shows relatively high frequency of promoter methylation in six hepatoma cell lines. DNA methyltransferase inhibitor could significantly decrease the frequency of promoter methylation of the OY-TES-1 gene in hepatoma cell line BEL-7404.
- Citation: Wang WW, Fan R, Luo B, Guo WW, Zhang QM, Lin YD, Xiao SW, Xie XX. Methylation status of CpG sites in the promoter region of the OY-TES-1 gene in hepatoma cell lines. Shijie Huaren Xiaohua Zazhi 2011; 19(21): 2274-2278
- URL: https://www.wjgnet.com/1009-3079/full/v19/i21/2274.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i21.2274
癌-睾丸抗原(cancer-testis antigen, CT抗原)是一类能在多种肿瘤表达, 而在除睾丸以外的正常组织不表达或限制性表达的抗原. 研究发现, 许多CT抗原基因的特异性表达与其启动子区域的甲基化状态有关[1], 应用甲基转移酶抑制剂可以使CT抗原基因去甲基化而增加其表达[2]. 尽管目前对大多数CT抗原的功能尚不甚清楚, 但利用其表达特性开展的肿瘤免疫治疗, 如Mage和NY-ESO-1等一些CT抗原已用于临床试验, 在一些肿瘤的治疗中收到了较好的疗效[3,4]. OY-TES-1(又称CT23)是CT抗原家族中的一员, 由Ono等[5]于2001年首次报道. OY-TES-1具有CT抗原的表达特性; 而OY-TES-1特异性的在肿瘤和睾丸组织表达是否与其启动子区域的甲基化状态有关系, 目前尚未见报道. 本研究通过生物信息学在线软件预测OY-TES-1的启动子区域及其潜在的转录因子结合位点, 并运用亚硫酸氢盐测序法(bisulfite-sequencing PCR, BSP)检测其启动子的CpG位点, 观察甲基转移酶抑制剂5-氮杂-2'脱氧胞苷(5-aza-2'-deoxycytidine, 5-Aza-CdR)对其甲基化的影响, 为后续研究OY-TES-1甲基化和表达的关系奠定基础.
人肝癌细胞株QGY-7703、BEL-7404、BEL-7402、QGY-7701、SMMC-7721和HepG-2 (上海细胞所); 胎牛血清(Hyclone公司); 5-Aza-CdR(Sigma公司); UNIQ-10柱式基因组DNA抽提试剂盒、琼脂糖凝胶DNA回收试剂盒(北京天根生化公司); Hotstar酶(TaKaRa公司); PucM-T载体(上海生工); CpGenomeTM DNA Modification Kit试剂盒(Chemicon公司); BSP引物(上海捷瑞生物公司).
1.2.1 生物信息学分析: 获取上游序列: NCBI: http://www.ncbi.nlm.nih.gov/gene; UCSC: http://genome.ucsc.edu/cgi-bin/hgNear; 启动子预测: Proscan: http://www-bimas.cit.nih.gov/molbio/proscan/; 序列比对: http://www.ncbi.nlm.nih.gov/blast/: http://medgen.ugent.be/methBLAST/; 转录因子结合位点预测: Tess: http://www.cbil.upenn.edu/cgi-bin/tess; CpG岛分析: Cpgplot http://www.ebi.ac.uk/emboss/cpgplot; Mehtprimer: http://www.urogene.org/methprimer; BSP引物设计: Methprimer: http://www.urogene.org/methprimer.
1.2.2 细胞培养及药物处理: 含100 mL/L胎牛血清的RPMI 1640, 在37 ℃、50 mL/L CO2饱和湿度条件下培养肝癌细胞株. 取对数生长期的BEL-7404, 按103/孔的细胞数接种于96孔板, 24 h后去上清, 加入含5-Aza-CdR的培养液, 至终浓度为2 μmol/L, 继续培养72 h.
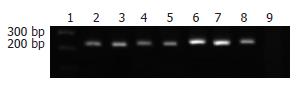
1.2.3 基因组DNA的修饰和BSP: 提取肝癌细胞株基因组DNA, 按试剂盒要求操作; 检测基因组DNA的纯度和含量, -20 ℃保存DNA. 取1 μg基因组DNA, 按照CpGenomeTM DNA Modification Kit试剂盒进行亚硫酸氢盐修饰, 修饰后DNA直接用于PCR. OY-TES-1的引物序列为: 上游: 5'-TTTTGGTTATTTTAGGGTTTTGAGA-3'; 下游: 5'-ACAAATATAAACCTCTCACCCTTCA-3', PCR产物为237 bp. 反应体系为: 修饰后DNA 1 μL, 10×PCR缓冲液2.5 μL, dNTP 2 μL, HotStar 0.2 μL, 上下游引物各1 μL. 反应条件: 95 ℃预变性5 min, 94 ℃变性30 s, 50 ℃退火30 s, 72 ℃延伸30 s, 共计35循环, 最终72 ℃延伸10 min, PCR产物分子量为237 bp, PCR反应完毕后进行1.5%琼脂糖凝胶电泳, 并通过凝胶回收和纯化PCR产物. 将纯化后PCR产物与PucM-T质粒连接, 转化大肠杆菌DH5α菌株, 接种于LB-x-gal琼脂平板, 进行蓝白斑筛选, 每个肝癌细胞株挑取10个单菌落进行培养扩增, 提取质粒, PCR鉴定, 将验证正确的标本送公司进行DNA测序. 对测序结果进行CpG位点的甲基化状态分析.
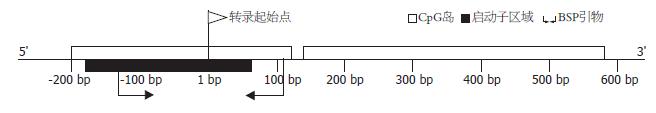
2.1.1 OY-TES-1启动子位置: 首先从GenBank和UCSC获取OY-TES-1基因的5'上游调控序列(共3 124 bp), 然后通过Proscan确定OY-TES-1启动子区域为其转录起始点-184 bp-+67 bp, 共251 bp(Promoter Score: 83.73; Promoter Cutoff:53.0000)(图1). 此区域内无典型的TATA和CAAT盒, 但富含GC盒; 通过TESS分析, 发现该启动子序列中存在多个SP1转录因子结合位点.
2.1.2 OY-TES-1基因CpG岛: 对OY-TES-1基因全长序列进行CpG岛的分析, cpgplot与methprimer分析结果一致, 即OY-TES-1全长存在2个CpG岛(观测值与期望值的比率>0.60; C含量+G含量>50%; 长度>200 bp)[6]; 第1个CpG岛位于-200 bp-+126 bp, 包含第1外显子及部分第1内含子, 第2个位于CpG岛+137 bp-+587 bp, 包含第1内含子和第2外显子的大部分区域.
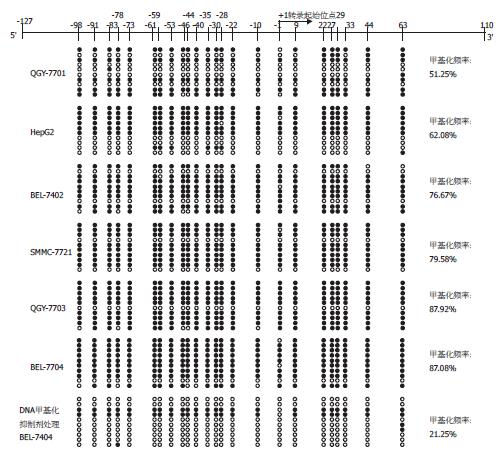
2.1.3 OY-TES-1启动子BSP引物: 用methprimer在OY-TES-1启动子区域设计BSP引物, 其引物位于OY-TES-1基因转录起始点-127 bp-+110 bp之间和第1个CpG岛内, 该区域共含24个CpG位点(图1, 2).
用OY-TES-1启动子BSP引物对亚硫酸氢盐修饰的DNA进行PCR扩增, 获得的产物与预期分子量一致(图3). 为了避免由于亚硫酸氢盐修饰不完全所造成的误差, 我们选用所有单个C已变成T的克隆进行methblast比对分析; 结果如图2所示, 各肝癌细胞株的总甲基化频率分别为51.25%(QGY-7701); 62.08%(HepG-2); 76.67%(BEL-7402); 79.58%(SMMC-7721); 87.08%(BEL-7404)和87.92%(QGY-7703); 经5-Aza-CdR处理后的BEL-7404其总甲基化频率为21.25%, 与5-Aza-CdR处理前比较, 总甲基化频率明显的降低.
目前已有研究发现, OY-TES-1 mRNA能在多种肿瘤组织中表达, 而在正常组织中基本不表达(除睾丸外); 其相应的抗体可出现在多种肿瘤患者血清中, 而正常人为阴性[7,8]; 用OY-TES-1抗原肽诱导的细胞毒性T淋巴细胞(cytotoxic T lymphocyte, CTL)能特异性杀伤OY-TES-1 mRNA阳性的肺癌细胞[9], 这些研究结果提示OY-TES-1可能具有潜在的肿瘤免疫治疗价值. 本课题组前期的研究发现, 在肝癌组织中OY-TES-1 mRNA表达量显著高于对应的癌旁组织及正常肝组织; 对这些肝癌组织进行免疫组织化学染色, 我们发现OY-TES-1蛋白的分布不均一, 呈现明显的异质性[10]. 我们推测OY-TES-1蛋白表达异质性的产生原因, 可能也像许多CT抗原一样, 与其启动子甲基化状态有关. 通过生物信息学分析, 我们发现OY-TES-1启动子含多个CpG位点, 进一步对其中的24个CpG位点进行甲基化分析, 结果显示所检测的6种肝癌细胞株甲基化频率为51.25%-87.92%, 呈现一种较高的甲基化状态.
甲基转移酶抑制剂(5-Aza-CdR等)通过与DNA甲基转移酶共价结合, 形成复合物进而抑制该酶的活性, 使基因去甲基化而促进基因的表达. 在CT抗原的肿瘤免疫治疗中, 研究人员也尝试配合使用DNA甲基转移酶抑制剂, 以诱导不表达CT抗原的肿瘤细胞重新表达CT抗原, 同时还可以上调原来呈低表达的CT抗原, 使其表达量增加[11-13]. 目前, 5-Aza-CdR等已用于一些肿瘤的临床治疗, 不少研究报道肯定了他在肿瘤免疫治疗中的作用. 为了解DNA甲基转移酶抑制剂是否能使肝癌细胞由高甲基化状态转变为低甲基化状态, 本研究用5-Aza-CdR培养肝癌细胞株BEL-7404, 结果显示OY-TES-1启动子总甲基化频率从87.08%下降至21.25%, 这说明DNA甲基转移酶抑制剂能诱导该细胞株的OY-TES-1启动子去甲基化, 下一步需检测是否能引起其蛋白质表达上调.
我们的生物信息学分析结果还显示了OY-TES-1启动子区域富含刺激转录蛋白1(stimulatory protein 1, Sp1)结合位点. 已有研究报道Sp1与靶基因启动子结合可促进靶基因的转录活化[14], 然而, 靶基因启动子上CpG位点的甲基化又可抑制Sp1的转录激活作用, 导致基因表达沉默[15,16]; 因此, 靶基因启动子的甲基化状态与Sp1如何发挥作用有密切的关系. 鉴于OY-TES-1启动子的结构特点, 我们推测OY-TES-1的表达调控在甲基化作用下也可能涉及到Sp1的参与, 遗憾的是就这一问题目前尚无任何文献报道, 下一步需要通过相应的分子生物学实验来验证这一推测.
OY-TES-1是一种限制性表达于正常组织、选择性高表达于肿瘤组织的CT抗原. 目前利用CT抗原的表达特性开展的肿瘤免疫治疗, 已用于临床试验并收到较好的疗效. 因此, 有必要研究OY-TES-1的特异性表达与哪些因素相关, 从而为后继研究奠定基础.
施晓雷, 副主任医师, 南京大学医学院附属鼓楼医院肝胆外科
De Smet等研究发现, 癌-睾丸抗原MAGE-A1的激活与该基因转录起始点上、下游200-1 500 bp区域的甲基化水平有关, 而且该区域内CpG位点去甲基化是该基因转录激活的必须条件.
目前尚无OY-TES-1启动子区域及其甲基化状态的相关报道. 本研究运用生物信息学法预测OY-TES-1启动子区域, BSP法分析OY-TES-1启动子CpG位点的甲基化状态; 并用DNA甲基转移酶抑制剂干预肝癌细胞株, 比较干预前后OY-TES-1启动子甲基化状态的改变.
本文方法确切, 结果可靠, 具有较高的科学价值.
编辑: 李薇 电编:何基才
| 1. | Shichijo S, Yamada A, Sagawa K, Iwamoto O, Sakata M, Nagai K, Itoh K. Induction of MAGE genes in lymphoid cells by the demethylating agent 5-aza-2'-deoxycytidine. Jpn J Cancer Res. 1996;87:751-756. [PubMed] [DOI] |
| 2. | Grunau C, Sanchez C, Ehrlich M, van der Bruggen P, Hindermann W, Rodriguez C, Krieger S, Dubeau L, Fiala E, De Sario A. Frequent DNA hypomethylation of human juxtacentromeric BAGE loci in cancer. Genes Chromosomes Cancer. 2005;43:11-24. [PubMed] [DOI] |
| 3. | Dutoit V, Taub RN, Papadopoulos KP, Talbot S, Keohan ML, Brehm M, Gnjatic S, Harris PE, Bisikirska B, Guillaume P. Multiepitope CD8(+) T cell response to a NY-ESO-1 peptide vaccine results in imprecise tumor targeting. J Clin Invest. 2002;110:1813-1822. [PubMed] |
| 4. | Lonchay C, van der Bruggen P, Connerotte T, Hanagiri T, Coulie P, Colau D, Lucas S, Van Pel A, Thielemans K, van Baren N. Correlation between tumor regression and T cell responses in melanoma patients vaccinated with a MAGE antigen. Proc Natl Acad Sci U S A. 2004;101 Suppl 2:14631-14638. [PubMed] [DOI] |
| 5. | Ono T, Kurashige T, Harada N, Noguchi Y, Saika T, Niikawa N, Aoe M, Nakamura S, Higashi T, Hiraki A. Identification of proacrosin binding protein sp32 precursor as a human cancer/testis antigen. Proc Natl Acad Sci U S A. 2001;98:3282-3287. [PubMed] [DOI] |
| 6. | Gardiner-Garden M, Frommer M. CpG islands in vertebrate genomes. J Mol Biol. 1987;196:261-282. [PubMed] [DOI] |
| 7. | 范 蓉, 黄 巍, 肖 绍文, 罗 彬, 何 少健, 罗 国容, 谢 小薰. OY-TES-1在肝细胞癌中的表达及抗体血清学分析. 世界华人消化杂志. 2009;17:3307-3312. [DOI] |
| 8. | Tammela J, Uenaka A, Ono T, Noguchi Y, Jungbluth AA, Mhawech-Fauceglia P, Qian F, Schneider S, Sharma S, Driscoll D. OY-TES-1 expression and serum immunoreactivity in epithelial ovarian cancer. Int J Oncol. 2006;29:903-910. [PubMed] |
| 9. | Okumura H, Noguchi Y, Uenaka A, Aji T, Ono T, Nakagawa K, Aoe M, Shimizu N, Nakayama E. Identification of an HLA-A24-restricted OY-TES-1 epitope recognized by cytotoxic T-cells. Microbiol Immunol. 2005;49:1009-1016. [PubMed] |
| 10. | 范 蓉, 余 良, 肖 绍文, 何 少健, 罗 彬, 黄 绍明, 罗 国容, 谢 小薰. 精子蛋白sp32/OY-TES-1mRNA及其蛋白在正常组织表达的探讨. 解剖学报. 2007;38:675-680. |
| 11. | Honda T, Tamura G, Waki T, Kawata S, Terashima M, Nishizuka S, Motoyama T. Demethylation of MAGE promoters during gastric cancer progression. Br J Cancer. 2004;90:838-843. [PubMed] [DOI] |
| 12. | Jang SJ, Soria JC, Wang L, Hassan KA, Morice RC, Walsh GL, Hong WK, Mao L. Activation of melanoma antigen tumor antigens occurs early in lung carcinogenesis. Cancer Res. 2001;61:7959-7963. [PubMed] |
| 13. | Mellor AL, Chandler P, Baban B, Hansen AM, Marshall B, Pihkala J, Waldmann H, Cobbold S, Adams E, Munn DH. Specific subsets of murine dendritic cells acquire potent T cell regulatory functions following CTLA4-mediated induction of indoleamine 2,3 dioxygenase. Int Immunol. 2004;16:1391-1401. [PubMed] [DOI] |
| 15. | Clark SJ, Harrison J, Molloy PL. Sp1 binding is inhibited by (m)Cp(m)CpG methylation. Gene. 1997;195:67-71. [PubMed] [DOI] |
| 16. | Zhang D, Ai D, Tanaka H, Hammock BD, Zhu Y. DNA methylation of the promoter of soluble epoxide hydrolase silences its expression by an SP-1-dependent mechanism. Biochim Biophys Acta. 2010;1799:659-667. [PubMed] |