修回日期: 2009-12-30
接受日期: 2010-01-11
在线出版日期: 2010-02-28
目的: 探讨hMSH2基因启动子区5'CpG岛高甲基化在胃癌发生过程中的作用.
方法: 应用甲基化特异性PCR(methylation specific PCR, MSP)方法检测胃癌及非癌组织中hMSH2基因启动子区甲基化状态.
结果: 40例胃癌中hMSH2基因启动子区高甲基化24例(60%), 其癌旁黏膜组织中有15例(37.5%)发生甲基化, 14例慢性萎缩性胃炎组织中有5例(35.7%)发生甲基化, 6例慢性浅表性胃炎组织中未见甲基化. 四组甲基化水平相比, 差别有统计意义(P<0.05). 胃癌组甲基化水平高于癌旁组, 差别有统计意义(P<0.05). 癌旁组、慢性萎缩性胃炎组、慢性浅表性胃炎组三组甲基化水平相比, 差别无统计意义. 胃癌各临床病理参数组之间相比差别无统计意义.
结论: 胃癌组织中hMSH2基因启动子区高甲基化可能是导致其错配修复功能缺陷的重要原因之一; 而错配修复功能缺陷在胃癌的发生中起着重要作用, 但可能与其发展关系不大.
引文著录: 毛庆东, 刘希双, 杨堃. 错配修复基因hMSH2启动子甲基化与胃癌的关系. 世界华人消化杂志 2010; 18(6): 606-609
Revised: December 30, 2009
Accepted: January 11, 2010
Published online: February 28, 2010
AIM: To detect the role of methylation of the 5'CpG island located in the promoter region of the human mutS homolog-2 (hMSH2) gene in the pathogenesis of gastric carcinoma.
METHODS: The methylation of the hMSH2 promoter was examined by methylation-specific polymerase chain reaction (MSP) in 40 gastric carcinoma specimens, 40 matched cancer-adjacent mucosa specimens, 14 chronic atrophic gastritis (CAG) specimens, and 6 chronic superficial gastritis (CSG) specimens.
RESULTS: The methylation of the hMSH2 promoter was detected in 24 out of the 40 (60%) gastric cancer specimens, 15 out of the 40 (37.5%) cancer-adjacent mucosa specimens, and 5 out of the 14 (35.7%) CAG specimens. No methylation was detected in 6 CSG specimens. The rate of hMSH2 promoter methylation was significantly higher in gastric cancer tissue than in non-carcinoma tissue (P < 0.05). No significant differences were detected in the rates of hMSH2 promoter methylation among cancer-adjacent mucosa specimens, CAG specimens and CSG specimens. The rate of hMSH2 promoter methylation is not correlated with the clinicopathological parameters of gastric cancer.
CONCLUSION: The hMSH2 promoter methylation may be a main mechanism responsible for mismatch repair (MMR) dysfunction that is involved in the development, but not the progression, of gastric cancer.
- Citation: Mao QD, Liu XS, Yang K. Relationship between mismatch repair gene hMSH2 promoter methylation and gastric carcinoma. Shijie Huaren Xiaohua Zazhi 2010; 18(6): 606-609
- URL: https://www.wjgnet.com/1009-3079/full/v18/i6/606.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i6.606
胃癌是消化系最常见的恶性肿瘤, 其发生是一个多步骤, 多因素参与进行性发展的过程. 作为多基因相关性肿瘤, 目前认为多种基因在不同阶段参与了胃癌的发生, 已有研究发现, 肿瘤抑制基因启动子区甲基化与肿瘤的发生密切相关[1,2]. 本研究采用甲基化特异性PCR(methylation specific PCR, MSP)方法, 检测40例胃癌组织及其癌旁黏膜组织和20例慢性胃炎组织中hMSH2基因启动子区的甲基化状态, 探讨hMSH2基因启动区高甲基化与胃癌发生的关系.
2008-01/2008-08自青岛大学医学院附属医院普外科手术切除的胃癌组织40例, 男29例, 女11例, 年龄31-81(中位年龄57)岁, 男女比2.6:1, 经病理诊断低分化腺癌32例, 中分化腺癌6例, 印戒细胞癌2例. 患者术前均未接受放化疗和免疫治疗, 每例均取相应癌旁组织, 即距离癌组织边缘5 cm的组织, 经病理诊断慢性浅表性胃炎13例, 慢性萎缩性胃炎伴肠上皮化生27例. 胃镜普通活检钳取20例慢性胃炎胃窦中部组织(慢性浅表性胃炎6例, 慢性萎缩性胃炎14例)4块, 男4例, 女16例, 年龄26-59(中位年龄47)岁. 组织取出立即置于液氮冻存, 迅速转移至-70 ℃超低温冰箱冻存备用. 组织DNA提取使用上海华舜生物有限公司的柱式小量组织/细胞基因组DNA抽提试剂盒(W6501). 甲基化修饰使用CHEMICON的CpGenomeTM DNA Modification kit S7820. 引物序列参考文献[3], 由上海生工生物工程技术有限公司合成(表1).
| hMSH2引物 | 引物序列(5'-3') | 产物长度(bp) | 温度(℃) | |
| 甲基化(M) | 正义 | TCGTGGTCGGACGTCGTTC | 132 | 60 |
| 反义 | CAACGTCTCCTTCGACTACACCG | |||
| 非甲基化(U) | 正义 | GGTTGTTGTGGTTGGATGTTGTTT | 137 | 60 |
| 反义 | CAACTACAACATCTCCTTCAACTACACCA |
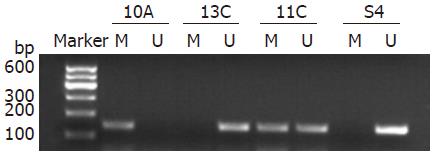
MSP检测hMSH2基因甲基化: 组织基因组DNA的提取按照DNA抽提试剂盒说明操作. 甲基化修饰严格按照试剂盒说明书进行. 处理后DNA样本保存于-70 ℃超低温冰箱备用. 然后用PCR仪扩增目的基因, 同一标本用甲基化引物和非甲基化引物分别扩增, 引物序列、目的片段大小见表1. 25 μL PCR反应体系: TaKaRa Taq HS(5 U/μL)0.125 μL, 10×PCR Buffer(Mg2+ Plus)2.5 μL, dNTP Mixture(各2.5 mmol/L)2 μL, Forward Primer(6 μmol/L)1 μL, Reverse Primer (6 μmol/L)1 μL, 甲基化修饰后DNA 10 μL, dH2O 8.375 μL. PCR扩增条件参考文献[4]并行梯度PCR找出最佳条件: 94 ℃预变性3 min, 94 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 40 s, 共35个循环, 最后72 ℃延伸10 min. 取PCR产物5 μL, 与1 μL 6×Loading Buffer混合, 进行琼脂糖凝胶(浓度20 g/L)电泳, 紫外光凝胶成像系统观察结果. 若启动子区发生甲基化, 则甲基化特异性引物可扩增出相应条带; 若未发生甲基化, 则非甲基化特异性引物可扩增出相应大小目的条带.
统计学处理 采用SPSS13.0软件进行统计学分析. 甲基化频率的差异及其与肿瘤临床病理特征之间的关系采用χ2检验. 以P<0.05为差别有统计学意义.
胃癌组、癌旁组、慢性萎缩性胃炎组、慢性浅表性胃炎组四组甲基化水平相比, 差别有统计意义(χ2 = 9.946, P = 0.019); 胃癌组甲基化水平高于癌旁组, 差别有统计学意义(χ2 = 4.053, P = 0.044); 癌旁组、慢性萎缩性胃炎组、慢性浅表性胃炎组三组甲基化水平相比, 差别无统计学意义(χ2 = 3.348, P = 0.187); 慢性萎缩性胃炎组甲基化水平高于慢性浅表性胃炎组, 但差别无统计学意义(P = 0.129, 表2). 各组织基因组DNA甲基化修饰行MSP, 琼脂糖凝胶电泳结果见图1.
| 分组 | n | hMSH2甲基化 n(%) |
| 胃癌组 | 40 | 24(60.0) |
| 癌旁组 | 40 | 15(37.5) |
| 慢性萎缩性胃炎组 | 14 | 5(35.7) |
| 慢性浅表性胃炎组 | 6 | 0(0) |
hMSH2基因启动子区甲基化状态与胃癌患者的年龄、性别及肿瘤的部位、大小、TNM临床分期之间无明显联系(表3).
| 病理参数 | n | hMSH2甲基化n(%) | χ2值 | P值 |
| 年龄(岁) | ||||
| ≥60 | 18 | 11(61) | 0.017 | 0.897 |
| <60 | 22 | 13(59) | ||
| 性别 | ||||
| 男 | 29 | 17(59) | 0.000 | 1.000 |
| 女 | 11 | 7(64) | ||
| 癌肿部位 | ||||
| 胃窦 | 22 | 15(68) | 1.364 | 0.243 |
| 胃体底 | 18 | 9(50) | ||
| 肿瘤大小(cm) | ||||
| ≥5 | 23 | 11(48) | 3.342 | 0.068 |
| <5 | 17 | 13(76) | ||
| 临床分期 | ||||
| Ⅰ、Ⅱ期 | 11 | 6(55) | 0.188 | 0.665 |
| Ⅲ、Ⅳ期 | 29 | 18(62) |
错配修复基因(mismatch repair gene, MMR)是人体内一组高度保守的管家基因, 具有修复DNA碱基错配、增强DNA复制忠实性、维持基因组稳定性和降低自发性突变的功能[5,6]. 目前人类的MMR系统含有9个错配修复基因. MMR发生异常, 则其错配修复功能发生缺陷无法修复DNA复制中出现的碱基错配, 产生遗传不稳定性, 导致肿瘤易感. 引起错配修复基因表达缺陷的原因除基因突变外, 基因启动区的高甲基化也是其主要原因之一[7]. 肿瘤抑制基因CpG岛甲基化导致其转录失活, 是肿瘤发生高度一致的特征[8].
hMSH2定位于染色体2p22-21或2p16-15, 基因组DNA全长(不包括启动子)为73 kb, 含16个外显子, cDNA全长为3 116 bp, 其中开放阅读框架为2 727 bp, 编码909个氨基酸序列, 与966个氨基酸序列的酿酒酵母菌性MSH2蛋白有41%同源性, 且高度保守片段的同源性达85%, 位于密码子573-764处[9,10]. hMSH2与hMSH6或hMSH3形成二聚体复合物, hMSH2与hMSH6形成的二聚体复合物与单个碱基或一个碱基的缺失/插入错配位置结合, hMSH2与hMSH3形成的二聚体复合物与2-4个碱基的缺失/插入错配位置结合. 由此可见, 与错配结合有关的hMSH2是必须的, 而hMSH6和hMSH3则有一定的互补性. 因此, 研究了解hMSH2表达失活的机制及其与胃癌发生的关系有十分重要的临床意义. 本实验应用MSP技术对胃癌组织, 癌旁组织和慢性胃炎组织中的hMSH2启动子甲基化情况进行检测.
有研究发现, 启动子甲基化是hMLH1和hMSH2表达下调的主要机制[11]. 本研究结果显示, 40例胃癌组织中有24例(60%)hMSH2启动子区发生甲基化, 甲基化的比例明显高于非癌组织, 差别有统计学意义(P<0.05). 支持hMSH2基因由于启动子区髙甲基化导致其基因沉默, 引起表达下调, 不能发挥其应有错配修复功能, 从而引起肿瘤易感发生的机制假设. 亦有研究显示, hMLH1和hMSH2启动子髙甲基化参与了口腔鳞状细胞癌和葡萄胎的发生[12,13], 同样也有研究发现原发性乳癌患者中有16% hMSH2启动子区高甲基化[14]. 上述事实可从另一方面补充说明由于启动子区甲基化导致肿瘤发生的机制. 实验结果中, 40例胃癌癌旁组织中, 有15例(37.5%)hMSH2启动子区发生甲基化, 低于胃癌组织启动子区甲基化水平, 差别有统计学意义(P<0.05). 癌旁组织细胞与癌细胞处于同一内外环境中, 并具有相同的遗传物质, 因此被认为是肿瘤的易发组织[15]. 本实验显示, 癌旁组织启动子区hMSH2甲基化水平介于胃癌组和慢性浅表性胃炎组之间, 说明癌旁组织处于胃癌组织与慢性浅表性胃炎组织之间的一种中间状态, 从而推断hMSH2甲基化程度逐渐累积至一定水平可导致基因表达的改变即基因沉默, 促使组织向肿瘤的转变.
癌旁组甲基化水平虽高于慢性萎缩性胃炎组, 但差别无统计意义. 5例慢性萎缩性胃炎(35.7%)hMSH2启动子区发生甲基化, 慢性浅表性胃炎hMSH2启动子区未见甲基化, 前者甲基化水平高于后者, 但差别无统计意义. 上述结果说明慢性萎缩性胃炎hMSH2启动子区甲基化水平已接近癌旁组水平, 两者在向胃癌的转化几率方面已无太大差别. 慢性萎缩性胃炎组织中hMSH2启动子区出现低水平的甲基化, 从另一层面说明慢性萎缩性胃炎作为胃癌的癌前疾病, 其基因层面已经出现一定的改变, 但该结论尚需大样本研究资料证实.
总之, 慢性浅表性胃炎hMSH2启动子区尚未见甲基化, 慢性萎缩性胃炎(35.7%)和胃癌癌旁组织(37.5%)已出现一定水平的甲基化, 胃癌组织出现较高水平的甲基化(60%), 说明异常基因的积累最终导致肿瘤的发生, 因此检测hMSH2启动子区甲基化的水平对于胃癌的预防有一定的临床意义. 但hMSH2启动子区甲基化水平与患者的年龄、性别以及肿瘤的部位、大小、临床分期无关, 提示hMSH2启动子区甲基化水平可能只与胃癌的发生有关, 而与其发展关系不大.
胃癌的发生是一个多因素, 多步骤, 多阶段发展的过程, 亦是一个多基因参与的复杂过程, 其中包括癌基因的激活、抑癌基因的失活、错配修复基因的表达异常. 就错配修复基因发生启动区甲基化而言, 已涉及众多基因, 虽然其中研究较多较为透彻的有hMLH1基因, hMSH2基因也有一定研究, 但其具体作用机制十分复杂尚需进一步研究论证. 同时亦需大样本研究证实其特异性和敏感性.
细胞错配修复功能缺陷, 不能及时修复DNA复制时的错误, 会导致肿瘤相关基因基因组DNA突变的累积, 最终可导致肿瘤发生. DNA错配修复基因系统中任何一种基因突变, 都会引起错配修复功能缺陷, 产生遗传不稳定性, 导致肿瘤易感. 近来研究发现, 错配修复基因表达缺陷的原因不仅有基因突变, 基因启动子区的高甲基化也是其主要原因之一.
陈卫昌, 教授, 苏州大学附属第一医院消化内科; 孙学英, 教授, 哈尔滨医科大学第一附属医院黑龙江省肝脾外科中心
DNA甲基化导致错配修复缺陷是胃癌发病机制中一个重要途径. 目前研究较多的是hMLH1基因, hMSH2基因也有一定研究, 但研究结果不一致, 有研究说胃癌组织中hMSH2的表达正常, 亦有报道其表达缺失或减少.
大部分的研究表明胃癌患者存在hMLH1启动子甲基化及其表达消失, 亦有学者对hMSH2的表达情况进行了检测. Leung等在对MSI-H胃癌患者研究中发现hMLH1表达消失, 而hMSH2的表达却正常. Halling等在MSI胃癌中却观察到hMSH2表达消失. Zhang等发现hMSH2 mRNA表达减少. Grogg等在对17例MSI检测中却发现hMSH2均正常表达.
本文测定从慢性胃炎到胃癌发生过程中的几个重要阶段中的hMSH2启动子区甲基化水平, 发现hMSH2启动子区甲基化水平有逐渐升高的趋势, 推测hMSH2启动子区高甲基化与胃癌发生有一定的关系.
胃癌缺乏特异的诊断手段, 大部分发现时已是晚期, 因此早期诊断是治疗的前提. 本文发现慢性萎缩性胃炎组织中hMSH2启动子区已出现一定水平的甲基化, 胃癌组织则出现高水平的甲基化, 推测慢性萎缩性胃炎有进一步发展至胃癌的可能, 为胃癌的早期诊断提供一个思路. 同时, 推测对于已发生甲基化的DNA组织进行去甲基化治疗有可能阻断其向胃癌的发展, 为基因治疗提供一定的帮助.
本研究设计合理, 结果可靠, 有一定的可读性.
编辑: 李军亮 电编:何基才
| 1. | Cai JC, Liu D, Zhang HP, Zhong S, Xia NS. [Frequent promoter hypermethylation of several tumor suppressor genes in gastric carcinoma and foveolar epithelium]. Zhonghua Zhongliu Zazhi. 2007;29:510-513. [PubMed] |
| 2. | Yoshikawa H. [DNA methylation and cancer]. Gan To Kagaku Ryoho. 2007;34:145-149. [PubMed] |
| 3. | Herman JG, Umar A, Polyak K, Graff JR, Ahuja N, Issa JP, Markowitz S, Willson JK, Hamilton SR, Kinzler KW. Incidence and functional consequences of hMLH1 promoter hypermethylation in colorectal carcinoma. Proc Natl Acad Sci U S A. 1998;95:6870-6875. [PubMed] [DOI] |
| 4. | Mokarram P, Naghibalhossaini F, Saberi Firoozi M, Hosseini SV, Izadpanah A, Salahi H, Malek-Hosseini SA, Talei A, Mojallal M. Methylenetetrahydrofolate reductase C677T genotype affects promoter methylation of tumor-specific genes in sporadic colorectal cancer through an interaction with folate/vitamin B12 status. World J Gastroenterol. 2008;14:3662-3671. [PubMed] [DOI] |
| 5. | Jacob S, Praz F. DNA mismatch repair defects: role in colorectal carcinogenesis. Biochimie. 2002;84:27-47. [PubMed] [DOI] |
| 6. | Kulke MH, Thakore KS, Thomas G, Wang H, Loda M, Eng C, Odze RD. Microsatellite instability and hMLH1/hMSH2 expression in Barrett esophagus-associated adenocarcinoma. Cancer. 2001;91:1451-1457. [PubMed] [DOI] |
| 7. | Wheeler JM, Beck NE, Kim HC, Tomlinson IP, Mortensen NJ, Bodmer WF. Mechanisms of inactivation of mismatch repair genes in human colorectal cancer cell lines: the predominant role of hMLH1. Proc Natl Acad Sci U S A. 1999;96:10296-10301. [PubMed] [DOI] |
| 8. | Hong SH, Kim HG, Chung WB, Kim EY, Lee JY, Yoon SM, Kwon JG, Sohn YK, Kwak EK, Kim JW. DNA hypermethylation of tumor-related genes in gastric carcinoma. J Korean Med Sci. 2005;20:236-241. [PubMed] [DOI] |
| 9. | Fishel R, Lescoe MK, Rao MR, Copeland NG, Jenkins NA, Garber J, Kane M, Kolodner R. The human mutator gene homolog MSH2 and its association with hereditary nonpolyposis colon cancer. Cell. 1993;75:1027-1038. [PubMed] [DOI] |
| 10. | Lynch HT, Watson P, Shaw TG, Lynch JF, Harty AE, Franklin BA, Kapler CR, Tinley ST, Liu B, Lerman C. Clinical impact of molecular genetic diagnosis, genetic counseling, and management of hereditary cancer. Part II: Hereditary nonpolyposis colorectal carcinoma as a model. Cancer. 1999;86:2457-2463. [PubMed] [DOI] |
| 11. | Hsu HS, Wen CK, Tang YA, Lin RK, Li WY, Hsu WH, Wang YC. Promoter hypermethylation is the predominant mechanism in hMLH1 and hMSH2 deregulation and is a poor prognostic factor in nonsmoking lung cancer. Clin Cancer Res. 2005;11:5410-5416. [PubMed] [DOI] |
| 12. | Czerninski R, Krichevsky S, Ashhab Y, Gazit D, Patel V, Ben-Yehuda D. Promoter hypermethylation of mismatch repair genes, hMLH1 and hMSH2 in oral squamous cell carcinoma. Oral Dis. 2009;15:206-213. [PubMed] [DOI] |
| 13. | Chen H, Ye D, Xie X, Lu W, Zhu C, Chen X. Mismatch repair gene promoter methylation and expression in hydatidiform moles. Arch Gynecol Obstet. 2005;272:35-39. [PubMed] [DOI] |
| 14. | Naqvi RA, Hussain A, Deo SS, Kukreti H, Chauhan M, Sarin R, Saxena A, Asim M, Shukla NK, Husain SA. Hypermethylation analysis of mismatch repair genes (hmlh1 and hmsh2) in locally advanced breast cancers in Indian women. Hum Pathol. 2008;39:672-680. [PubMed] [DOI] |