修回日期: 2010-11-07
接受日期: 2010-11-10
在线出版日期: 2010-12-18
目的: 研究从不同病程的大肠癌患者血清蛋白指纹图谱中筛选出的特征蛋白质组, 以期对大肠癌患者进行预后判断以及帮助制定个体化的医疗措施.
方法: 用高通量高灵敏的表面增强激光解析电离飞行时间质谱技术(SELDI-TOF-MS)检测大肠癌组(45例)、术后预后良好组(术后无复发和无转移14例)、术后预后不良组(术后复发或转移13例)、肠道良性疾病组(24例)和健康人群组(155例)的血清蛋白指纹图谱, 利用Biomarker Wizard软件进行差异蛋白质筛选, 人工神经网络技术建立大肠癌的诊断模型和预后模型, 并检验模型的诊断效率.
结果: 筛选出7个表达有明显差异的标志蛋白质(P<0.01), 其相对分子质量为4 955 Da、5 325 Da、5 890 Da、6 615 Da、7 739 Da、8 109 Da、8 575 Da. 利用这7个标志蛋白质建立人工神经网络大肠癌的诊断模型, 再利用相对分子质量为4 955 Da、5 325 Da、5 890 Da、6 615 Da、7 739 Da的5个差异蛋白建立人工神经网络大肠癌的预后模型. 其中诊断模型对大肠癌的诊断灵敏度和特异度分别为82.22%和80.45%, 阴性预测值94.74%, 阳性预测值51.39%, 准确度为80.80%. 预后模型对大肠癌术后复发或转移的诊断符合率为62.96%.
结论: 从SELDI-TOF-MS血清蛋白指纹图谱中找到了大肠癌不同病程时期的标志蛋白质组, 并建立了大肠癌的分类树模型.
引文著录: 王专, 李小琼, 王开正, 邓明明, 徐亮. 血清蛋白指纹图与大肠癌预后诊断. 世界华人消化杂志 2010; 18(35): 3745-3751
Revised: November 7, 2010
Accepted: November 10, 2010
Published online: December 18, 2010
AIM: To identify differentially expressed proteins for diagnosis and prognosis evaluation of colorectal cancer by serum protein fingerprint in colorectal cancer patients.
METHODS: Serum protein fingerprinting was performed by surface-enhanced laser desorption ionization time-of-flight mass spectrometry (SELDI-TOF-MS) in 45 colorectal cancer patients, 14 colorectal cancer having a good prognosis (no postoperative recurrence and metastasis), 13 colorectal cancer patients having a poor prognosis (having recurrence or metastasis), 24 patients with benign gastrointestinal disease, and 155 healthy controls. The Biomarker Wizard software was used to identify differential proteins. Two respective artificial neural network (ANN) models were developed for diagnosis and prognosis evaluation of colorectal cancer.
RESULTS: Seven proteins that displayed significant differential expression were identified (all P < 0.01), and their molecular weight was 4 955 Da, 5 325 Da, 5 890 Da, 6 615 Da, 7 739 Da, 8 109 Da, and 8 575 Da, respectively. Using these seven protein markers, we developed an artificial neural network model for diagnosis of colorectal cancer. Furthermore, five proteins that had a molecular weight of 4 955 Da, 5 325 Da, 5 890 Da, 6 615 Da, and 7 739 Da were used to develop an artificial neural network model for evaluation of the prognosis of colorectal cancer. The sensitivity, specificity, negative predictive value, positive predictive value, and accuracy of the diagnostic model were 82.22%, 80.45%, 94.74%, 51.39% and 80.80%, respectively. The coincidence rate of the prognostic model for evaluation of recurrence and metastasis was 62.96%.
CONCLUSION: SELDI-TOF-MS serum protein fingerprinting allows identification of differentially expressed proteins in colorectal cancer to develop models for diagnosis and prognosis evaluation of the disease.
- Citation: Wang Z, Li XQ, Wang KZ, Deng MM, Xu L. Serum protein fingerprinting for diagnosis and prognosis evaluation of colorectal cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(35): 3745-3751
- URL: https://www.wjgnet.com/1009-3079/full/v18/i35/3745.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i35.3745
大肠癌的发病率和死亡率在欧美及中国均居前列. 目前大肠癌的诊断及治疗较30年前发生了很大的改进, 但其发病率和死亡率并未发生根本的变化, 主要原因在于缺乏高灵敏度、特异度的早期诊断方法. 另外大肠癌也有年轻化的趋势, 且症状隐匿, 病情发展快, 多数患者被诊断为大肠癌时已属中晚期. 目前还没有任何一种优良的大肠癌血清肿瘤标志物可用于临床作为检测的区分标准.
癌细胞在生长和代谢过程中, 必定会留下蛛丝马迹, 只是限于一般检测手段的灵敏度低而不易发现. 高通量和高灵敏度的表面增强激光解析电离飞行时间质谱技术(surface-enhanced laser desorption ionization time of flight mass spectrometry, SELDI-TOF-MS)技术的应用, 是蛋白质的表达水平、翻译后的修饰、蛋白与蛋白的相互作用的重要工具, 由此能够获得蛋白质水平上的关于疾病发生、发展、细胞代谢等过程的整体而全面的认识.
大肠癌细胞侵袭和转移的分子生物学机制的研究是近几年的研究热点, 已发现一些癌基因、抑癌基因在大肠癌的侵袭和转移中起着重要作用[1]. 我们应用蛋白质组学方法筛选与大肠癌复发或转移相关的蛋白质. 从而筛选特异性蛋白标志物, 以期对大肠癌患者个体化医疗提供帮助.
标本为2009-07/2010-04收集的泸州医学院附属医院的门诊及住院患者共251例血清标本. 分为大肠癌组、预后不良组、预后良好组、肠道良性疾病组和健康人组. 大肠癌组45例: 大肠癌患者均经结直肠镜病理检查确诊全部为初发大肠腺癌45例, 无周围淋巴结转移, 其中结肠癌10例, 直肠癌35例, 男27例, 女18例, 年龄33-76岁; 预后不良组13例: 术后1年后有复发或转移, 男9例, 女4例, 年龄26-81岁; 预后良好组14例: 术后1年内无复发和无转移组, 男10例, 女4例, 年龄41-71岁; 肠道良性疾病组24例(其中结直肠息肉15例、慢性炎症8例、溃疡性结肠炎1例), 男10例, 女14例, 年龄21-65岁; 正常人组155 例, 男80例, 女75例, 年龄35-65岁. 其中各组在性别比例和年龄上无统计学差异.
1.2.1 样品的收集: 各组血清标本均为清晨空腹采血5 mL后立即放入4℃冰箱静置2 h , 4 ℃ 、1 000 r/min离心30 min, 吸取血清分装成50 μL/管, 置于-80 ℃冰箱保存.
1.2.2 样本的准备: 将保存于-80 ℃的样品取出常温下融解, 10 000 r/min离心2 min, 后将分离的血清标本与10 μL PBS Buffer(×10)按一定比例混合, 混匀. 吸取2倍体积的半饱和葡萄球菌蛋白A(staphylococcal protein A, SPA)加入样品混合管, 充分混匀, 静置5-10 min. 吸取待测混合血清样品2 μL, 于活化的金芯片(gold ship, AU)上点样, 待检.
1.2.3 芯片的预处理: 50 μL丙酮, 在层析柜中600 r/min, 5 min. 50 μL盐酸, 在层析柜中600 r/min, 5 min. 50 μL盐酸甲醇液, 在层析柜中600 r/min, 5 min. 50 μL甲醇液, 在层析柜中600 r/min, 5 min. 拍干, 下架, 空气中晾干待用.
1.2.4 芯片上样: 将处理好的样品2 μL点样于芯池中央. 干后用半饱和的SPA 1 μL, 5 min后再点1 μL.
1.2.5 芯片检测: AU蛋白芯片, 基质峰Average Intensity为60左右的仪器参数: 相对分子质量范围: 1 000-100 000; 优化范围: 2 000-20 000; 检测激光强度为175; 检测敏感性为9; 数据收集方法: Seldi Quantitation. 仪器采用Ciphergen公司提供的all-in-one蛋白质标准分子进行校正, 设定仪器参数, 系统的质量偏差≤0.1%.
1.2.6 差异蛋白质的筛选: 利用SELDI-TOF-MS技术筛选大肠癌患者组、肠道良性疾病组和正常人组共3组224例的差异蛋白, 建立人工神经网络(artificial neural network, ANN)大肠癌的诊断模型; 再利用SELDI-TOF-MS技术筛选术后预后良好组和术后预后不良组共2组27例的差异蛋白, 建立ANN大肠癌的预后模型.
1.2.7 ANN诊断模型和预后模型的建立: (1)ANN诊断模型: 用大肠癌患者组、肠道良性疾病组和正常人组共3组224例血清标本建立大肠癌的ANN诊断模型. 采用ANN技术, 将其输入层、隐含层1、输出层的节点分别设置7、2、1. 设定训练集大肠癌组的目标输出值为1, 其他2组的目标输出值为0, 训练次数为1 000, 学习率为0.01, 采用动量反向传播(back-propagation)算法, 建立并存储ANN诊断模型. (2)预后模型: 用大肠癌术后预后良好组和术后预后不良组共2组27例血清标本建立大肠癌的ANN预后模型. 采用ANN技术, 将其输入层、隐含层1、隐含层2、输出层的节点分别设置5、3、3、1. 设定训练集大肠癌术后复发或转移组的目标输出值为1, 术后预后良好组的目标输出值为 0, 训练次数为1 000, 学习率为 0.01, 采用动量反向传播算法, 建立并存储ANN预后模型.
1.2.8 诊断模型和预后模型的回验: (1)诊断模型: 利用双盲的方法, 将验证集中大肠癌组和其他2组血清蛋白标志物质谱数据输入模型进行模拟仿真计算, 设定cut-off值为0.5. 当输出值在0.5(含0.5)-1时, 归为大肠癌; 当输出值在0-0.5时, 归为非大肠癌. 通过模拟计算的结果, 对实验诊断模型的诊断效能进行评价. (2)预后模型: 将验证集中大肠癌术后预后良好组和术后预后不良组数据输入模型进行回验, 设定cut-off值为0.45. 当输出值在0.45(含0.45)-1时, 归为大肠癌术后复发或转移; 当输出值在0-0.45时, 归为术后预后良好. 通过模拟计算的结果, 对实验预后模型的符合率进行评价.
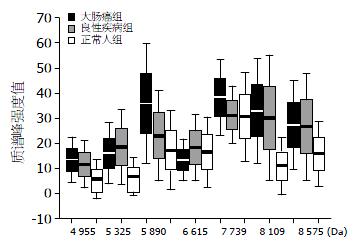
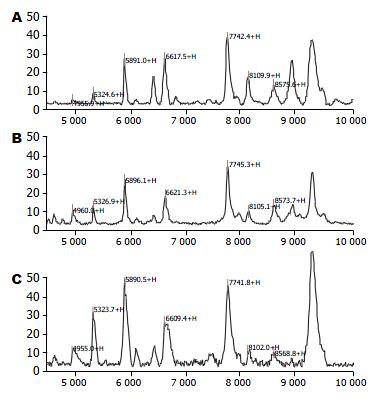
大肠癌的诊断模型中, 从大肠癌患者组、肠道良性疾病组和正常人组共3组224个样品的血清蛋白质谱指纹图中筛选出差异蛋白质316个(P<0.05), 从中筛选出7个表达有明显差异的标志蛋白质(P<0.01), 其相对分子质量为4 955、5 325、5 890、6 615、7 739、8 109、8 575 Da, 其质谱值见表1, 强度值变化趋势见图1, 典型样本蛋白质谱指纹图见图2.
| 分组 | n | 4 955 Da | 5 325 Da | 5 890 Da | 6 615 Da | 7 739 Da | 8 109 Da | 8 575 Da |
| 大肠癌组 | 45 | 13.59±8.98 | 16.14±12.08 | 35.92±23.89 | 13.42±8.17 | 38.30±14.96 | 33.00±20.73 | 27.44±17.58 |
| 良性疾病组 | 24 | 11.69±9.36 | 18.60±14.73 | 23.12±17.87 | 18.37±13.25 | 31.36±11.34 | 30.23±24.85 | 26.66±21.17 |
| 正常人组 | 155 | 5.81±7.96 | 6.87±7.65 | 17.32±15.76 | 16.56±13.97 | 30.71±17.50 | 11.05±11.30 | 15.83±12.95 |
| 组间F检验 | P<0.001 | P<0.001 | P<0.001 | P<0.01 | P<0.01 | P<0.001 | P<0.001 |
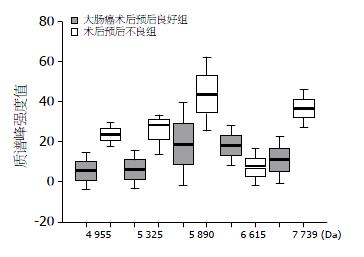
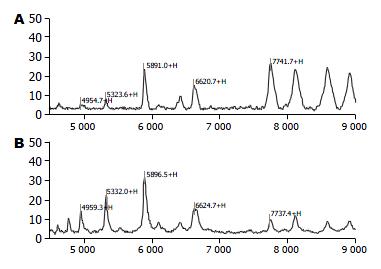
从大肠癌术后预后良好组和预后不良组共2组27个样品的血清蛋白指纹图谱, 得到差异蛋白质23个(P<0.05), 从中筛选出5个表达有明显差异的标志蛋白质(P<0.01), 其相对分子质量为4 955、5 325、5 890、6 615、7 739 Da, 其质谱峰强度值见表2, 强度值变化趋势见图3, 典型样本蛋白质谱指纹图见图4.
| 分组 | n | 4 955 Da | 5 325 Da | 5 890 Da | 6 615 Da | 7 739 Da |
| 大肠癌术后预后良好组 | 14 | 5.37±9.31 | 6.24±9.63 | 18.90±20.59 | 18.13±10.11 | 10.88±11.56 |
| 大肠癌术后预后不良组 | 13 | 23.57±5.93 | 28.48±14.76 | 43.71±18.51 | 7.32±8.94 | 36.70±9.27 |
| 组间F检验 | P<0.01 | P<0.01 | P<0.01 | P<0.01 | P<0.01 |
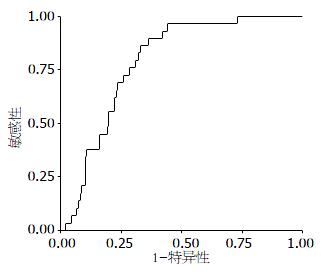
用大肠癌患者术前组45 例(目标输出值为1), 肠道良性疾病组24例和正常人组155例(目标输出值为0)共224例, 将7个差异蛋白质质谱峰强度作为7个输入节点, 建立大肠癌的ANN诊断模型. 其ROC曲线和数据集(表3, 图5), 该模型对大肠癌的诊断灵敏度和特异度分别为82.22%和80.45%, 阴性预测值94.74%, 阳性预测值51.39%, 准确度为80.80%. 大肠癌的ANN诊断模型, 其ROC曲线下面积为0.786.
| ANN仿真计算值 | 灵敏度 | 特异度 | 准确度 | 阴性预测值 | 阳性预测值 |
| 0.1 | 100.00 | 10.06 | 28.13 | 100.00 | 21.84 |
| 0.2 | 100.00 | 19.55 | 35.71 | 100.00 | 23.81 |
| 0.3 | 97.78 | 41.90 | 53.31 | 98.68 | 29.73 |
| 0.4 | 95.56 | 45.25 | 55.36 | 97.59 | 30.50 |
| 0.5 | 82.22 | 80.45 | 80.80 | 94.74 | 51.39 |
| 0.6 | 53.33 | 91.62 | 83.93 | 88.65 | 61.54 |
用筛选出的大肠癌患者术后无复发和无转移组与术后复发或转移组的5个差异蛋白质组建立大肠癌术后预后模型. 通过回验, 该模型的检验符合率为62.96%.
通过SELDI-TOF-MS技术得到蛋白质指纹图谱, 再搜索数据库, 鉴定出相应的蛋白质, 以达到进一步研究蛋白质的组成、结构和功能的目的, 最终阐明该蛋白质在疾病的发生, 发展中的作用机制, 以期通过阻断致癌机制或加强抑癌机制而达到基因预防和基因治疗的目的. Shiwa等[2]利用疏水性(H4)芯片、SELDI-TOF-MS和数据库搜索, 鉴定与相对分子质量12 000 Da对应的蛋白质是前胸腺素-α(prothymosin-α). 本研究中筛选出的7个差异蛋白为4 955、5 325、5 890、6 615、7 739、8 109、8 575 Da, 经Swiss-Prot蛋白数据库检索, 其中5 325和5 890 Da分别为脱羧酶蛋白和UPF蛋白, 可能是加强了致癌基因的作用, 而使在大肠癌术后复发或转移时升高为高表达, 其详细的作用机制尚需进一步的深入研究. 6 615 Da为复制时的DNA解螺旋酶. 生物基因信息的表达是通过基因DNA的翻译, 即蛋白质的生物合成而完成, 蛋白质结构和功能直接影响着生命的生理或病理条件下的变化过程. 在机体发生肿瘤时, 癌基因大量活化, 肿瘤蛋白大量通过翻译而生物合成从而使机体产生癌症病理变化. DNA解螺旋酶在癌基因大量活化的复制、转录和翻译的过程中起促癌作用. 差异蛋白6 615 Da在大肠癌组和大肠癌术后复发或转移组中是低表达, 其作用机制可能是DNA解螺旋酶活性下降, 癌基因的活化和肿瘤蛋白的产生可能受到了抑制, 这样间接加强了抑癌因素的作用, 但其发生的精确机制尚需进一步的深入研究.
筛选出的7个差异蛋白中4 955、5 890、7 739、8 109和8 575 Da在正常人组、肠道良性疾病组和大肠癌组中是一个逐渐增高的趋势, 在大肠癌组中的表达均数是最高的. 而差异蛋白6 615 Da在大肠癌组中为低表达, 而在肠道良性疾病组和正常人组为高表达, 在大肠癌术后无复发和无转移组中为高表达, 而在复发或转移时降低为低表达.
大肠癌是消化系常见的恶性肿瘤之一, 5年生存率随肿瘤分期的增高而显著降低[3-5]. 因此早期诊断、及时治疗是提高结直肠癌预后的主要因素. 目前大肠癌的诊断主要依赖于对高危人群进行体格检查、大便潜血试验、X线、CT、纤维结肠镜, 但是这些方法对大肠癌的诊断, 尤其是对Ⅰ期的诊断较为困难. 发现对大肠癌不同分期和亚型有意义的标志物对结直肠癌的预防、诊断及治疗有重要意义[6,7].
肿瘤是一个多因素多阶段的慢性疾病, 在疾病发生发展的过程中, 一些肿瘤细胞的蛋白质或多肽可以被分泌到循环系统中, 血液的蛋白质组学可发生相应变化, 反映出机体的疾病状态, 从而使得应用血清进行大肠癌蛋白质组学研究、发现患者的血清特异标志物成为一种可能. 而血清标志物的检测具有微创、患者易于接受等优点.
大肠癌的发生是多因素、多步骤共同作用的结果. 涉及多个基因事件包括癌基因激活、抑癌基因失活和这些基因相关的表达水平变化、离子通道改变等, 从而导致细胞的失控性生长, 侵袭、损害正常组织. 尽管目前已经发现很多与大癌相关的基因[8-10], 但大肠癌发生的精确机制仍然未知. 控制细胞增殖和分化的机制似乎受多种因素影响, 如细胞表面黏附蛋白, 趋化因子, 细胞外基质蛋白, 生长因子及其受体等. 蛋白质作为发挥基因功能的载体, 在疾病的发生、发展中有更直接的意义. 故近年来, 蛋白组学技术在大肠癌的发生、发展研究中显示出越来越重要的作用.
由美国Ciphergen Biosystems公司发明的SELDI-TOF-MS技术是蛋白质芯片和质谱技术的结合, 主要由蛋白质芯片、芯片阅读器和生物信息学3大部分组成, 其表面为可结合蛋白质的固体点表面, 检测的灵敏度高, 可把传统方法检测不到的蛋白和多肽检测出来, 可直接分析患者血清、淋巴液、脑脊液、尿液、细胞分泌液等样品而无需复杂的样品制备, 继Petricoinetal等[11]利用SELDI-TOF-MS技术分析了116例血清标本(其中卵巢癌50例, 正常对照66例)得出可以完全检出卵巢癌的血清蛋白质分类树模型之后, SELDI-TOF-MS广泛用于卵巢癌、前列腺癌、肝癌、食管癌、结直肠癌等多种肿瘤的研究, 已成为肿瘤蛋白质组学主要技术平台之一. 在大肠癌的应用中, Roboz等[12]采用SELDI-TOF-MS, 选择H4芯片分析了大肠癌患者与正常对照之间的血清蛋白图谱之间的差异, 其中大肠癌患者高表达8 942 Da蛋白, 而9 300 Da的蛋白呈低表达, 正常对照组上述两个蛋白的表达情况与患者组正好相反. Jungblut等[13]对15例结直肠癌和13例正常结肠上皮标本进行2DE分析, 发现在相对分子质量为13 000和PI值为56处的蛋白质仅出现在癌组织蛋白质图谱上, 该低相对分子质量蛋白亦表达于溃疡性肠炎和息肉性腺瘤.
与这些有关大肠癌蛋白标志物研究相比较, 我们选择了IMAC-Cu2+芯片和SELDI-TOF-MS技术共分析了251例血清标本, 检测了大肠癌术前组45例、术后无复发和无转移组14例、术后复发或转移组13例、肠道良性疾病组24例和健康人群组155例的蛋白质谱图, 利用大肠癌术前组、肠道良性疾病组和健康人群组建立的大肠癌诊断模型筛选出7个表达有明显差异的标志蛋白质, 其相对分子质量为4 955、5 325、5 890、6 615、7 739、8 109、8 575 Da, 差异具有统计学意义(P<0.01). 利用大肠癌术后无复发和无转移组和术后复发或转移组建立的大肠癌复发或转移模型筛选出5个表达有明显差异的标志蛋白质, 其相对分子质量为4 955、5 325、5 890、6 615、7 739 Da, 差异具有统计学意义(P<0.01). 分别建立了大肠癌的诊断和预后的分类树模型, 利用大肠癌的诊断模型进行盲法预测, 得到该模型对大肠癌的诊断灵敏度和特异度分别为82.22%和80.45%, 阴性预测值94.74%, 阳性预测值51.39%, 准确度为80.80%. 利用大肠癌的预后模型进行盲法预测, 通过回验, 该模型的检验符合率为62.96%.
相对于CEA和CA242等诊断大肠癌的血清标志物, 目前常用的血清标志物如CEA、CA19-9、CA242、CA72-4以及组织多肽抗原等对大肠癌诊断的灵敏度、特异度都较低[14], 在CEA的研究中, 总体的敏感性为43%-69%; 在CA19-9的大多数研究中, 总体的敏感性则为18%-65%; CA-242的敏感性和特异性都只有40%左右, 而在组织多肽抗原的研究中, 其敏感性则在50%以下[15], 相比较SELDI-TOF-MS分析方法具有较高的诊断率和较好的诊断意义.
本研究不仅选取了健康人群和具有一定癌变趋势的良性肠道病变进行研究, 还选取了大肠癌术前的患者、术后无复发和无转移的患者和术后复发或转移的患者, 旨在了解肠道良性病变向大肠癌演变过程中的变化, 在大肠癌和肠道良性疾病之间存在一些蛋白质分子的表达有显著性差异, 这些蛋白可能是良性病变向大肠癌发展过程中呈连续变化趋势的蛋白, 并了解大肠癌术前、术后、复发或转移时的一些蛋白分子的表达的差异, 以期达到对大肠癌的早期诊断和远期预后判断的目的.
我们注意到在7个差异蛋白中6 615 Da的差异蛋白, 查阅结直肠癌的蛋白质库中显示为复制时的DNA解螺旋酶. 差异蛋白6 615 Da在大肠癌组和大肠癌术后复发或转移组中是低表达, 其作用机制可能是DNA解螺旋酶活性下降, 癌基因的活化、表达和肿瘤蛋白的产生可能受到了抑制, 这样间接加强了抑癌因素的作用, 其发生的精确机制尚需进一步深入研究. 此途径的研究明确, 将对研究阐述癌症的发生机制和癌症的基因治疗方面有所帮助.
今后我们将应用蛋白质组学技术鉴定所发现的差异蛋白及分类树模型中M/Z峰对应蛋白, 以便进一步提高诊断大肠癌的灵敏度和特异度, 并明确这些蛋白的来源, 如宿主器官、肿瘤或代谢产物, 以研究肿瘤蛋白在术前、术后和复发或转移时的变化情况, 用于初步判断患者的远期预后情况.
总之, 我们应用SELDI-TOF-MS和IMAC-Cu2+芯片发现了可以快速识别经病理确诊为大肠癌患者的血清蛋白标志物. 结果表明应用SELDI-TOF-MS和IMAC-Cu2+芯片和有效的分析软件可以得到能准确鉴别大肠癌术前、术后和复发或转移的血清分类树模型, 具有较高的灵敏度、特异度, 对提高大肠癌的早期诊断水平、改善大肠癌预后具有重要的潜在意义. 同时对其进行鉴定也有助于了解大肠癌发生、进展及转移的机制, 为深入进行肿瘤的蛋白质组研究提供新的有效方法.
癌细胞在生长和代谢过程中, 必定会留下蛛丝马迹, 只是限于一般检测手段的灵敏度低而不易被发现. 高通量和高灵敏度的SELDI-TOF-MS技术的应用, 是研究蛋白质的表达水平、翻译后的修饰、蛋白与蛋白的相互作用的重要工具, 由此能够获得蛋白质水平上的关于疾病发生、发展、细胞代谢等过程的整体而全面的认识.
曹志成, 英国生物医学科学研究所院士, 香港伊利沙伯医院临床肿瘤科
大肠癌细胞侵袭和转移的分子生物学机制的研究是近几年的研究热点, 已发现一些癌基因、抑癌基因在大肠癌的侵袭和转移中起着重要作用.
Roboz等采用SELDI-TOF-MS, 选择H4芯片分析了大肠癌患者与正常对照之间的血清蛋白图谱之间的差异, 其中大肠癌患者高表达8 942 Da蛋白, 而9 300 Da的蛋白呈低表达, 上述两种蛋白在正常对照组中的表达情况与患者组正好相反.
本项研究采用Au芯片分离血清蛋白并采集质谱指纹图, 发现了可以快速识别经病理确诊为大肠癌患者的血清蛋白标志物. 通过数据挖掘可以得到能准确鉴别大肠癌术前、术后和复发或转移的血清蛋白分类树模型, 能够提高大肠癌的早期诊断水平, 对大肠癌预后判别具有重要意义.
帮助诊断和筛查大肠癌患者, 有助于了解大肠癌发生、进展及转移的机制, 为深入进行癌细胞浸润的蛋白质组和基因组学研究提供新的有效方法.
本文可读性较好, 对临床医师诊断和筛查大肠癌患者具有一定的指导意义.
编辑: 李薇 电编:何基才
| 2. | Shiwa M, Nishimura Y, Wakatabe R, Fukawa A, Arikuni H, Ota H, Kato Y, Yamori T. Rapid discovery and identification of a tissue-specific tumor biomarker from 39 human cancer cell lines using the SELDI ProteinChip platform. Biochem Biophys Res Commun. 2003;309:18-25. [PubMed] [DOI] |
| 3. | Jemal A, Siegel R, Ward E, Hao Y, Xu J, Murray T, Thun MJ. Cancer statistics, 2008. CA Cancer J Clin. 2008;58:71-96. [PubMed] [DOI] |
| 5. | O'Connell JB, Maggard MA, Liu JH, Etzioni DA, Ko CY. Are survival rates different for young and older patients with rectal cancer? Dis Colon Rectum. 2004;47:2064-2069. [PubMed] [DOI] |
| 6. | Crawford NP, Colliver DW, Galandiuk S. Tumor markers and colorectal cancer: utility in management. J Surg Oncol. 2003;84:239-248. [PubMed] [DOI] |
| 7. | Duffy MJ, van Dalen A, Haglund C, Hansson L, Klapdor R, Lamerz R, Nilsson O, Sturgeon C, Topolcan O. Clinical utility of biochemical markers in colorectal cancer: European Group on Tumour Markers (EGTM) guidelines. Eur J Cancer. 2003;39:718-727. [PubMed] [DOI] |
| 8. | Rajagopalan H, Nowak MA, Vogelstein B, Lengauer C. The significance of unstable chromosomes in colorectal cancer. Nat Rev Cancer. 2003;3:695-701. [PubMed] [DOI] |
| 9. | Cho WC, Yip TT, Yip C, Yip V, Thulasiraman V, Ngan RK, Yip TT, Lau WH, Au JS, Law SC. Identification of serum amyloid a protein as a potentially useful biomarker to monitor relapse of nasopharyngeal cancer by serum proteomic profiling. Clin Cancer Res. 2004;10:43-52. [PubMed] [DOI] |
| 10. | Hundt S, Haug U, Brenner H. Blood markers for early detection of colorectal cancer: a systematic review. Cancer Epidemiol Biomarkers Prev. 2007;16:1935-1953. [PubMed] [DOI] |
| 11. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] [DOI] |
| 12. | Roboz J, Ma LH, Sung M. Protein Profiles of Serum in Colon Cancer by SELDI-TOF Mass Spectrometry. Proteomics, Poster Session A AACR. 2002;190. |
| 13. | Jungblut PR, Zimny-Arndt U, Zeindl-Eberhart E, Stulik J, Koupilova K, Pleissner KP, Otto A, Müller EC, Sokolowska-Köhler W, Grabher G. Proteomics in human disease: cancer, heart and infectious diseases. Electrophoresis. 1999;20:2100-2110. [PubMed] [DOI] |
| 14. | Bellacosa A. Genetic hits and mutation rate in colorectal tumorigenesis: versatility of Knudson's theory and implications for cancer prevention. Genes Chromosomes Cancer. 2003;38:382-388. [PubMed] [DOI] |