修回日期: 2010-10-26
接受日期: 2010-11-02
在线出版日期: 2010-11-28
目的: 探讨甲基化寡核苷酸诱导灭活SW480结肠癌细胞CHFR基因甲基化和表达情况对生物学行为的影响.
方法: 将与CHFR基因启动子序列互补的甲基化寡核苷酸转染进SW480结肠癌细胞内, 分别应用MSP和RT-PCR检测转染前后CHFR基因启动子甲基化状态和mRNA表达. 应用MTT法检测转染前后细胞增殖状态.
结果: 正常未转染寡核苷酸组和对照组SW480结肠癌细胞CHFR基因启动子表现未甲基化状态并表达CHFR基因mRNA. 转染互补甲基化寡核苷酸后, CHFR基因启动子被诱导成甲基化状态, mRNA表达受到抑制而缺失, 细胞增殖速度明显高于正常未转染寡核苷酸组和对照组; 正常未转染寡核苷酸组和对照组细胞增殖速度无明显差异.
结论: 互补甲基化寡核苷酸可诱导SW480结肠癌细胞CHFR基因启动子甲基化状态并抑制其mRNA表达, 促进细胞增殖.
引文著录: 邱庆安, 吕云福, 陈一明, 林友刚, 伍海鹰, 刘宁. 甲基化寡核苷酸对SW480结肠癌细胞CHFR基因的诱导灭活作用及其临床意义. 世界华人消化杂志 2010; 18(33): 3565-3568
Revised: October 26, 2010
Accepted: November 2, 2010
Published online: November 28, 2010
AIM: To investigate whether transfection of methylated oligonucleotides that are complementary to the CHFR (checkpoint with forkhead and ring finger domains) gene promoter inactivates the CHFR gene in human colon cancer cell line SW480 and to analyze the effect of CHFR inactivation on the biological behavior of SW480 cells.
METHODS: After methylated oligonucleotides that are complementary to the CHFR gene promoter were transfected into SW480 cells, MSP and RT-PCR were employed to detect the promoter methylation status and mRNA expression of the CHFR gene, respectively. MTT assay was used to detect the proliferation of SW480 cells before and after transfection.
RESULTS: In control SW480 cells, the promoter region of the CHFR gene was unmethylated and CHFR mRNA expression could be detected. After transfection of methylated oligonucleotides, CHFR gene promoter methylation was induced and CHFR mRNA expression was suppressed. The proliferation of SW480 cells transfected with methylated oligonucleotides was significantly higher than that of control cells.
CONCLUSION: Methylated oligonucleotides that are complementary to the CHFR gene promoter can induce CHFR gene promoter methylation, suppress CHFR mRNA expression, and promote cell proliferation in human colon cancer cell line SW480.
- Citation: Qiu QA, Lv YF, Chen YM, Lin YG, Wu HY, Liu N. Transfection of methylated oligonucleotides inactivates the CHFR gene in human colon cancer cell line SW480. Shijie Huaren Xiaohua Zazhi 2010; 18(33): 3565-3568
- URL: https://www.wjgnet.com/1009-3079/full/v18/i33/3565.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i33.3565
含叉头和环指域检控点基因(checkpoint with forkhead and ring finger domains, CHFR)位于染色体12q24.3, 其编码产物为664个氨基酸的蛋白质, 含有3个结构域: N-端有叉头相关区(FHA)和环指区(RF), 羧基端有半胱氨酸富集区(CR)[1,2]. CHFR基因是一个新发现的有丝分裂前期检查点基因, 具有E3泛素蛋白连接酶作用. 细胞微管靶向抑制剂诱发有丝分裂应激时, CHFR通过调控Cdc2/Cyclin B底物, 延迟染色体凝集、阻止细胞由有丝分裂前期进入中前期, 增强细胞对应激的生存能力[3,4]. CHFR基因在正常人体组织中普遍表达, 文献报道其基因启动子在许多肿瘤组织中呈现高甲基化状态, 基因表达完全缺失, 目前认为CHFR基因是一个重要的肿瘤抑制基因, 在肿瘤发病机制中扮演重要角色[5-8]. 目前研究表明, CHFR基因启动子在结肠癌组织和某些细胞系中处于甲基化灭活状态[9,10], 而CHFR基因启动子甲基化导致结肠癌发生的具体机制仍然不清楚. 本研究应用互补甲基化寡核苷酸转染SW480结肠癌细胞, 检测转染前后CHFR基因启动子甲基化状态和mRNA表达改变及其对SW480结肠癌细胞生物学行为的影响, 进一步探讨CHFR基因在结肠癌发病机制中的作用.
结肠癌细胞系SW480购于美国典型培养物收藏中心(ATCC); 胎牛血清、RPMI 1640购自Hyclone公司; DNA、RNA提取试剂购自天根生化科技有限公司; 甲基化试剂购自ZYMO公司; Effectene转染试剂购买自Qiagen公司; PCR和RT-PCR试剂盒购自Chemicon公司; 引物和甲基化寡核苷酸由上海生工公司合成; 测序由TaKaRa公司完成; 由DBTSS数据库检索人类CHFR基因启动子序列并根据文献设计甲基化寡核苷酸序列[11-13], 甲基化寡核苷酸(methylated oligonucleotide, MON)序列: 5'-GmCGCmCGCmCGCCAGCCAATmCGmCGGGGmCGC-3'(MON组); 对照寡核苷酸(control oligonucleotide, CON)序列: 5'-GCGCCGCCGCCAGCCAATCGCGGGGCGC-3'(CON组), MON序列针对转录起始点上游50 bp序列, 包含6个CG位点.
1.2.1 Effectene转染SW480结肠癌细胞: 按照Effectene说明书, 准备24孔板SW480结肠癌细胞, 细胞汇合度为40%左右. 寡核苷酸0.5 μg、Enhancer和Effectene试剂按照1:10比例配制转染混合物. 转染混合物与细胞在培养箱共培养24 h后, 洗涤去掉转染混合物并更换新鲜培养基.
1.2.2 甲基化特异性PCR检测CHFR基因启动子甲基化状态: 提取SW480结肠癌细胞DNA, 按照甲基化试剂盒说明书将DNA上未甲基化C转化成U, 进行PCR扩增. 采用MethPrimer[14]设计引物, 甲基化引物: 上游引物5'-TAGGATTAAAGATGGTCGAGC-3'和下游引物为5'-TTAACTAACGACGACGCTCAC-3', 非甲基化引物: 上游引物5'-GGTTAGGATTAAAGATGGTTGAGT-3'和下游引物5'-ATTAACTAACAACAACACTCACCA-3'. PCR反应体系为25 μL, 反应参数为: 95 ℃预变性10 min, 95 ℃变性30 s, 62 ℃退火30 s, 72 ℃延伸30 s, 共35个循环, 最后72 ℃延伸10 min, PCR产物进行琼脂糖凝胶电泳分离检测.
1.2.3 RT-PCR检测CHFR基因mRNA表达: 上游引物: 5'-TAAAGGAAGTGGTCCCTCTGTG-3', 下游引物: 5'-GGTTTGGGCATTTCTACGC-3', 产物大小为205 bp, 以β-actin为内参照物. PCR反应条件为: 95 ℃预变性5 min, 95 ℃变性1 min, 58 ℃退火45 s, 72 ℃延伸60 s, 35个循环, 终末72 ℃延伸10 min, 将PCR产物进行琼脂糖凝胶电泳分离检测结果.
1.2.4 MTT法检测SW480细胞增殖: 正常未处理组、MON转染后甲基化特异性PCR(methylation specific PCR, MSP)检测显示CHFR基因启动子甲基化和CON转染后MSP检测显示CHFR基因未甲基化的SW480结肠癌细胞继续培养, 按每孔1 000个细胞接种于96孔板, 每孔200 μL, 空白对照组不含SW480结肠癌细胞. 每天取出1板, 每孔加入5 g/L MTT溶液20 μL, 37 ℃孵育4 h后弃去, 加入DMSO 50 μL, 振荡器振荡15 min充分溶解结晶, 酶标检测仪上测定波长为490 nm下的吸光值(A值), 以吸光值为纵坐标, 时间(d)为横坐标, 绘制CON组、MON组和正常未处理组SW480结肠癌细胞增殖曲线.
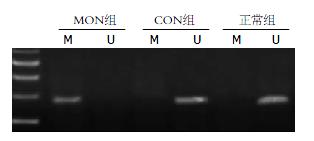
在正常未转染寡核苷酸的SW480结肠癌细胞中, CHFR基因启动子MSP结果显示其处于未甲基化状态, 该结果与Bertholon等[10]的实验结果一致. CON转染SW480结肠癌细胞后, CHFR基因启动子未受到影响, 呈现与正常未处理细胞一致的未甲基化状态, 而MON转染后, CHFR基因启动子M条带阳性, 呈现甲基化状态见图1.
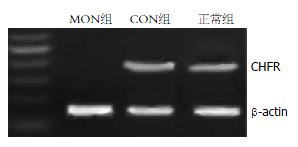
在正常未转染寡核苷酸的SW480结肠癌细胞中, CHFR基因mRNA表达正常, 转染CON后, 表达未受影响; 在转染MON后, CHFR基因mRNA表达完全受到抑制, 基因表达缺失(图2).
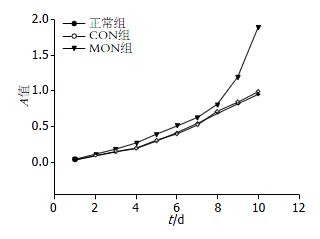
CON转染SW480结肠癌细胞后, 与正常未转染寡核苷酸组细胞增殖速度无差异, SW480结肠癌细胞增殖不受CON影响, 表现指数增殖现象; MON转染SW480结肠癌细胞后, 细胞增殖速度明显高于正常未处理组和CON组(图3).
研究表明在真核生物DNA中约80% CpG存在甲基化[15], 甲基化主要发生在基因启动子的核心序列和转录起始点, 甚至延伸至第一外显子, 这些区域构成CpG岛[16,17]. DNA甲基化可引起基因组中相应区域染色质结构变化, 使染色质高度螺旋化, 凝缩成团, 甲基化自身或结合DNA甲基结合蛋白(methyl-binding protein, MBP)阻止转录相关因子结合DNA, 从而抑制mRNA转录和翻译, 是最重要的非DNA序列突变方式调控真核生物基因表达[15,18,19], 在肿瘤发生[20,21]、X染色体灭活等机制方面扮演重要角色.
人工设计合成的互补甲基化寡核苷酸能与基因启动子区域的其中一条链互补结合形成半甲基化DNA链, 产生复制叉样结构, 为DNA甲基化转移酶1(DNA methyltransferase 1, DNTM1)提供优先底物, DNMT1催化导致其迅速甲基化, 甲基化寡核苷酸与结合位点分离后, 甲基化第一链与未甲基化互补链互补形成第2个半甲基化复制叉结构, 在DNTM1催化下, 最终两条链均发生甲基化, 导致启动子无法和转录因子结合, 基因转录灭活而沉默[22]. 本研究基于此机制, 人工合成互补甲基化寡核苷酸诱导SW480结肠癌细胞系CHFR基因启动子甲基化灭活状态, 模拟CHFR基因启动子甲基化导致肿瘤发生过程, 为肿瘤基因靶向治疗提供新思路.
本研究结果表明, SW480结肠癌细胞系CHFR基因启动子表现未甲基化状态, 其mRNA表达正常, 这些证据表明结肠癌的发生可能是多因素和多基因共同决定的结果, 也可能因MSP自身技术缺陷导致无法检测到其余CG位点甲基化状态. CHFR作为有丝分裂前期检查点基因, 缺乏结肠癌相关的特异性, 导入互补外源性MON和CHFR基因启动子结合后, 在DNA甲基转移酶催化下, 成功诱导启动子产生甲基化状态, 导致转录因子不能与其结合而灭活基因表达, 产生基因沉默效应. 在SW480结肠癌细胞有丝分裂前期, 诱导灭活CHFR导致细胞失去周期调控, 产生恶性增殖现象, 说明MON诱导的有效性. 在未来的肿瘤治疗中, 我们可以人工合成互补甲基化灭活肿瘤相关基因, 靶向导入体内治疗肿瘤, 也可以利用去甲基化试剂使得抑癌基因从新表达而治疗恶性肿瘤.
CHFR基因是一个新发现的有丝分裂前期检查点基因, 具有E3泛素蛋白连接酶作用. 细胞微管靶向抑制剂诱发有丝分裂应激时, CHFR通过调控Cdc2/Cyclin B底物, 延迟染色体凝集、阻止细胞由有丝分裂前期进入中前期, 增强细胞对应激的生存能力.
蔡开琳, 副教授, 华中科技大学同济医学院附属协和医院普通外科
文献报道CHFR基因启动子在许多肿瘤组织中呈现高甲基化状态, 基因表达完全缺失, 目前认为CHFR基因是一个重要的肿瘤抑制基因, 在肿瘤发病机制中扮演重要角色.
本实验证实了CHFR基因启动子甲基化与结肠癌之间的关系, 在未来的肿瘤靶向治疗中, 可以将体外合成的与肿瘤基因启动子互补的甲基化寡核苷酸导入人体内, 灭活肿瘤基因, 治疗各种良恶性肿瘤.
本文技术和理论新颖, 为肿瘤基因靶向治疗提供新思路.
编辑: 李薇 电编:何基才
| 1. | Scolnick DM, Halazonetis TD. Chfr defines a mitotic stress checkpoint that delays entry into metaphase. Nature. 2000;406:430-435. [PubMed] [DOI] |
| 3. | Chaturvedi P, Sudakin V, Bobiak ML, Fisher PW, Mattern MR, Jablonski SA, Hurle MR, Zhu Y, Yen TJ, Zhou BB. Chfr regulates a mitotic stress pathway through its RING-finger domain with ubiquitin ligase activity. Cancer Res. 2002;62:1797-1801. [PubMed] |
| 4. | Kang D, Chen J, Wong J, Fang G. The checkpoint protein Chfr is a ligase that ubiquitinates Plk1 and inhibits Cdc2 at the G2 to M transition. J Cell Biol. 2002;156:249-259. [PubMed] [DOI] |
| 5. | Koga T, Takeshita M, Yano T, Maehara Y, Sueishi K. CHFR hypermethylation and EGFR mutation are mutually exclusive and exhibit contrastive clinical backgrounds and outcomes in non-small cell lung cancer. Int J Cancer. 2010; May 12. [Epub ahead of print]. [PubMed] |
| 6. | Hu SL, Huang DB, Sun YB, Wu L, Xu WP, Yin S, Chen J, Jiang XD, Shen G. Pathobiologic implications of methylation and expression status of Runx3 and CHFR genes in gastric cancer. Med Oncol. 2010; Mar 19. [Epub ahead of print]. [PubMed] |
| 7. | Cheng ZD, Hu SL, Sun YB, Xu WP, Shen G, Kong XY. Promoter methylation of CHFR gene in gastric carcinoma tissues detected using two methods. Chin J Cancer. 2010;29:163-166. [PubMed] |
| 8. | Gao Y, Lou G, Zhang GM, Sun XW, Ma YY, Yang YM, Liu G. CHFR promoter hypermethylation and reduced CHFR mRNA expression in ovarian cancer. Int J Biol Markers. 2009;24:83-89. [PubMed] |
| 9. | Brandes JC, van Engeland M, Wouters KA, Weijenberg MP, Herman JG. CHFR promoter hypermethylation in colon cancer correlates with the microsatellite instability phenotype. Carcinogenesis. 2005;26:1152-1156. [PubMed] [DOI] |
| 10. | Bertholon J, Wang Q, Falette N, Verny C, Auclair J, Chassot C, Navarro C, Saurin JC, Puisieux A. Chfr inactivation is not associated to chromosomal instability in colon cancers. Oncogene. 2003;22:8956-8960. [PubMed] [DOI] |
| 11. | Ishii T, Fujishiro M, Masuda M, Teramoto S, Matsuse T. A methylated oligonucleotide induced methylation of GSTP1 promoter and suppressed its expression in A549 lung adenocarcinoma cells. Cancer Lett. 2004;212:211-223. [PubMed] [DOI] |
| 12. | Yao X, Hu JF, Daniels M, Shiran H, Zhou X, Yan H, Lu H, Zeng Z, Wang Q, Li T. A methylated oligonucleotide inhibits IGF2 expression and enhances survival in a model of hepatocellular carcinoma. J Clin Invest. 2003;111:265-273. [PubMed] |
| 14. | Li LC, Dahiya R. MethPrimer: designing primers for methylation PCRs. Bioinformatics. 2002;18:1427-1431. [PubMed] [DOI] |
| 15. | Levenson VV. DNA methylation as a universal biomarker. Expert Rev Mol Diagn. 2010;10:481-488. [PubMed] [DOI] |
| 16. | Ushijima T, Asada K. Aberrant DNA methylation in contrast with mutations. Cancer Sci. 2010;101:300-305. [PubMed] [DOI] |
| 17. | Tost J. DNA methylation: an introduction to the biology and the disease-associated changes of a promising biomarker. Mol Biotechnol. 2010;44:71-81. [PubMed] [DOI] |
| 18. | Qureshi SA, Bashir MU, Yaqinuddin A. Utility of DNA methylation markers for diagnosing cancer. Int J Surg. 2010;8:194-198. [PubMed] [DOI] |
| 19. | Poke FS, Qadi A, Holloway AF. Reversing aberrant methylation patterns in cancer. Curr Med Chem. 2010;17:1246-1254. [PubMed] [DOI] |
| 20. | Teyssier C, Le Romancer M, Sentis S, Jalaguier S, Corbo L, Cavaillès V. Protein arginine methylation in estrogen signaling and estrogen-related cancers. Trends Endocrinol Metab. 2010;21:181-189. [PubMed] [DOI] |
| 21. | Shivapurkar N, Gazdar AF. DNA methylation based biomarkers in non-invasive cancer screening. Curr Mol Med. 2010;10:123-132. [PubMed] [DOI] |
| 22. | Szyf M, Detich N. Regulation of the DNA methylation machinery and its role in cellular transformation. Prog Nucleic Acid Res Mol Biol. 2001;69:47-79. [PubMed] [DOI] |