修回日期: 2010-09-21
接受日期: 2010-09-27
在线出版日期: 2010-10-28
目的: 研究人结直肠癌、腺瘤和癌旁正常组织中微小核糖核酸(microRNAs, miRNAs)的差异表达谱, 并初步探讨其临床意义.
方法: 选取2008-01/07苏州大学附属第一医院和泰州市人民医院结直肠癌、对应癌旁正常组织以及结直肠腺瘤标本, 提取组织总RNA, 采用illumina microRNA芯片技术检测3种不同类型组织中miRNAs的表达, 采用实时定量PCR技术对芯片检测结果进行验证. 将结直肠癌组织中异常表达的miRNAs与结直肠癌患者的临床病理资料进行分析.
结果: 3种不同类型结直肠组织中miRNAs表达有明显差异, 结直肠癌与癌旁正常组织相比, 有65个miRNAs表达异常(P<0.001), 其中35个上调, 30个下调, 而腺瘤与正常结直肠组织相比, 有55个miRNAs表达差异(P<0.001), 其中上调29个, 下调26个. 有25个miRNAs相对于正常组织同时在结直肠癌和腺瘤中异常表达, 其中高表达12个, 低表达有13个. 与腺瘤相比, 结直肠癌中有25个miRNAs表达异常(P<0.01), 其中13个上调, 12个下调. 进一步定量PCR验证结果显示与正常结直肠黏膜组织相比, 在癌组织中miR-552, miR-142-3p上调(2.97±2.72 vs 0.98±0.48; 3.64±3.41 vs 1.31±0.61, 均P<0.05), miR-139-3p和miR-133b下调(0.10±0.26 vs 0.82±0.70, 0.81±0.67 vs 1.71±1.29, 均P<0.05), 与芯片结果相吻合. miR-552和miR-139-3p与结直肠癌患者年龄、性别、组织学、肿瘤大小和浸润深度无关(P>0.05), 而miR-552与TNM分期、淋巴结和肝转移相关(P<0.05), miR-139-3p与TNM分期和肝转移相关(P<0.05), miR-142-3p除与组织学有关外, 与其他临床病理参数无关, miR-133b与年龄相关, 而与其他临床病理参数无关.
结论: miRNAs参与了结直肠的癌变过程, 特异的miRNAs的表达可能成为结直肠癌潜在的早期诊断和治疗新靶点.
引文著录: 陈卫昌, 林茂松, 黄俊星, 叶玉兰, 郜恒骏, 宋振云, 沈晓莹. 人结直肠癌微小核糖核酸的差异表达及其临床意义. 世界华人消化杂志 2010; 18(30): 3187-3194
Revised: September 21, 2010
Accepted: September 27, 2010
Published online: October 28, 2010
AIM: To investigate the expression profile of microRNAs (miRNAs) in human colorectal cancer and to analyze the clinical significance of aberrant miRNA expression.
METHODS: The expression of miRNAs in colorectal cancer specimens, matched normal mucosal specimens, and adenoma specimens was detected by miRNA microarray. The differential expression of some miRNAs was verified by real-time RT-PCR. The correlation of aberrant miRNA expression with clinical and biopathologic features in colorectal cancer, such as age, gender, tumor size, lymph node and distance metastasis, clinical stage as well as histological grade, was analyzed.
RESULTS: Microarray analysis revealed many miRNAs that could clearly differentiate colorectal cancer from adenoma and normal tissue. Thirty-five significantly up-regulated miRNAs and 30 down-regulated miRNAs were identified in carcinoma in comparison to normal tissue (P < 0.001), whereas 25 aberrantly expressed miRNAs were identified in cancer in comparison to adenoma (P < 0.01). In addition, 55 miRNAs could distinguish adenoma from normal colorectal mucosa (P < 0.001). Twelve up-regulated miRNAs and 13 down-regulated ones were simultaneously aberrantly expressed in cancer and adenoma. Quantitative RT-PCR analysis showed that miR-552 and miR-142-3p were significantly up-regulated (2.97 ± 2.72 vs 0.98 ± 0.48, P < 0.05; 3.64 ± 3.41 vs 1.31 ± 0.61, P < 0.05) and miR-139-3p and miR-133b were down-regulated in cancer tissue compared with normal mucosa (0.10 ± 0.26 vs 0.82 ± 0.70, P < 0.05; 0.81 ± 0.67 vs 1.71 ± 1.29, P < 0.05), which is consistent with the microarray results. Aberrant miR-552 expression was correlated with TNM stage and lymph node and distance metastasis (all P < 0.05), but not with age, gender, tumor size or histological grade. Aberrant miR-139-3P expression was correlated with TNM stage and distance metastasis, while differential expression of miR-142-3p was correlated with histological grade.
CONCLUSION: Differential miRNA expression could help distinguish malignant tissue from adenoma and normal mucosa, suggesting that aberrant miRNA expression might be involved in colorectal tumorigenesis. Differentially expressed miRNAs might be used as early diagnosis markers or therapeutic targets for human colorectal cancer.
- Citation: Chen WC, Lin MS, Huang JX, Ye YL, Gao HJ, Song ZY, Shen XY. Clinical significance of aberrant microRNAs expression in human colorectal cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(30): 3187-3194
- URL: https://www.wjgnet.com/1009-3079/full/v18/i30/3187.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i30.3187
近年来我国的结直肠癌发病率逐渐升高. 研究认为结直肠癌的发生是多种基因畸变的结果[1,2], 但是至今尚未发现具有真正早期诊断意义和确切疗效的靶点. 微小RNA(microRNAs, miRNAs)是近来在动植物中发现的一个大小约为19-25个核苷酸, 非蛋白质编码的小分子RNA家族[3,4]. 研究认为miRNAs可以通过干扰其靶基因mRNAs的翻译或者导致靶mRNAs的降解, 下调靶基因的表达[5,6], 从而参与体内许多重要的生命活动, 如细胞增殖、分化以及凋亡等, 因此认为miRNAs的表达失调可能与肿瘤发生密切相关[7-12].
本研究中我们拟先采用illumina microRNA芯片检测人结直肠癌、结直肠腺瘤和良性癌旁组织的miRNAs表达, 通过比较他们的表达谱, 寻找与结直肠癌发生相关的特异miRNAs, 从而为进一步寻找具有早期诊断意义的标志物和治疗靶点提供一定的理论依据, 然后我们将在结直肠癌中发现的一些异常表达的miRNAs与结直肠癌患者的临床资料进行比较分析, 初步探讨他们异常表达的临床意义.
所有标本来自于苏州大学附属第一医院和泰州市人民医院2008-01/07住院和/或门诊收治的结直肠癌和结肠腺瘤患者, 所有患者均签署知情同意书, 并获得苏州大学附属第一医院伦理委员会批准. 其中, 结直肠癌组织标本来自于手术切除标本, 而腺瘤标本则来自于内镜下摘除组织, 所有组织标本术后立即液氮保存. 所有癌症患者术前均未接受放、化疗. 在这些标本中, 结直肠癌25例, 腺瘤3例, 癌旁正常结直肠黏膜组织10例. 结直肠癌组织参照全国结直肠癌协作组结直肠癌诊治规范以及美国癌症协会/国际抗癌组织(American Joint Committee on Cancer/Union for International Cancer Control, AJCC/UICC). TNM分期标准行诊断、分级和分期. 结直肠癌患者中男6例, 女19例, 年龄43-83(中位年龄63)岁. 根据组织学分化程度分为: 高分化组1例, 中分化组19例, 低分化组5例(黏液腺癌及印戒细胞癌并入低分化组). 其中淋巴结转移阳性10例, 阴性15例, 远处转移(肝转移)阳性5例, 阴性20例. 根据临床TNM分期分为: Ⅰ期5例, Ⅱ期8例, Ⅲ期7例和Ⅳ期5例. 根据肿瘤大小分为: ≥5 cm 14例, <5 cm 11例, 根据肿瘤浸润深度: 肌层以下6例, 全层19例. 肿瘤组织均取自肿瘤中心非坏死部位, 癌旁正常组织取自距肿瘤≥5 cm的正常组织, 腺瘤组织来自于大肠镜下摘除标本, 所有组织均经病理证实. microRNA芯片(human microRNA assay pool, version 2, 12样本universal Beadchip, 含1 146个最新人类miRNAs, 包括覆盖>97% miRbase 12.0版本中收录最新人类miRNAs以及illumina公司通过测序法发现的最新符合miRNAs特征, 但是尚未命名的序列)、miRNAs标志杂交试剂盒购自illumina公司. miScript Reverse Transcription Kit购自Qiagen, SYBR Green Realtime PCR Master Mix购自TaKaRa.
1.2.1 样本RNA的提取与标记: 取液氮保存组织5 mg, 在液氮下研磨, 采用Invitrogen® TRIzol来提取组织中的总RNA, 然后用QIAGEN RNeasy Kit进一步纯化总RNA, 使用琼脂糖凝胶电泳对样品总RNA质量进行完整性检测分析. 使用Eppendorf Biophotometer系统对样品总RNA进行质量(定量)检测, Nano Drop分光光度仪测定吸光度A260/A280值. 取200 ng的总RNA, 按照Illumina® microRNA Expression Profiling Assay kit实验手册操作, 用Polyadenylation Single Reagent将其多聚腺苷酸化. 取多聚腺苷酸化的总RNA, 以带有通用引物扩增位点并用生物素标记的Oligo(dT)为引物进行逆转录合成cDNA一链并纯化, 将含miRNAs特异序列的通用引物退火至cDNA一链上, 延伸引物合成双链cDNA. 洗去多余的引物和错配的产物, 以荧光标记的通用引物进行PCR扩增.
1.2.2 芯片杂交、清洗和扫描: 选取6例结直肠癌、6例癌旁正常组织和3例腺瘤组织进行芯片检测. 将标记好的PCR产物洗涤后加到芯片上60 ℃杂交30 min, 45 ℃下杂交14 h, 将芯片用XC4和UB2缓冲液进行清洗, 真空干燥器干燥. 用高精度激光共聚焦扫描仪(Illumina scanner: 0.8 μmol/L)对杂交后的芯片进行扫描及信号提取.
1.2.3 数据处理: 运用Feature Extraction软件对芯片图像进行分析, 把图像信号转化为数字信号.
1.2.4 荧光定量RT-PCR: 挑选差异表达的miRNAs中的4个miRNAs(miR-552、miR-142-3p、miR-133b和miR-139-3P)进行荧光定量RT-PCR的验证, microRNA的RT-PCR方法参照QIGEN-miScript system中操作步骤进行. 上游引物采用QIAGEN公司miScript SYBR Green PCR Kit中提供的通用引物, 相应的下游引物由上海英俊生物技术有限公司合成, 引物序列见表1, 采用U6 RNA作为内标进行归一化. 结果运用QIGEN软件数据库分析. 判断标准为癌组织与癌旁组织的表达量比值>2为表达上调, <0.5为表达下调.
| 基因 | 引物序列 |
| Has-mir-133b | 5'-TTTGGTCCCCTTCAACCAGCTA-3' |
| Has-mir-139-3p | 5'-GGAGACGCGGCCCTGTTGGAGT-3' |
| Has-mir-142-3p | 5'-TGTAGTGTTTCCTACTTTATGGA-3' |
| Has-mir-552 | 5'-GCAACAGGTGACTGGTTAGACA-3' |
| U6 | 5'-CAAGGATGACACGCAAATTCG-3' |
统计学处理 利用随机方差模型的t检验对结直肠癌、腺瘤相对于癌旁正常组织的两组microRNA芯片数据进行分析, 计算出每个miRNA的显著性水平(P值)和误判率(false discovery rate, FDR), 根据P值筛选出两组样本的差异表达miRNAs, 差异筛选的标准为P<0.001. Illumina BeadStudio Application软件对癌和腺瘤组织microRNA芯片数据进行分析, 计算相比差异分值(Diffscore)和相比差异基因P值(Diff Pvalue)(Diffscore值为正值, 代表两组相比基因表达上调; Diffscore值为负值, 代表两组相比基因表达下调; Diffscore绝对值与Diff P值存在一定的关系: Diffscore>13, P<0.05; Diffscore>20, P<0.01; Diffscore>30, P<0.001), 根据P值筛选出两组样本的差异表达miRNAs, 差异筛选的标准为P<0.01. 定量RT-PCR结果相对表达量采用2-ΔΔCt法表示, 不同组别之间miRNAs表达的比较采用One-way ANOVA分析, P<0.05差异具有统计学意义.
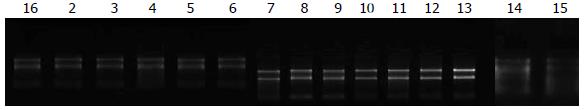
每份样本总RNA的A260/A280值在1.75-2.00, 电泳结果显示每份样品总RNA均有清晰的28S和18S条带(图1), RNA完好无降解, 其质量符合microRNA芯片的质量要求.
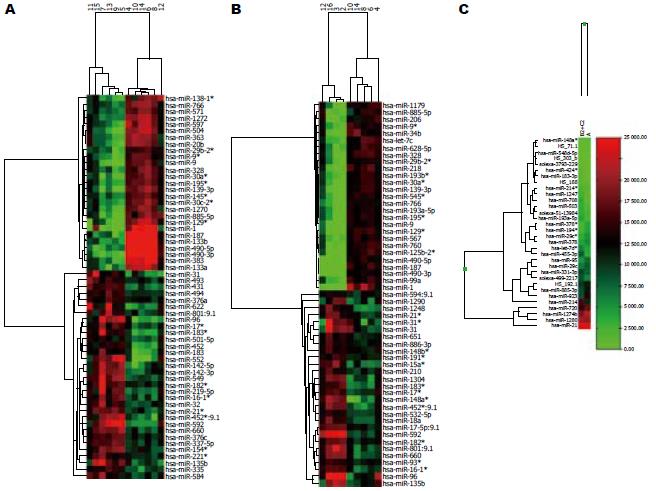
有65种miRNAs在结直肠癌和癌旁正常组织中有异常表达, 其中高表达有35个, 低表达30个(P<0.001). 而腺瘤与正常结肠组织相比, 有55个miRNAs表达有差异, 其中上调29个, 下调26个(P<0.001). Illumina BeadStudio Application软件分析发现与腺瘤相比, 结直肠癌中有25个miRNAs异常表达, 其中13个上调, 12个下调(P<0.01). 进一步分析结果显示相对于癌旁正常组织, 在肿瘤组织(结直肠癌和腺瘤)中同时异常表达的有25个miRNAs, 其中高表达12个, 分别为miR-592, miR-96, miR-801:9.1, miR-31, miR-135b, miR-660, miR-452*:9.1, miR-183*, miR-16-1*, miR-21*, miR-182*, miR-17*, 低表达有13个, 分别为miR-29b-2*, miR-195*, miR-9*, miR-885-5p, miR-328, miR-766, miR-30a*, miR-139-3p, miR-9, miR-1, miR-490-5p, miR-490-3p, miR-187(图2).
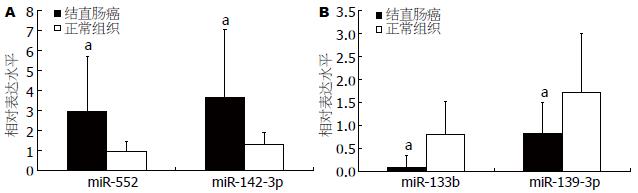
在结直肠癌和癌旁组织中明显异常表达的4个miRNAs包括miR-133b, miR-139-3p, miR-142-3p, miR-552, 并采用U6进行归一化. miRNAs定量PCR扩增曲线和溶解曲线均为单一峰, 说明PCR扩增特异性好. PCR结果显示与癌旁正常组织相比在结直肠癌组织中miR-552和miR-142-3p表达均上调(2.97±2.72 vs 0.98±0.48; 3.64±3.41 vs 1.31±0.61, 均P<0.05, 图3A), 而miR-133b和miR-139-3p下调(0.10±0.26 vs 0.82±0.70; 0.81±0.67 vs 1.71±1.29, 均P<0.05, 图3B), microRNA芯片结果与定量RT-PCR结果一致.
进一步将miR-133b, miR-139-3p, miR-142-3p, miR-552的表达与25例结直肠癌患者的临床病理资料进行对比分析, 其结果显示: miR-552表达与肿瘤的TNM分期、淋巴结和肝转移有关(P<0.05),而miR-142-3p表达与肿瘤的组织学分化程度有关(P<0.05), 与其他临床病理参数无关. miR-133b表达与患者年龄有关(P<0.05), miR-139-3p表达与TNM分期和肝转移有关(P<0.05), 与其他病理参数无关(表2).
| P值 | |||||
| miR-552 | miR-142-3p | miR-133b | miR-139-3p | ||
| 年龄(岁) | 0.642 | 0.193 | 0.028 | 0.429 | |
| >50 | 21 | ||||
| <50 | 4 | ||||
| 性别 | 0.471 | 0.391 | 0.567 | 0.252 | |
| 男 | 6 | ||||
| 女 | 19 | ||||
| 组织学分化程度 | 0.755 | 0.000 | 0.680 | 0.303 | |
| 高 | 1 | ||||
| 中 | 19 | ||||
| 低 | 5 | ||||
| TNM分期 | 0.024 | 0.856 | 0.476 | 0.012 | |
| Ⅰ期 | 5 | ||||
| Ⅱ期 | 8 | ||||
| Ⅲ期 | 7 | ||||
| Ⅳ期 | 5 | ||||
| 淋巴结转移 | 0.004 | 0.728 | 0.255 | 0.057 | |
| 有 | 12 | ||||
| 无 | 13 | ||||
| 肝转移 | 0.016 | 0.882 | 0.466 | 0.001 | |
| 有 | 5 | ||||
| 无 | 20 | ||||
| 肿瘤大小(cm) | 0.136 | 0.239 | 0.585 | 0.811 | |
| >5 | 14 | ||||
| <5 | 11 | ||||
| 浸润深度 | 0.268 | 0.542 | 0.614 | 0.178 | |
| 肌层 | 6 | ||||
| 全层 | 19 | ||||
目前miRNAs的异常表达已经在一些血液系统和实体肿瘤中有过报道, 国外学者也已在消化系肿瘤中对于miRNAs的作用作了相关研究[13-18], 他们发现了一些与各个器官肿瘤形成有关的特异表达的miRNAs, 认为miRNAs的异常表达可能参与了食管、胃、结直肠、肝以及胰腺等肿瘤的形成过程, 由此丰富了这些消化系肿瘤的发病机制, 为更全面的认识这些肿瘤的内在分子机制提出了新的思路.
本研究中我们采用了腺瘤和结直肠癌来与癌旁正常组织进行对比研究, 以期寻找一些与结直肠癌发生和发展相关的miRNAs, 从而发现具有早期诊断意义的标志物, 甚至可作为治疗靶点的miRNAs. 通过芯片结果分析我们发现有65个miRNAs在癌与癌旁正常组织中差异表达, 其中35个miRNAs在结直肠癌中上调, 30个下调, 25个miRNAs在癌与腺瘤中差异表达, 而有55个miRNAs在腺瘤与正常组织中差异表达. 进一步分析我们发现有25个miRNAs相对于正常组织同时在结直肠癌和腺瘤中异常表达, 其中高表达的有12个, 分别为miR-592, miR-96, miR-801:9.1, miR-31, miR-135b, miR-660, miR-452*:9.1, miR-183*, miR-16-1*, miR-21*, miR-182*, miR-17*, 低表达有13个, 分别为miR-29b-2*, miR-195*, miR-9*, miR-885-5p, miR-328, miR-766, miR-30a*, miR-139-3p, miR-9, miR-1, miR-490-5p, miR-490-3p, miR-187. 以上结果表明了这些异常表达的miRNAs不仅参与了结直肠黏膜细胞从正常形态开始转化的早期过程, 导致了结直肠腺瘤的形成, 而且他们的持续异常表达作用下对于结直肠腺瘤的最终恶变也起了关键作用, 因而这25个miRNAs的异常表达可能是结直肠癌发生的早期事件, 均可能具有结直肠癌早期诊断的意义, 通过对这些miRNAs的检测有望尽早发现早期结直肠癌, 从而进行早期干预达到提高生存率的目的. 而另一些相对于结直肠腺瘤在结直肠癌中异常表达的miRNAs(如miR-let-7d*, miR-214, miR-95, miR-29c等)则可能在以上miRNAs异常表达的基础上, 进一步使得这些癌前病变组织获得了恶变的潜能, 导致了结直肠癌的发生, 是结直肠肿瘤形成过程中的重要步骤, 在临床上检测腺瘤组织中这些异常表达的miRNAs的表达, 有助于早期区分良、恶性组织, 从而能够发现相对早期的结直肠癌, 早期进行干预如行早期黏膜下切除或手术切除, 减少了将来腺瘤的恶变和浸润性肿瘤的发生, 对于减少结直肠癌的发生和提高早期诊断率以及总体生存率均具有重要的意义.
实际上既往研究还认为肿瘤组织的miRNAs表达具有组织特异性[19,20], 他表示的含义之一即不同的肿瘤组织来源其miRNAs的表达谱往往不同, 这也是miRNAs在肿瘤研究中的优势之一, 通过对于肿瘤组织中miRNAs表达谱的研究, 我们不仅可以区分良、恶性组织, 而且对于一些来源不明的肿瘤组织, 通过其miRNAs表达谱的研究可能判断其起源部位. 因此, 这对于提高临床上一些起源不明的转移性肿瘤的治疗效果和预后有着重要的意义. Rosenfeld等[21]发现83例含22种不同肿瘤组织中, 有48个miRNAs的表达可以成功地将他们区分开来, 准确率可达90%, 且由于miRNAs的数量明显少于可编码蛋白的基因, 体现出较检测编码蛋白的基因的优越性. 此外, 类似的研究还发现miRNAs的检测在区分组织类型不明的低分化肿瘤时明显优于mRNA表达的检测[22]. 既往研究发现miR-21, miR-191多在胰腺癌中异常表达[17], 乳腺癌中常有miR-125, miR-145异常表达[23], 这就在一定程度上体现了这些miRNAs的相对特异性. 本研究中, 我们发现相对于正常结肠组织在结直肠癌异常表达的miRNAs中, 也具有一些类似特点. 其中一部分miRNAs在其他系统肿瘤中少有报道, 却在结直肠癌组织中多见异常表达[24-27], 他们包括13个miRNAs, 其中上调为miR-96, miR-31, miR-183, miR-135b, miR-142-3p, miR-335, miR-32, 下调的有miR-9, miR-9*, miR-1, miR-133a, miR-133b, miR-187. 在这些异常表达的miRNAs中, 研究发现miR-96, miR-31, miR-183和miR-135b的同时异常表达在结直肠癌变中较为多见[24,25], 这与本研究结果相似, 由此可见检测这些miRNAs的表达有助于对于结直肠癌组织类型的特异判断, 体现了他们对于结直肠癌的相对组织特异性. 文献报道和本研究发现miR-592与结直肠癌发病相关[25], 但是在其他肿瘤中未见相关报道, 一定程度上也体现了其在结直肠组织的特异性表达. 因此, 我们有理由认为在结直肠癌中也具有其组织特异性的miRNAs表达谱, 在组织中检测一组包括miR-96, miR-31, miR-183, miR-592, miR-135b, miR-133b的表达, 不仅对于其早期结直肠癌的诊断有帮助, 而且对于其组织来源也提供了一定信息, 他们同时异常表达可能提示来源于结直肠组织的恶变.
第1个报道miRNAs在结直肠癌中异常表达的是2003年Michael等[28], 他们发现在结直肠癌组织中有2个miRNAs, 即miR-143和miR-145, 出现明显下调. 随后多个学者相继做了一些研究[29-31], 但是他们的研究结果中miRNAs表达谱不尽相同. 本研究结果中我们发现有65个miRNAs相对于正常结直肠组织在结直肠癌中异常表达, 其中有13个与既往的研究相同, 包括: miR-96, miR-31, miR-592, miR-135b, miR-183, miR-142-3p, miR-335, miR-32, miR-1, miR-9, miR-133a, miR-133b和miR-187, 而在既往研究中, 另外52个miRNAs未见在结直肠癌中有相关报道, 说明本研究结果与其他学者的研究也有不同之处, 究其原因可能有几个方面. 首先, 由于种族和地区的差异, 所选取的患者其本身也存在遗传学的异质性. 其次, 由于各个试验所采用方法的不同也会导致结果的差异, 例如使用芯片的种类不同, 就可以导致芯片筛选的结果不同, 我们此次使用的illumina芯片含有1146个miRNAs, 涉及的miRNAs是根据目前最新的miRNAs版本进行设计的, 不仅有确定的成熟miRNAs, 也包括了一些测序具有miRNAs特征但尚未最终确定的小RNAs, 所以与一些试验采用的自行设计的芯片或Agilent芯片等结果存在一定的差异. 最后, 在确定筛选差异基因的统计学方法上可能有不同, 也与研究者确定的差异显著性范围有关, 如P值的确定(<0.01或0.05)等. 解决以上问题的关键在于建立一个芯片检测的全球"金标准", 如统一的数据库和统计方法等.
本研究中, 我们进一步发现miR-552与结直肠癌的TNM分期、淋巴结和肝转移有关, miR-139-3p与TNM分期和肝转移有关, 表明了他们的异常表达可能与结直肠癌的进一步转移有关, 具有潜在的预后意义, 提示miR-552和miR-139-3p异常表达的患者可能预后差. 此外, miR-142-3p不仅可以区分肿瘤和正常组织, 而且我们还发现他在不同的分期中表达也有所不同, 提示他可能不仅参与了结直肠癌的早期发生过程, 而且在肿瘤的进一步进展中也起到了一定的作用. 至于在不同年龄患者中miR-133b的表达, >50岁年龄组表达明显低于<50岁年龄组, 这可能反应了不同年龄段患者肿瘤发生和发展的内在分子变化的不同. 当然由于本试验中的样本数量的限制, 这些异常表达的miRNAs的临床意义尚有待于更大规模的试验去证实, 其他一些异常表达的miRNAs的临床意义也有待于今后的大规模样本去发现.
总之, 通过本研究我们成功地构建了结直肠癌、结直肠腺瘤和癌旁正常组织的miRNAs表达谱, 发现了一些结直肠癌差异表达的miRNAs, 推测他们可能具有结直肠癌的早期诊断意义和成为潜在的靶向治疗靶点, 为miRNAs参与结直肠癌的发生提供了一定的理论依据, 丰富了对结直肠癌的发病机制的阐述. 相信随着越来越多学者进一步地深入研究, 我们对于miRNAs在结直肠癌中作用的认识会越加清晰, 从而为miRNAs在结直肠癌的早期诊断和开发新的治疗靶点带来更多的证据.
目前为止对于结直肠癌尚无一个确切的具有早期诊断意义的临床指标以及有明确疗效的治疗靶点. 近来研究发现微小核糖核酸(miRNAs)可以结合到靶基因中发挥调控靶基因表达的功能, 从而参与肿瘤的发生和发展.
王晓艳, 副教授, 湖南长沙中南大学湘雅三医院消化内科
目前在miRNAs与肿瘤关系的研究领域中, 重点在于寻找与各种肿瘤发生、发展相关的特异miRNAs, 期望获得具有早期诊断意义的标志物以及具有治疗干预的新靶点. 亟待进一步研究的是建立统一的研究标准. 此外, 对于特异的miRNAs的功能及其与靶点之间的复杂调控网络有待于进一步明确.
Rosenfeld等发现83例含22种不同肿瘤组织中, 有48个miRNAs的表达可以成功地将他们区分开来, 准确率可达90%, 且由于miRNAs的数量明显少于可编码蛋白的基因, 体现出较检测编码蛋白的基因的优越性.
本研究创新之处在于采用了目前最新的miRNAs芯片技术. 此外, 同时研究了结直肠3种组织(正常、癌前病变及癌组织)的miRNAs表达, 而既往的研究中较少同时研究这3种不同生物学行为的组织.
本文发现了一些在结直肠中特异表达的miRNAs, 有助于在将来临床标本中进一步扩大样本验证, 从而获得具有真正临床意义的早期诊断标志物和治疗靶点. 一旦证实后可以通过RT-PCR等方法在临床上检测患者的血液或实体标本来提高结直肠癌的早期诊断率.
本文为在转录后层面揭示结肠癌发生发展的机制奠定了一定的基础, 具有一定的科研价值.
编辑: 李薇 电编:何基才
| 1. | Derks S, Postma C, Moerkerk PT, van den Bosch SM, Carvalho B, Hermsen MA, Giaretti W, Herman JG, Weijenberg MP, de Bruïne AP. Promoter methylation precedes chromosomal alterations in colorectal cancer development. Cell Oncol. 2006;28:247-257. [PubMed] |
| 2. | Hermsen M, Postma C, Baak J, Weiss M, Rapallo A, Sciutto A, Roemen G, Arends JW, Williams R, Giaretti W. Colorectal adenoma to carcinoma progression follows multiple pathways of chromosomal instability. Gastroenterology. 2002;123:1109-1119. [PubMed] [DOI] |
| 3. | Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S. The nuclear RNase III Drosha initiates microRNA processing. Nature. 2003;425:415-419. [PubMed] [DOI] |
| 4. | Lund E, Güttinger S, Calado A, Dahlberg JE, Kutay U. Nuclear export of microRNA precursors. Science. 2004;303:95-98. [PubMed] [DOI] |
| 5. | Lai EC. Micro RNAs are complementary to 3' UTR sequence motifs that mediate negative post-transcriptional regulation. Nat Genet. 2002;30:363-364. [PubMed] [DOI] |
| 6. | Liu J, Rivas FV, Wohlschlegel J, Yates JR 3rd, Parker R, Hannon GJ. A role for the P-body component GW182 in microRNA function. Nat Cell Biol. 2005;7:1261-1266. [PubMed] [DOI] |
| 7. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 8. | Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, Shimizu M, Rattan S, Bullrich F, Negrini M. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci U S A. 2004;101:2999-3004. [PubMed] [DOI] |
| 9. | Cho WC. OncomiRs: the discovery and progress of microRNAs in cancers. Mol Cancer. 2007;6:60. [PubMed] [DOI] |
| 10. | Hammond SM. MicroRNAs as oncogenes. Curr Opin Genet Dev. 2006;16:4-9. [PubMed] [DOI] |
| 11. | Kumar MS, Lu J, Mercer KL, Golub TR, Jacks T. Impaired microRNA processing enhances cellular transformation and tumorigenesis. Nat Genet. 2007;39:673-677. [PubMed] [DOI] |
| 12. | Wang Y, Lee CG. MicroRNA and cancer--focus on apoptosis. J Cell Mol Med. 2009;13:12-23. [PubMed] [DOI] |
| 13. | Feber A, Xi L, Luketich JD, Pennathur A, Landreneau RJ, Wu M, Swanson SJ, Godfrey TE, Litle VR. MicroRNA expression profiles of esophageal cancer. J Thorac Cardiovasc Surg. 2008;135:255-260; discussion 260. [PubMed] [DOI] |
| 14. | Katada T, Ishiguro H, Kuwabara Y, Kimura M, Mitui A, Mori Y, Ogawa R, Harata K, Fujii Y. microRNA expression profile in undifferentiated gastric cancer. Int J Oncol. 2009;34:537-542. [PubMed] |
| 15. | Asangani IA, Rasheed SA, Nikolova DA, Leupold JH, Colburn NH, Post S, Allgayer H. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer. Oncogene. 2008;27:2128-2136. [PubMed] [DOI] |
| 16. | Su H, Yang JR, Xu T, Huang J, Xu L, Yuan Y, Zhuang SM. MicroRNA-101, down-regulated in hepatocellular carcinoma, promotes apoptosis and suppresses tumorigenicity. Cancer Res. 2009;69:1135-1142. [PubMed] [DOI] |
| 17. | Bloomston M, Frankel WL, Petrocca F, Volinia S, Alder H, Hagan JP, Liu CG, Bhatt D, Taccioli C, Croce CM. MicroRNA expression patterns to differentiate pancreatic adenocarcinoma from normal pancreas and chronic pancreatitis. JAMA. 2007;297:1901-1908. [PubMed] |
| 18. | Meng F, Henson R, Lang M, Wehbe H, Maheshwari S, Mendell JT, Jiang J, Schmittgen TD, Patel T. Involvement of human micro-RNA in growth and response to chemotherapy in human cholangiocarcinoma cell lines. Gastroenterology. 2006;130:2113-2129. [PubMed] [DOI] |
| 19. | Liu CG, Calin GA, Meloon B, Gamliel N, Sevignani C, Ferracin M, Dumitru CD, Shimizu M, Zupo S, Dono M. An oligonucleotide microchip for genome-wide microRNA profiling in human and mouse tissues. Proc Natl Acad Sci U S A. 2004;101:9740-9744. [PubMed] [DOI] |
| 20. | Barad O, Meiri E, Avniel A, Aharonov R, Barzilai A, Bentwich I, Einav U, Gilad S, Hurban P, Karov Y. MicroRNA expression detected by oligonucleotide microarrays: system establishment and expression profiling in human tissues. Genome Res. 2004;14:2486-2494. [PubMed] [DOI] |
| 21. | Rosenfeld N, Aharonov R, Meiri E, Rosenwald S, Spector Y, Zepeniuk M, Benjamin H, Shabes N, Tabak S, Levy A. MicroRNAs accurately identify cancer tissue origin. Nat Biotechnol. 2008;26:462-469. [PubMed] [DOI] |
| 22. | Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, Sweet-Cordero A, Ebert BL, Mak RH, Ferrando AA. MicroRNA expression profiles classify human cancers. Nature. 2005;435:834-838. [PubMed] [DOI] |
| 23. | Iorio MV, Ferracin M, Liu CG, Veronese A, Spizzo R, Sabbioni S, Magri E, Pedriali M, Fabbri M, Campiglio M. MicroRNA gene expression deregulation in human breast cancer. Cancer Res. 2005;65:7065-7070. [PubMed] [DOI] |
| 24. | Bandrés E, Cubedo E, Agirre X, Malumbres R, Zárate R, Ramirez N, Abajo A, Navarro A, Moreno I, Monzó M. Identification by Real-time PCR of 13 mature microRNAs differentially expressed in colorectal cancer and non-tumoral tissues. Mol Cancer. 2006;5:29. [PubMed] [DOI] |
| 25. | Sarver AL, French AJ, Borralho PM, Thayanithy V, Oberg AL, Silverstein KA, Morlan BW, Riska SM, Boardman LA, Cunningham JM. Human colon cancer profiles show differential microRNA expression depending on mismatch repair status and are characteristic of undifferentiated proliferative states. BMC Cancer. 2009;9:401. [PubMed] [DOI] |
| 26. | Bandres E, Agirre X, Bitarte N, Ramirez N, Zarate R, Roman-Gomez J, Prosper F, Garcia-Foncillas J. Epigenetic regulation of microRNA expression in colorectal cancer. Int J Cancer. 2009;125:2737-2743. [PubMed] [DOI] |
| 27. | Schepeler T, Reinert JT, Ostenfeld MS, Christensen LL, Silahtaroglu AN, Dyrskjøt L, Wiuf C, Sørensen FJ, Kruhøffer M, Laurberg S. Diagnostic and prognostic microRNAs in stage II colon cancer. Cancer Res. 2008;68:6416-6424. [PubMed] [DOI] |
| 28. | Michael MZ, O' Connor SM, van Holst Pellekaan NG, Young GP, James RJ. Reduced accumulation of specific microRNAs in colorectal neoplasia. Mol Cancer Res. 2003;1:882-891. [PubMed] |
| 29. | Cummins JM, He Y, Leary RJ, Pagliarini R, Diaz LA Jr, Sjoblom T, Barad O, Bentwich Z, Szafranska AE, Labourier E, Raymond CK, Roberts BS, Juhl H, Kinzler KW, Vogelstein B, Velculescu VE. The colorectal microRNAome. Proc Natl Acad Sci U S A. 2006;103:3687-3692. [PubMed] [DOI] |
| 30. | Akao Y, Nakagawa Y, Naoe T. MicroRNA-143 and -145 in colon cancer. DNA Cell Biol. 2007;26:311-320. [PubMed] [DOI] |