修回日期: 2009-11-21
接受日期: 2009-11-30
在线出版日期: 2010-01-18
目的: 探讨微小RNA(microRNA, miRNA)在人结肠癌干细胞中的表达, 为进一步研究miRNA调控结肠癌干细胞向结肠癌细胞分化的分子机制奠定基础.
方法: 应用miRNA表达谱芯片检测人结肠癌干细胞和已分化结肠癌细胞中miRNA的表达谱. 利用实时定量PCR技术检测两种细胞中差异表达的miRNA, 验证miRNA芯片结果的可靠性. 应用软件对筛选出的显著差异表达miRNA的靶基因进行预测.
结果: 与已分化结肠癌细胞相比, 人结肠癌干细胞中表达上调超过1.5倍的miRNA有35个, 为: hsa-miR-192, hsa-miR-29b, hsa-miR-215, hsa-miR-194, hsa-miR-33a, hsa-miR-32等; 表达下调超过1.5倍的miRNA有11个, 为: hsa-miR-93, hsa-miR-1231, hsa-miR-524-3p, hsa-miR-886-3p等. PCR技术验证, 与miRNA芯片结果相符合. 表达显著上调miRNA的共同靶mRNA有: AFF2、MTF1、RUNDC2C和ZFHX4. 表达显著下调miRNA的共同靶mRNA有: ONECUT2、SH3TC2、PTPRT、RNABP10、NR3C1、RGSL1、RNASEL和 TANC2.
结论: 筛选出的差异表达miRNA可能参与结肠癌的发病, 为该病诊治提供了新的思路. 其共同靶基因可能具有重要的调控结肠癌干细胞生长和分化的作用.
引文著录: 邹健, 于晓峰, 虞阳, 张颖, 史冬梅, 董洁, 李天奇, 刘雪静. MicroRNA在人结肠癌干细胞中的表达谱. 世界华人消化杂志 2010; 18(2): 173-178
Revised: November 21, 2009
Accepted: November 30, 2009
Published online: January 18, 2010
AIM: To identify microRNAs differentially expressed in human colon cancer stem cells to provide a basis for further study of the role of microRNAs in the differentiation of colon cancer stem cells.
METHODS: MicroRNA microarray was used to detect the differential expression of microRNAs between human colon cancer stem cells and differentiated colon cancer cells. Real-time quantitative reverse transcription-polymerase chain reaction (RT-PCR) was performed to verify the differential expression of candidate microRNAs obtained from microarray experiment. Bioinformatic software tools were used to predict the target genes of identified microRNAs.
RESULTS: Compared with differentiated colon cancer cells, 35 microRNAs (hsa-miR-192, hsa-miR-29b, hsa-miR-215, hsa-miR-194, hsa-miR-33a, hsa-miR-32, etc.) were upregulated more than 1.5-fold, and 11 microRNAs (hsa-miR-93, hsa-miR-1231, hsa-miRPlus-F1080, hsa-miR-524-3p, hsa-miR-886-3p, hsa-miR-561, etc.) were downregulated in colon cancer stem cells. The results of real-time quantitative RT-PCR were consistent with those of microRNA microarray. The common mRNA targets of obviously upregulated microRNAs were AFF2, MTF1, RUNDC2C and ZFHX4, while those of obviously downregulated microRNAs were ONECUT2, SH3TC2, PTPRT, RNABP10, NR3C1, RGSL1, RNASEL and TANC2.
CONCLUSION: Some microRNAs are differentially expressed and may play important roles during the differentiation of colon cancer stem cells towards colon cancer cells.
- Citation: Zou J, Yu XF, Yu Y, Zhang Y, Shi DM, Dong J, Li TQ, Liu XJ. Expression profile of microRNAs in human colon cancer stem cells. Shijie Huaren Xiaohua Zazhi 2010; 18(2): 173-178
- URL: https://www.wjgnet.com/1009-3079/full/v18/i2/173.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i2.173
肿瘤干细胞(cancer stem cell, CSC)是在造血干细胞(hematopoietic stem cell, HSC)、白血病干细胞(leukemic stem cell, LSC)概念的基础上总结延伸出来的, 这一概念试图从一个全新的角度诠释癌细胞的起源、癌组织的异质性、部分癌细胞的永生性和转移能力差异等肿瘤生物学行为, 一经提出立即引起广大科研工作者的浓厚兴趣[1-5]. 微小RNA(microRNA, miRNA)是一种包含19-25个碱基的单链非编码小分子RNA, 由具有发夹结构的70-90个碱基大小的单链RNA前体经过酶切加工后生成. miRNA参与生命过程中一系列的重要进程, 包括发育、造血、器官形成、凋亡、细胞增殖, 甚至肿瘤发生[6-8]. 近年, 有研究显示miRNA参与肿瘤干细胞的自我更新、多能性维持、分化和分裂增殖等过程的调控[9,10]. 本研究采用miRNA表达谱芯片技术对前期分离的人结肠癌干细胞, 以及已分化的结肠癌细胞中miRNA表达谱进行检测, 以发现可能参与调控结肠癌干细胞定向分化为结肠癌细胞的相关miRNA, 为阐明结肠癌干细胞向结肠癌细胞分化的miRNA调控机制奠定基础.
人结肠癌细胞株(SW1116)购自中国科学院上海生科院细胞资源中心. 人结肠癌干细胞株(SW1116csc)为本课题组前期应用无血清的肿瘤干细胞培养基, 从SW1116细胞株中分离出, 并对其进行了纯化和鉴定. SW1116csc细胞置于含各种生长因子的无血清DMEM/F12(1:1, Invitrogen-Gibco)培养基中培养; SW1116细胞培养于含100 mL/L胎牛血清的DMEM/F12(1:1)培养基中. 两种细胞在含50 mL/L CO2、950 mL/L空气的CO2孵箱中, 在37 ℃、95%湿度条件下培养.
1.2.1 细胞总RNA的抽提及质量检测: SW1116细胞按每毫升1×104个细胞、每瓶5 mL接种于25 cm2培养瓶, 48 h后采用TRIzol试剂(Invitrogen)按操作说明书抽取总RNA. SW1116csc细胞每瓶1×105个, 悬浮培养96 h后, 离心收集细胞抽取总RNA. 每瓶细胞加入1 mL TRIzol, 吸取细胞裂解液转入Eppendoff管, 加入200 µL氯仿, 混匀后置室温5 min. 4 ℃, 12 000 g离心15 min. 吸取上层水相至新Eppendoff管, 加入0.5 mL异丙醇, 混匀, 4 ℃, 12 000 g离心10 min使RNA沉淀, 倾去上清液, 750 mL/L乙醇洗涤, 空气中干燥15 min. 加入50 µL无RNA酶的去离子水, 重新溶解RNA, 紫外/可见分光光度计(Ultraspec 2000型, Pharmacia Biotech)测定RNA浓度. 甲醛变性凝胶电泳检测RNA纯度. RNA产物置-70 ℃冰箱保存.
1.2.2 miRNA芯片检测结肠癌干细胞表达谱: 应用丹麦Exiqon公司生产的miRNA芯片miRCURYTM LNA Array(v13.0)进行检测, 每张miRNA芯片中包含有至少1 700个特异性探针、435个Exiqon特有的miRPlusTM探针(提供miRBase数据库外的新miRNA信息)、对照探针以及无探针的空白对照, 可以检测Sanger miRBase 13.0数据库中人、小鼠、大鼠全部miRNA(其中人约847条, 小鼠约609条, 大鼠约351条). 每个探针在芯片内重复4次, 即每张芯片对同一样本重复检测4次. 分别取5 µg上述2种细胞样本的总RNA, 采用miRCURYTM Hy3TM/Hy5TM Power labeling kit(Exiqon)标记H荧光, RNeasy Mini Kit(Qiagen)浓缩标记样品, 然后应用miRCURYTM Array microarray kit(Exiqon)和Hybridization Chamber Ⅱ(Ambion)进行芯片杂交. 具体步骤按各试剂说明书进行. 杂交后芯片用Axon GenePix 4000B microarray scanner (Molecular Devices)进行图像扫描, 所得数据使用Genepix Pro V6.0软件(Molecular Devices)分析, 通过原始值减去背景值来做修正, 并用中值做标准化, 分别计算出两种样本中miRNA的标准值及比值.
1.2.3 荧光实时定量PCR检测目的miRNA表达量: 取100 ng总RNA, 应用miRNA Isolation Kit(Ambion)分离小于100 nt的小分子RNA, 然后应用SuperScript Ⅲ Reverse Trandcriptase Kit(Invitrogen)逆转录合成cDNA, 再进行定量PCR检测. 引物序列见表1, PCR条件: 95 ℃ 15 s, 60 ℃ 30 s, 72 ℃ 3 s, 共40个循环. 采用U6 RNA作为内标, 进行归一化. 样品目的基因的相对表达率(relative expression, RE)采用ΔΔCT方法计算, RE = 2ΔΔCT(CT表示反应荧光强度显著大于背景值时的循环数, ΔCT sample = CT sample-CT U6sample, ΔCT control = CT control-CT U6 control, ΔΔCT = ΔCT sample-ΔCT control).
| 引物 | 5'→3' |
| hsa-miR-93-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTACCT |
| hsa-miR-93-F | ACACTCCAGCTGGGCAAAGTGCTGTTCGTGC |
| hsa-miR-1231-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGCAGCT |
| hsa-miR-1231-F | ACACTCCAGCTGGGGTGTCTGGGCGGAC |
| hsa-miR-32-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGCAAC |
| hsa-miR-32-F | ACACTCCAGCTGGGTATTGCACATTACTAA |
| hsa-miR-33a-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGCAAT |
| hsa-miR-33a-F | ACACTCCAGCTGGGGTGCATTGTAGTTGC |
| hsa-miR-194-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTCCACA |
| hsa-miR-194-F | ACACTCCAGCTGGGTGTAACAGCAACTCCA |
| hsa-miR-215-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGTCTGT |
| hsa-miR-215-F | ACACTCCAGCTGGGATGACCTATGAATTG |
| hsa-miR-29b-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAACACT |
| hsa-miR-29b-F | ACACTCCAGCTGGGTAGCACCATTTGAAATC |
| hsa-miR-192-RT | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGGCTGT |
| hsa-miR-192-F | ACACTCCAGCTGGGCTGACCTATGAATTG |
| URP | TGGTGTCGTGGAGTCG |
| U6 | F-CTCGCTTCGGCAGCACA R-AACGCTTCACGAATTTGCGT |
1.2.4 差异表达miRNA的生物信息学分析: 对于显著差异表达的miRNA, 应用miRanda、mirBase、TargetScan和PicTar软件联合进行生物信息学分析. 搜索明显差异表达miRNA预测的作用靶基因, 对共同的靶基因进行综合预测, 选取至少3种软件的共同结果.
提取RNA的A260/A280值为1.8-2.0, 从甲醛变性琼脂糖凝胶电泳结果来看, 其18S和28S条带都很清晰, 亮度比约为2:1, 说明总RNA纯度基本达到实验要求且无明显降解(图1).
杂交后的miRNA芯片(图2)经扫描、软件分析及标准化处理后, 结果显示与已分化结肠癌细胞相比, 结肠癌干细胞中表达上调超过1.5倍的miRNA有35个, 分别为: hsa-miR-192、hsa-miR-29b、hsa-miR-215、hsa-miR-194、hsa-miR-33a、hsa-miR-32等; 表达下调超过1.5倍的miRNA有11个: hsa-miR-93、hsa-miR-1231、hsa-miRPlus-F1080、hsa-miR-524-3p、hsa-miR-886-3p、hsa-miR-561等(表2).
| miRNA | SW1116csc信号强度 | SW1116信号强度 | 表达改变倍数 |
| hsa-miR-93 | 0.46 | 8.13 | 0.06 |
| hsa-miR-1231 | 0.02 | 0.08 | 0.22 |
| hsa-miRPlus-F1080 | 0.01 | 0.05 | 0.23 |
| hsa-miR-524-3p | 0.05 | 0.16 | 0.34 |
| hsa-miR-886-3p | 3.34 | 8.37 | 0.40 |
| hsa-miR-561 | 0.02 | 0.04 | 0.45 |
| hsa-miR-497 | 0.01 | 0.03 | 0.51 |
| hsa-miR-23a | 11.92 | 22.51 | 0.53 |
| hsa-miR-886-5p | 2.89 | 5.18 | 0.56 |
| hsa-miRPlus-A1087 | 0.47 | 0.79 | 0.59 |
| hsa-miRPlus-E1170 | 30.71 | 49.93 | 0.61 |
| hsa-miRPlus-E1102 | 0.42 | 0.28 | 1.50 |
| hsa-miR-138-2 | 0.39 | 0.26 | 1.50 |
| hsa-miR-31 | 0.45 | 0.30 | 1.52 |
| hsa-miR-17 | 2.50 | 1.64 | 1.52 |
| hsa-miR-374a | 5.40 | 3.55 | 1.52 |
| hsa-miR-424 | 3.38 | 2.21 | 1.53 |
| hsa-miRPlus-F1181 | 0.64 | 0.42 | 1.53 |
| hsa-miRPlus-E1238 | 0.28 | 0.18 | 1.54 |
| hsa-miR-542-3p | 0.38 | 0.24 | 1.56 |
| hsa-miR-582-3p | 0.27 | 0.17 | 1.57 |
| hsa-miR-584 | 0.28 | 0.17 | 1.61 |
| hsa-miR-522 | 0.29 | 0.18 | 1.64 |
| hsa-miR-590-5p | 0.41 | 0.25 | 1.67 |
| hsa-miR-487b | 2.88 | 1.72 | 1.68 |
| hsa-miR-29c | 1.53 | 0.89 | 1.72 |
| hsa-miR-96 | 2.39 | 1.34 | 1.78 |
| hsa-miR-193a-3p | 22.24 | 12.50 | 1.78 |
| hsa-miR-20a | 0.74 | 0.41 | 1.79 |
| hsa-miR-301a | 2.28 | 1.27 | 1.80 |
| hsa-miRPlus-E1106 | 0.56 | 0.31 | 1.82 |
| hsa-miR-30e | 1.21 | 0.65 | 1.86 |
| hsa-miR-874 | 0.20 | 0.10 | 1.99 |
| hsa-miR-19a | 17.38 | 8.71 | 2.00 |
| hsa-miR-519a | 1.73 | 0.77 | 2.24 |
| hsa-miRPlus-A1065 | 0.30 | 0.13 | 2.32 |
| hsa-miR-521 | 0.94 | 0.40 | 2.36 |
| hsa-miR-876-5p | 0.23 | 0.10 | 2.37 |
| hsa-miR-493 | 0.28 | 0.11 | 2.53 |
| hsa-miR-101 | 4.92 | 1.28 | 3.85 |
| hsa-miR-32 | 1.89 | 0.35 | 5.48 |
| hsa-miR-33a | 3.97 | 0.64 | 6.16 |
| hsa-miR-194 | 0.79 | 0.11 | 7.11 |
| hsa-miR-215 | 0.97 | 0.11 | 9.09 |
| hsa-miR-29b | 3.00 | 0.33 | 9.20 |
| hsa-miR-192 | 1.47 | 0.14 | 10.67 |
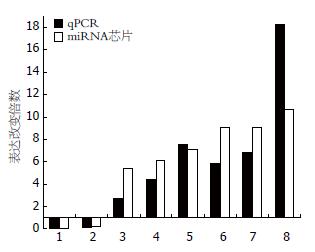
取上述miRNA芯片检测的2种细胞总RNA为模板, 实时定量PCR检测在芯片结果中显著差异表达的miRNA(图3). 提示定量PCR与miRNA芯片结果基本一致, 表明miRNA芯片的结果具有可靠性.
表达显著上调的miRNA(hsa-miR-192, hsa-miR-29b, hsa-miR-215, hsa-miR-194, hsa-miR-33a, hsa-miR-32)共同靶mRNA有: AFF2、MTF1、RUNDC2C和ZFHX4. 表达显著下调的miRNA(hsa-miR-93, hsa-miR-1231, hsa-miR-524-3p, hsa-miR-886-3p)共同靶mRNA有: ONECUT2、SH3TC2、PTPRT、RNABP10、NR3C1、RGSL1、RNASEL和TANC2.
miRNA作为一类成熟状态下只有22 nt左右的非编码单链小RNA, 广泛存在于从线虫、植物、动物到人类的多种生物中, 其在个体生长发育、细胞增殖凋亡、炎症和肿瘤等多种生理病理过程中起着十分重要的作用, 受到广泛关注. 从Du等[11]在线虫中发现第1个miRNA(lin-4)开始, 截止2009-09, 已经在115个物种中发现miRNA共约10 883个, 其中人类约800个(数据来源于MiRBase Sequence Database 14, http://www.mirbase.org/), 据估计这些miRNA具有调控人类至少30%基因的潜能[12]. 大量研究发现, 在多种人类肿瘤都存在miRNA的表达异常现象. 如: 弥漫性大B细胞淋巴瘤中miR-155表达上调[13]、胰腺癌中miR-221表达上调[14]、肺癌中的let-7表达下调[15,16]、结肠直肠癌中的miR-143和miR-145表达下调[17]、慢性淋巴细胞白血病中的miR-15a[18]和miR-16-1表达下调[19]等, 提示miRNA在肿瘤的发生发展中可能发挥类似癌基因或抑癌基因的作用. 在正常情况下, 成熟的miRNA通过调控靶基因mRNA的翻译或其稳定性来参与正常细胞稳态的维持, 故而miRNA的异常表达可能导致其相应靶基因转录后水平的表达异常. 目前, 对miRNA在肿瘤发生发展中的具体作用机制还不是太清楚, 认为其主要作用机制为: miRNA通过RNA诱导的沉默复合物(RNA-induced silencing complex, RISC)与靶基因mRNA的3'端UTR区互补结合[20], 当两者能完全互补配对结合时, 导致靶基因mRNA的降解, 这种现象在植物中多见; 在动物中则多数是通过两者的不完全互补结合, 抑制靶基因mRNA的翻译, 进而影响靶基因蛋白的表达. 近年来, 有学者研究发现在胚胎干细胞(ES细胞)分化过程中, 一些miRNA是未分化ES细胞所特有的, 并随着细胞分化而逐渐下调, 提示miRNA可能在维持ES细胞全能性和自我更新能力中起重要作用, 而这类miRNA也可作为ES细胞的分子标志[21,22]. 此外, 在造血细胞、脂肪、神经、肌肉和心肌细胞分化过程中也发现miRNA的参与并发挥着关键作用[23-27]. 如发现肌肉特异性miR-206是调控肌细胞和心肌细胞发育的关键分子[23,24]; 在造血干细胞分化过程中; 沿不同谱系发育受不同的miRNA调控, miR-146调控淋巴系前体向T细胞、NK细胞和B细胞分化, miR-155/24a/17则调控髓系前体向粒细胞、巨噬细胞和红细胞发育[25]. 但是, 对于miRNA在肿瘤干细胞中的表达情况, 目前研究比较少. 本研究采用miRNA表达谱芯片技术筛选人结肠癌干细胞分化相关miRNA, 通过检测发现表达改变的miRNA有46个, 其中: 表达上调的miRNA 35个; 表达下调11个. 这些miRNA有可能对结肠癌干细胞的分化起重要的调控作用. 通过miRanda、mirBase、TargetScan和PicTar软件检索, 发现了表达显著改变miRNA的共同作用的靶基因. 结肠癌干细胞向结肠癌细胞的定向分化, 有可能通过这些基因起作用, 其作用机制需要更进一步的实验来阐明. 总之, 本研究通过对前期分离的人结肠癌干细胞miRNA表达谱进行检测, 发现了多条表达改变的特异性miRNA, 并进行了初步验证, 为阐明结肠癌干细胞向结肠癌细胞定向分化的miRNA调控机制奠定了基础.
肿瘤中存在一类数目极少的干细胞样细胞群, 具有无限增殖的能力和多向分化的潜能, 是肿瘤发生发展、侵袭转移和复发耐药的根本动力, 称之为肿瘤干细胞(CSC). 微小RNA(miRNA)是一类非编码的小分子RNA, 他与靶mRNA的3'非翻译区互补配对, 从而抑制蛋白质合成或诱导mRNA降解. 研究发现miRNA对肿瘤干细胞的增殖和分化起重要的调控作用.
曹秀峰, 教授, 南京医科大学附属南京第一医院肿瘤中心; 刘宝瑞, 教授, 南京大学医学院附属鼓楼医院肿瘤中心
大量研究发现, 在多种人类肿瘤都存在miRNA的表达异常现象, 后者起着癌基因或抑癌基因的作用. 但是, 对于miRNA在肿瘤干细胞中的表达以及其在增殖和定向分化中的调控机制亟待研究.
目前已发现的miRNA表达异常现象有: 弥漫性大B细胞淋巴瘤中miR-155表达上调、胰腺癌中miR-221表达上调、肺癌中的let-7表达下调、结肠直肠癌中的miR-143和miR-145表达下调、慢性淋巴细胞白血病中的miR-15a和miR-16-1表达下调等. 在某些肿瘤干细胞中也已证实存在特异性miRNA的表达.
本研究通过对前期分离的人结肠癌干细胞miRNA表达谱进行检测, 发现了多条表达改变的特异性miRNA, 为阐明结肠癌干细胞向结肠癌细胞定向分化的miRNA调控机制奠定了基础.
本研究发现人结肠癌干细胞分化相关miRNA中表达改变的有46个, 其中, 表达上调的miRNA有35个, 表达下调11个. 并发现了表达显著改变miRNA的共同作用的靶基因. 有可能作为肿瘤治疗的新的靶位点, 为结肠癌的诊治提供了新的思路.
本文研究了MicroRNA在人结肠癌干细胞中的表达谱, 选题新颖、简明而有特色, 结论具有较好的应用价值和可读性.
编辑: 李军亮 电编:何基才
| 1. | O'Brien CA, Pollett A, Gallinger S, Dick JE. A human colon cancer cell capable of initiating tumour growth in immunodeficient mice. Nature. 2007;445:106-110. [PubMed] [DOI] |
| 2. | Eramo A, Lotti F, Sette G, Pilozzi E, Biffoni M, Di Virgilio A, Conticello C, Ruco L, Peschle C, De Maria R. Identification and expansion of the tumorigenic lung cancer stem cell population. Cell Death Differ. 2008;15:504-514. [PubMed] [DOI] |
| 3. | Yang ZF, Ngai P, Ho DW, Yu WC, Ng MN, Lau CK, Li ML, Tam KH, Lam CT, Poon RT. Identification of local and circulating cancer stem cells in human liver cancer. Hepatology. 2008;47:919-928. [PubMed] [DOI] |
| 4. | Schatton T, Murphy GF, Frank NY, Yamaura K, Waaga-Gasser AM, Gasser M, Zhan Q, Jordan S, Duncan LM, Weishaupt C. Identification of cells initiating human melanomas. Nature. 2008;451:345-349. [PubMed] [DOI] |
| 5. | Li C, Heidt DG, Dalerba P, Burant CF, Zhang L, Adsay V, Wicha M, Clarke MF, Simeone DM. Identification of pancreatic cancer stem cells. Cancer Res. 2007;67:1030-1037. [PubMed] [DOI] |
| 6. | Hatfield S, Ruohola-Baker H. microRNA and stem cell function. Cell Tissue Res. 2008;331:57-66. [PubMed] [DOI] |
| 7. | Volinia S, Calin GA, Liu CG, Ambs S, Cimmino A, Petrocca F, Visone R, Iorio M, Roldo C, Ferracin M. A microRNA expression signature of human solid tumors defines cancer gene targets. Proc Natl Acad Sci U S A. 2006;103:2257-2261. [PubMed] [DOI] |
| 8. | Yu F, Yao H, Zhu P, Zhang X, Pan Q, Gong C, Huang Y, Hu X, Su F, Lieberman J. let-7 regulates self renewal and tumorigenicity of breast cancer cells. Cell. 2007;131:1109-1123. [PubMed] [DOI] |
| 10. | Zhang B, Pan X, Anderson TA. MicroRNA: a new player in stem cells. J Cell Physiol. 2006;209:266-269. [PubMed] [DOI] |
| 11. | Du T, Zamore PD. microPrimer: the biogenesis and function of microRNA. Development. 2005;132:4645-4652. [PubMed] [DOI] |
| 12. | Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell. 2003;115:787-798. [PubMed] [DOI] |
| 13. | Eis PS, Tam W, Sun L, Chadburn A, Li Z, Gomez MF, Lund E, Dahlberg JE. Accumulation of miR-155 and BIC RNA in human B cell lymphomas. Proc Natl Acad Sci U S A. 2005;102:3627-3632. [PubMed] [DOI] |
| 14. | Lee EJ, Gusev Y, Jiang J, Nuovo GJ, Lerner MR, Frankel WL, Morgan DL, Postier RG, Brackett DJ, Schmittgen TD. Expression profiling identifies microRNA signature in pancreatic cancer. Int J Cancer. 2007;120:1046-1054. [PubMed] [DOI] |
| 15. | Johnson SM, Grosshans H, Shingara J, Byrom M, Jarvis R, Cheng A, Labourier E, Reinert KL, Brown D, Slack FJ. RAS is regulated by the let-7 microRNA family. Cell. 2005;120:635-647. [PubMed] [DOI] |
| 16. | Takamizawa J, Konishi H, Yanagisawa K, Tomida S, Osada H, Endoh H, Harano T, Yatabe Y, Nagino M, Nimura Y. Reduced expression of the let-7 microRNAs in human lung cancers in association with shortened postoperative survival. Cancer Res. 2004;64:3753-3756. [PubMed] [DOI] |
| 17. | Michael MZ, O' Connor SM, van Holst Pellekaan NG, Young GP, James RJ. Reduced accumulation of specific microRNAs in colorectal neoplasia. Mol Cancer Res. 2003;1:882-891. [PubMed] |
| 18. | Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 2004;101:11755-11760. [PubMed] [DOI] |
| 19. | Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, Shimizu M, Rattan S, Bullrich F, Negrini M. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci U S A. 2004;101:2999-3004. [PubMed] [DOI] |
| 20. | Tang F, Hajkova P, O'Carroll D, Lee C, Tarakhovsky A, Lao K, Surani MA. MicroRNAs are tightly associated with RNA-induced gene silencing complexes in vivo. Biochem Biophys Res Commun. 2008;372:24-29. [PubMed] [DOI] |
| 21. | Houbaviy HB, Murray MF, Sharp PA. Embryonic stem cell-specific MicroRNAs. Dev Cell. 2003;5:351-358. [PubMed] [DOI] |
| 22. | Suh MR, Lee Y, Kim JY, Kim SK, Moon SH, Lee JY, Cha KY, Chung HM, Yoon HS, Moon SY. Human embryonic stem cells express a unique set of microRNAs. Dev Biol. 2004;270:488-498. [PubMed] [DOI] |
| 23. | Kim HK, Lee YS, Sivaprasad U, Malhotra A, Dutta A. Muscle-specific microRNA miR-206 promotes muscle differentiation. J Cell Biol. 2006;174:677-687. [PubMed] [DOI] |
| 24. | Politz JC, Zhang F, Pederson T. MicroRNA-206 colocalizes with ribosome-rich regions in both the nucleolus and cytoplasm of rat myogenic cells. Proc Natl Acad Sci U S A. 2006;103:18957-18962. [PubMed] [DOI] |
| 25. | Georgantas RW 3rd, Hildreth R, Morisot S, Alder J, Liu CG, Heimfeld S, Calin GA, Croce CM, Civin CI. CD34+ hematopoietic stem-progenitor cell microRNA expression and function: a circuit diagram of differentiation control. Proc Natl Acad Sci U S A. 2007;104:2750-2755. [PubMed] [DOI] |