修回日期: 2010-04-24
接受日期: 2010-04-27
在线出版日期: 2010-06-28
目的: 观察阿德福韦酯(ADV)单药序贯治疗拉米夫定(LAM)耐药慢性乙型肝炎(CHB)出现再耐药患者HBV变异株的动态变化.
方法: 28例初始LAM治疗的CHB患者, 出现LAM耐药后换用ADV单药序贯治疗再次出现不充分病毒学应答或病毒学突破. 采用实时荧光定量PCR检测DNA, 分析HBV DNA动态变化; 采用直接PCR产物测序法检测耐药变异, 分析变异模式变化. 对其中2例患者系列血清进行基因克隆分析其HBV耐药株动态变化.
结果: 28例入选患者HBV DNA载量随耐药变异株的消长而波动: 基线为(7.65±1.04) Log10copies/mL, LAM疗程中最低值中位数为3.68 Log10copies/mL; 出现病毒学突破或/和耐药变异时为(6.87±1.16) Log10copies/mL, 患者换用ADV后最低值中位数为3.78 Log10copies/mL; 患者出现耐药变异模式改变或再次病毒学突破时为(6.04±0.93) Log10copies/mL. 治疗基线及两次病毒突破3个时点HBV DNA载量均数逐渐下降, 且两两之间差异均有统计学意义. 28例患者LAM治疗后24例发生rtM204I/V伴或不伴rtL180M等变异, 4例无耐药变异; 换用ADV单药序贯治疗出现病毒学突破后, 进行测序检测耐药发现: LAM耐药变异13例(包括3例多重耐药), ADV耐药变异11例(包括3例多重耐药), 无耐药变异7例. 其中11例ADV耐药患者: rtA181V变异6例、rtA181T变异3例、rtN236T变异和rtA181V+rtN236T联合变异各1例. 对2例患者血清TA克隆发现ADV耐药准种出现和LAM耐药准种消失的过程, 同时发现对ADV耐药的rtI233M变异和对替诺福韦耐药的rtA194T变异.
结论: ADV单药序贯治疗LAM耐药CHB出现再耐药患者HBV变异株动态变化存在5种形式, 且克隆分析可观察耐药株动态变化过程和发现预存的耐药准种.
引文著录: 邵幼林, 张锁才, 陈良云, 吴金华, 赵卫峰. LAM耐药后ADV序贯治疗再耐药HBV变异株的动态变化. 世界华人消化杂志 2010; 18(18): 1895-1901
Revised: April 24, 2010
Accepted: April 27, 2010
Published online: June 28, 2010
AIM: To investigate the dynamic changes of hepatitis B virus (HBV) mutants in lamivudine-resistant chronic hepatitis B (LAM-R CHB) patients who received sequential monotherapy with adefovir dipivoxil (ADV).
METHODS: Twenty-eight consecutive LAM-R CHB patients with viral breakthrough or inadequate virologic response during ADV monotherapy were studied. The dynamics of HBV mutants were analyzed over time by detecting the quantity of HBV DNA using real-time fluorescent quantitative PCR and by sequencing PCR products. Additionally, PCR-TA cloning was performed to study the dynamics of HBV quasispecies in 2 patients.
RESULTS: HBV DNA load fluctuated with the evolution of HBV mutants. Of all patients, 24 developed LAM resistance-associated substitutions during LAM therapy, and 4 had no resistance-associated mutations; 13 had LAM resistance-associated mutations, 11 had ADV resistance-associated mutations, and 7 had no resistance-associated mutations. Of 11 patients with ADV resistance-associated mutations, 6 had A181V mutation, 3 had A181T mutation, 1 had N236T mutation, and 1 had A181T/V+N236T mutation. PCR-TA cloning analysis showed that LAM-resistant viral population was gradually replaced by ADV-resistant viruses during ADV sequential monotherapy. We also detected rtI233M and rtA194T mutations that are associated with resistance to ADV and tenofovir, respectively.
CONCLUSION: Viral quasispecies dynamics in LAM-R CHB patients during ADV sequential monotherapy may manifest as various forms, such as LAM resistance-associated mutations with/without ADV resistance-associated mutations, or absence of resistance-associated mutations. Potential resistance quasispecies could be detected by PCR-TA cloning.
- Citation: Shao YL, Zhang SC, Chen LY, Wu JH, Zhao WF. Dynamics of hepatitis B virus resistance to adefovir in lamivudine-resistant patients. Shijie Huaren Xiaohua Zazhi 2010; 18(18): 1895-1901
- URL: https://www.wjgnet.com/1009-3079/full/v18/i18/1895.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i18.1895
核苷/酸类似物(nucleos/tide analogues, NAs)治疗慢性乙型肝炎(chronic hepatitis B, CHB)患者能有效改善肝功能, 促进肝组织学重建、HBeAg消失、血清学转换并有良好的耐受性[1]. 乙型肝炎病毒(hepatitis B virus, HBV)聚合酶缺乏3'-5'外切酶校对功能, 因此在HBV慢性感染者体内存在不同准种[2]. 在NAs治疗压力选择下导致原有变异准种在成为优势株或P基因RT区变异成为耐药株. 拉米夫定(Lamivudine, LAM)治疗常发生YMDD耐药变异, 阿德福韦酯(Adefovir dipivoxil, ADV)可显著降低YMDD变异患者的血清HBV DNA滴度和丙氨酸氨基转移酶(alanine amino-transferase, ALT)水平[3]. Yeon等[4]报道ADV治疗LAM耐药患者较初治患者出现ADV耐药率高. 为了解ADV治疗LAM耐药患者体内HBV准种的变化, 我们对LAM耐药后换用ADV单药序贯治疗后再次出现不充分病毒学应答(inadequate virologic response)或病毒学突破(virologic breakthough)患者耐药株动力学变化进行了初步研究.
2002-03/2008-07在我院门诊或住院治疗的慢性乙型肝炎患者. 全部符合2005年制订慢性乙型肝炎防治指南[5]的慢性乙型肝炎诊断标准. 主要入选标准包括: (1)HBsAg阳性; (2)HBV DNA阳性; (3)血清ALT持续或反复升高; (4)服用LAM 6 mo以上. 出现病毒学突破或/和耐药变异(DNA测序法)后调整治疗方案. 排除标准包括: 入选时合并肝硬化、其他慢性肝病; 血清肌酐水平大于正常上限, 慢性肾病; 合并其他严重全身性疾病; AFP明显升高, ≥100 μg/L. 所有患者血清标本, 于-70 ℃保存待测. 治疗前、疗程中每3 mo或6 mo采用荧光定量PCR法检测HBV DNA(上海申友试剂)1次; 生物化学检测采用全自动分析仪, 病毒学突破时采用PCR产物在ABI3730型DNA自动荧光测序仪测序[上游引物: 5'-CCTGCTGGTGGCT CCAGTTCAGGAACAG-3'(nt 56-83); 下游引物5'-AAGCCCCAACCAGTGGGGGTTGCGTCA-3'(nt 1 188-1 214), 引物由上海申友公司合成].
从2例ADV耐药患者的系列血清标本中抽提HBV DNA(QIAGEN GmbH公司试剂盒), 在HBV聚合酶RT区A-E基序两侧设计PCR引物: RTS1: 5'-CTAGGACCCCTGCTCGTGTT-3'(nt 179-198), RTAS1: 5'-GCAAACC CCAAAAGACC CA-3'(nt 1 001-1 019), RTS2: 5'-AAAATCCTCACAATACCAC-3'(nt 224-242), RTAS2: 5'-GACATACTTTCCAATCAATAG-3'(nt 971-991), 引物由上海申友生物技术公司合成. 以上述抽提的HBV DNA为模板, 采用套式PCR方法进行扩增. 第1轮上游和下游引物分别为TRS1和RTAS1, 第2轮PCR上游和下游引物RTS2和RTAS2, PCR扩增条件均为: 94 ℃ 30 s, 56 ℃ 60 s, 72 ℃ 45 s, 共35个循环, 反应体系为50 µL. PCR扩增采用TaKaRa公司的Premix Taq酶. PCR产物经胶回收(上海华舜生物工程有限公司试剂盒)纯化后直接测序, 同时采用PMD18-T Vector TA克隆试剂盒(TaKaRa公司)筛选含目的片段的克隆. 阳性克隆在ABI3730型DNA自动荧光测序仪上进行测序.
统计学处理 采用Chromas2.0软件对HBV DNA P基因片段核苷酸编码的氨基酸的差异和变异类型分析, 采用Clustalx1.81.msw软件对HBV基因型分析. 采用SAS V8.1统计软件分析.
共有28例患者进入本研究, 其中男25例, 女3例, 年龄18-60(平均36.67±10.54)岁. 入选时HBeAg阳性CHB 24例, HBeAg阴性CHB 4例; 疗程中另有5例出现HBeAg消失, 5例进展至肝硬化, 2例发展为肝癌. 所有患者初始服用LAM 100 mg/d, 基线ALT为39-342 U/L(中位数109.5 U/L), 疗程24-180(中位数72) wk, 均出现病毒学突破或/和耐药变异, 初始疗程结束时ALT为21-500(中位数44) U/L. 出现LAM耐药后改用ADV 10 mg/d单药序贯治疗, 疗程24-180(中位数72) wk, 再次出现病毒学突破或耐药模式改变.
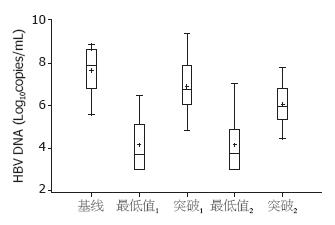
28例入选患者HBV DNA载量基线为(7.65±1.04)Log10copies/mL, 在24-180 wk LAM疗程中出现HBV DNA载量下降, 最低值为小于检测值下限至6.48Log10copies/mL (中位数3.68Log10copies/mL); 出现病毒学突破或/和耐药变异而终止初始治疗时HBV DNA载量为(6.87±1.16)Log10copies/mL, 患者在换用ADV后再次出现HBV DNA载量下降, 最低值为小于检测值下限至7.01Log10copies/mL(中位数3.78Log10copies/mL); 28例患者ADV治疗24-180 wk中再次出现病毒学突破或耐药变异模式改变, 此时HBV DNA载量为(6.04±0.93)Log10copies/mL, 患者HBV DNA动态变化见图1. 基线及两次病毒学突破后3个时点HBV DNA均数逐渐下降, 采用SNK检验结果显示3个时点两两之间差异均有统计学意义, 这可能与变异后病毒复制能力改变及部分患者发生HBeAg血清转换有关.
对28例LAM治疗时不充分病毒学应答或突破患者进行基因耐药检测发现: rtM204I变异9例, 其中2例伴随rtL180M等变异; rtM204V变异12例, 其中9例伴随rtL180M等变异; rtM204I+rtM204V变异同时存在2例, 其中1例伴随rtL180M变异. 另外, 1例患者为rtV207M+rtL180M变异, 4例未检测到耐药变异. 进入第二阶段ADV治疗28例患者再次出现不充分病毒学应答或突破, 再进行基因耐药检测: 存在LAM耐药变异13例(包括3例多重耐药), ADV耐药变异11例(包括3例多重耐药), 无耐药变异7例. 其中11例ADV耐药患者: rtA181V变异6例、rtA181T变异3例、rtN236T变异和rtA181V+rtN236T联合变异各1例. LAM耐药后换用ADV单药序贯治疗12例LAM耐药变异消失, 序贯治疗耐药准种动力学变化存在5种形式: (1)随HBV DNA逐渐下降LAM耐药株比例减少并发生完全病毒学应答; 随疗程的延长, HBV DNA突破, ADV耐药变异出现(8例); (2)换用ADV治疗后不充分病毒学应答, 在LAM耐药株的基础上出现ADV耐药变异, 即对LAM和ADV的多重耐药变异株(3例); (3)换用ADV治疗后不充分病毒学应答, LAM耐药株依然存在(10例); (4)换用ADV治疗后不充分病毒学应答, LAM耐药株消失后成为野毒株(4例); (5)LAM和ADV治疗过程中病毒学均不充分应答, 但无耐药变异(3例)(表1).
| 模式 | 患者 | 基因型 | LAM | ADV | ||
| 治疗时间(wk) | 变异形式 | 治疗时间(wk) | 变异形式 | |||
| A | 1 | C | 96 | M204V | 120 | A181V |
| 2 | C | 96 | M204I | 180 | A181V | |
| 3 | C | 48 | M204I | 24 | A181T | |
| 4 | C | 96 | M204V | 120 | A181T | |
| 5 | C | 48 | M204V+L180M | 96 | A181T | |
| 6 | C | 180 | M204V+L180M | 136 | A181V | |
| 7 | C | 24 | 204I+L180M | 72 | A181V | |
| 8 | C | 150 | M204V+L180M | 96 | N236T+A181V | |
| B | 9 | B | 48 | 无 | 96 | M204V+A181V |
| 10 | B | 48 | V207M+L180M | 96 | V207M+N236T | |
| 11 | C | 72 | M204I | 72 | M204I+A181V+T184I | |
| C | 16 | C | 96 | M204I/V+L180M | 48 | M204I/V+L180M |
| 17 | C | 72 | M204I/V | 48 | M204V+L180M | |
| 18 | C | 72 | M204V+L180M+V207L | 48 | M204V+L180M+V207L | |
| 19 | C | 48 | M204I | 48 | M204I | |
| 20 | C | 24 | M204V+L180M | 72 | M204V+L180M | |
| 21 | B | 96 | M204V+L180M | 72 | M204V+L180M | |
| 22 | B | 48 | M204V | 96 | M204V | |
| 23 | B | 72 | M204I+M250L | 24 | M204I | |
| 24 | C | 72 | M204V+L180M | 48 | M204V+L180M | |
| 25 | B | 48 | M204V+L180M | 72 | M204V+L180M | |
| D | 12 | B | 48 | M204I | 48 | 无 |
| 13 | B | 72 | M204I | 72 | 无 | |
| 14 | B | 96 | M204V+L180M | 120 | 无 | |
| 15 | C | 24 | M204I | 150 | 无 | |
| E | 26 | C | 24 | 无 | 72 | 无 |
| 27 | B | 72 | 无 | 72 | 无 | |
| 28 | B | 72 | 无 | 48 | 无 | |
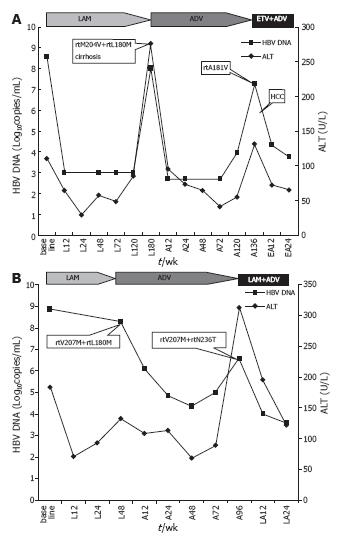
本研究28例患者ADV疗程中HBV DNA最低值12例<103 copies/mL, 16例>103 copies/mL, 是否发生病毒学完全应答与基线HBV DNA有关, 完全应答组为(7.02±1.07)Log10copies/mL, 而不充分应答组为(8.06±0.82)Log10copies/mL(t = 2.76, P = 0.011). 选取ADV疗程中病毒学完全应答(患者6)和不充分应答(患者10)各1例, 对该2例患者在LAM耐药时至ADV耐药出现各3个时间点的血清标本应用TA克隆技术分析各时间点病毒准种的构成比. 患者6在LAM和ADV治疗后均较快出现完全病毒学应答, 但LAM治疗180 wk和ADV治疗136 wk时分别发现相关耐药变异, 抗病毒治疗320 wk时发现肝脏占位, 手术病理证实为肝细胞癌. 患者10在LAM和ADV治疗过程中均发生不充分病毒学应答, LAM治疗48 wk和ADV治疗96 wk时分别发现相关耐药变异. 两患者HBV DNA、ALT、测序结果及治疗情况分别见图2, TA克隆分析该病例各时间点病毒准种的构成比分别见表2和表3, 其中患者6在ADV治疗136wk时克隆出1/13例rtA181V+rtI233M变异, 患者10在LAM治疗48 wk时克隆出1/10例rtV207M+rtA194T变异(图3).
| 时点(克隆数) | HBV DNA(Log10copies/mL) | rtM204V+rtL180M | rtM204V | rtA181V | rtA181V+rtI233M | 野毒株 |
| L180(10) | 8.31 | 80% | 20% | - | - | |
| A120(10) | 3.54 | - | - | 70.0% | - | 30% |
| A136(13) | 7.29 | - | - | 92.3% | 7.7% | - |
| 时点(克隆数) | HBV DNA(Log10copies/mL) | rtV207M | rtV207M+ rtL180M | rtV207M+ rtA194T | rtV207M+ rtN236T | rtN236T | 野毒株 |
| L48(10) | 8.29 | 60.0% | 30% | 10.0% | - | - | - |
| A72(11) | 5.01 | 36.4% | - | - | 45.4% | 9.1% | 9.1% |
| A96(10) | 6.56 | - | - | - | 60.0% | 40.0% | - |
核苷类似物治疗CHB患者能有效抑制HBV复制, 但治疗过程中可出现病毒学突破导致病情恶化, 病毒学突破常因耐药变异引起. Allen等[6]和赵卫峰等[7]都报道LAM治疗常发生rtM204V/I伴或不伴rtL180M变异. Gaillard等[8]报道rtM204V/I和rtL180M单独或联合变异病毒株的复制能力均较野毒株下降. 而ADV治疗出现耐药变异后DNA多聚酶对天然底物的亲合力下降, 使得变异株的复制能力降低[9]. 本研究发现出现LAM或ADV耐药变异后出现病毒学突破, 但变异株的病毒载量较治疗前低, 且存在统计学差异, 这可能因为: (1)耐药变异导致HBV复制能力下降; (2)部分患者出现HBeAg消失, 或血清转换; (3)抗病毒药物对变异株仍有较弱的抑制作用.
ADV可显著降低YMDD变异患者的血清HBV DNA滴度和ALT水平[3], 但Yeon等[4]报道ADV治疗LAM耐药患有较高的ADV耐药率, 1、2年耐药率分别是6.4%、25.4%. 这可能与LAM可诱导rtA181V/T变异有关. 本研究观察到ADV序贯治疗LAM耐药患者, 出现ADV耐药变异主要为rtA181V/T变异(9/11例), 这与ADV初治者以rtN236T变异为主不同. Villet等[10]报道rtA181V/T变异株的存在可使LAM敏感性下降5-11倍, 对ADV敏感性下降2-5倍, 导致LAM和ADV交叉耐药的出现. Idilman等[11]报道有限的时间里LAM耐药患者加用ADV并不能比换用ADV获得更多利益, 但Lee等[12]确认为LAM耐药患者加用ADV比换用ADV获益更多, Viganò等[13]报道20 mg ADV不能改善用LAM和10 mg ADV治疗的LAM耐药乙型肝炎患者的不完全应答. 因此, LAM和ADV耐药后应根据耐药模式选择挽救治疗方案, 但目前不同的报道存在差异, 有待更深入研究.
本研究通过测序发现ADV单药序贯治疗LAM耐药患者耐药准种动力学变化存在5种形式: (1)随HBV DNA逐渐下降LAM耐药株比例减少并发生完全病毒学应答; 随疗程的延长, HBV DNA突破, ADV耐药变异出现; (2)换用ADV治疗后不充分病毒学应答, 在LAM耐药株的基础上出现ADV耐药变异, 即对LAM和ADV的多重耐药变异株; (3)换用ADV治疗后不充分病毒学应答, LAM耐药株依然存在; (4)换用ADV治疗后不充分病毒学应答, LAM耐药株消失后成为野毒株; (5)LAM和ADV治疗过程中病毒学均不充分应答, 但无耐药变异. 从4例实施过3次测序患者的耐药株动态变化可以看出: 随治疗时间延长, LAM耐药株逐渐消失, ADV耐药株逐渐出现, 还因为耐药检测的时间不统一, 因此此种方法可能不够严谨, 其耐药动力学变化需要更严谨的大样本深入研究. 由于LAM可诱导rtA181V/T变异[14], LAM疗程中可能产生少量rtA181V/T变异的HBV准种, 而在ADV单药序贯治疗时被选择出来成为耐药优势株.
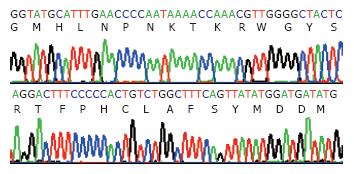
克隆分析发现LAM耐药后换用ADV序贯治疗, 随着LAM耐药株消失, 由于ADV抗病毒能力较弱, ADV疗程中病毒学突破早期野毒株占一定比率, 但随ADV治疗时间的延长, 野毒株消失, 全为ADV耐药株. 患者6在ADV治疗136 wk时克隆出1/13例rtA181V+rtI233M变异, 患者10在LAM治疗48wk时克隆出1/10例rtV207M+ rtA194T变异, 而rtI233M[15]和rtA194T[16]变异分别ADV和替诺福韦耐药, 最近有报道未治疗患者测序发现rtA194T变异[17]. 克隆出rtA194T变异准种说明耐药变异株在用药前就存在, rtA194T变异HBV对替诺福韦敏感性降低并导致变异株复制能力下降[18], 该患者目前rtA194T变异虽未成为优势株但如果后续治疗中使用了替诺福韦可能导致快速病毒学反弹. 另外, 对部分ADV治疗后出现病毒学突破的患者未能检测到ADV耐药变异, 这可能与测序敏感性较低、存在其他耐药变异或机体免疫因素参与导致耐药等因素有关, 其原因有待于进一步研究.
NAs抗HBV治疗产生耐药模式具有多样性, 出现NAs耐药后应根据耐药模式选择挽救治疗方案, 目前对NAs耐药模式及其准种变化正逐步深入, 并据此进行更科学的抗病毒管理.
杨江华, 副教授, 安徽省芜湖市皖南医学院弋矶山医院感染科
Idilman等报道有限的时间里LAM耐药患者加用ADV并不能比换用ADV获得更多利益, 但Lee等确认为LAM耐药患者加用ADV比换用ADV获益更多, Viganò等报道20 mg ADV不能改善用LAM和10 mg ADV治疗的LAM耐药乙型肝炎患者的不完全应答.
本文对28例初始LAM治疗出现LAM耐药后换用ADV单药序贯治疗再次出现不充分病毒学应答或病毒学突破的CHB患者, HBV DNA和耐药模式变化进行详细描述, 并对通过基因克隆分析了其中2例患者HBV耐药准种动态变化.
本文研究提示ADV单药序贯治疗LAM耐药患者耐药准种动力学变化存在5种形式, 提示HBV耐药的复杂性, 临床工作中应尽可能根据耐药模式进行挽救治疗.
1 不充分病毒学应答(inadequate virologic response): 指治疗24 wk, 血清中HBV DNA载量下降幅度大于2Log10copies/mL以上, 但仍≥2 000 IU/mL或≥104 copies/Ml; 2 病毒学突破(virologic break-through): 指在治疗过程中, 虽然患者对药物治疗有良好的依从性, 相隔l mo的连续两次检查, 血清HBV DNA载量比获得应答后的最低值的上升值均大于1Log10copies/mL, 病毒学突破常常提示耐药的产生.
本研究新颖性尚可, 设计合理, 内容有较好的意义.
编辑 李军亮 电编 何基才
| 1. | Dienstag JL, Goldin RD, Heathcote EJ, Hann HW, Woessner M, Stephenson SL, Gardner S, Gray DF, Schiff ER. Histological outcome during long-term lamivudine therapy. Gastroenterology. 2003;124:105-117. [PubMed] [DOI] |
| 2. | Cui XJ, Cho YK, Song HJ, Choi EK, Kim HU, Song BC. Molecular characteristics and functional analysis of full-length hepatitis B virus quasispecies from a patient with chronic hepatitis B virus infection. Virus Res. 2010;150:43-48. [PubMed] [DOI] |
| 3. | Westland CE, Yang H, Delaney WE, Wulfsohn M, Lama N, Gibbs CS, Miller MD, Fry J, Brosgart CL, Schiff ER. Activity of adefovir dipivoxil against all patterns of lamivudine-resistant hepatitis B viruses in patients. J Viral Hepat. 2005;12:67-73. [PubMed] [DOI] |
| 4. | Yeon JE, Yoo W, Hong SP, Chang YJ, Yu SK, Kim JH, Seo YS, Chung HJ, Moon MS, Kim SO. Resistance to adefovir dipivoxil in lamivudine resistant chronic hepatitis B patients treated with adefovir dipivoxil. Gut. 2006;55:1488-1495. [PubMed] |
| 6. | Allen MI, Deslauriers M, Andrews CW, Tipples GA, Walters KA, Tyrrell DL, Brown N, Condreay LD. Identification and characterization of mutations in hepatitis B virus resistant to lamivudine. Lamivudine Clinical Investigation Group. Hepatology. 1998;27:1670-1677. [PubMed] [DOI] |
| 8. | Gaillard RK, Barnard J, Lopez V, Hodges P, Bourne E, Johnson L, Allen MI, Condreay P, Miller WH, Condreay LD. Kinetic analysis of wild-type and YMDD mutant hepatitis B virus polymerases and effects of deoxyribonucleotide concentrations on polymerase activity. Antimicrob Agents Chemother. 2002;46:1005-1013. [PubMed] [DOI] |
| 9. | Yadav V, Chu CK. Molecular mechanisms of adefovir sensitivity and resistance in HBV polymerase mutants: a molecular dynamics study. Bioorg Med Chem Lett. 2004;14:4313-4317. [PubMed] [DOI] |
| 10. | Villet S, Pichoud C, Billioud G, Barraud L, Durantel S, Trépo C, Zoulim F. Impact of hepatitis B virus rtA181V/T mutants on hepatitis B treatment failure. J Hepatol. 2008;48:747-755. [PubMed] [DOI] |
| 11. | Idilman R, Kaymakoglu S, Oguz Onder F, Ahishali E, Bektas M, Cinar K, Pinarbasi B, Karayalcin S, Badur S, Cakaloglu Y. A short course of add-on adefovir dipivoxil treatment in lamivudine-resistant chronic hepatitis B patients. J Viral Hepat. 2009;16:279-285. [PubMed] [DOI] |
| 12. | Lee JM, Park JY, Kim DY, Nguyen T, Hong SP, Kim SO, Chon CY, Han KH, Ahn SH. Long-term adefovir dipivoxil monotherapy for up to 5 years in lamivudine-resistant chronic hepatitis B. Antivir Ther. 2010;15:235-241. [PubMed] [DOI] |
| 13. | Viganò M, Lampertico P, Facchetti F, Lunghi G, Colombo M. Failure of adefovir 20 mg to improve suboptimal response in lamivudine-resistant hepatitis B patients treated with adefovir 10 mg and lamivudine. J Viral Hepat. 2008;15:922-924. [PubMed] [DOI] |
| 14. | Yatsuji H, Noguchi C, Hiraga N, Mori N, Tsuge M, Imamura M, Takahashi S, Iwao E, Fujimoto Y, Ochi H. Emergence of a novel lamivudine-resistant hepatitis B virus variant with a substitution outside the YMDD motif. Antimicrob Agents Chemother. 2006;50:3867-3874. [PubMed] [DOI] |
| 15. | Schildgen O, Sirma H, Funk A, Olotu C, Wend UC, Hartmann H, Helm M, Rockstroh JK, Willems WR, Will H. Variant of hepatitis B virus with primary resistance to adefovir. N Engl J Med. 2006;354:1807-1812. [PubMed] [DOI] |
| 16. | Sheldon J, Camino N, Rodés B, Bartholomeusz A, Kuiper M, Tacke F, Núñez M, Mauss S, Lutz T, Klausen G. Selection of hepatitis B virus polymerase mutations in HIV-coinfected patients treated with tenofovir. Antivir Ther. 2005;10:727-734. [PubMed] |
| 17. | Pastor R, Habersetzer F, Fafi-Kremer S, Doffoel M, Baumert TF, Gut JP, Stoll-Keller F, Schvoerer E. Hepatitis B virus mutations potentially conferring adefovir/tenofovir resistance in treatment-naive patients. World J Gastroenterol. 2009;15:753-755. [PubMed] [DOI] |
| 18. | Amini-Bavil-Olyaee S, Herbers U, Sheldon J, Luedde T, Trautwein C, Tacke F. The rtA194T polymerase mutation impacts viral replication and susceptibility to tenofovir in hepatitis B e antigen-positive and hepatitis B e antigen-negative hepatitis B virus strains. Hepatology. 2009;49:1158-1165. [PubMed] [DOI] |