修回日期: 2010-04-30
接受日期: 2010-05-10
在线出版日期: 2010-06-08
目的: 探讨IL-10基因-1082、-819位点的单核苷酸多态性与HBV感染后疾病转归的关系.
方法: 采用TaqMan SNP基因分型和基因测序的方法检测187例健康人群、94例感染HBV后自愈者、130例慢性乙型肝炎患者、119例乙型肝炎肝硬化患者以及170例HBV相关性肝癌患者IL-10-1082、-819位点的基因型及等位基因的分布及其差异.
结果: 全部700例研究对象-1082位点的基因型分布为AA基因型占82.29%、AG占16.00%、GG占1.71%; 等位基因频率: A为90.29%, G为9.71%. -819位点的基因型分布为TT占41.43%、TC占48.14%、CC占10.43%; 等位基因频率: T为65.64%, C为34.36%. 两位点等位基因和基因型分布在各组无统计学差异. 以自愈组为对照, -1082 AG基因型和G等位基因对慢乙型肝炎和肝硬化组的风险度降低, -819 TC对肝癌组风险度降低, CC基因型和C等位基因对三个肝病组的风险度均有降低的趋势. 分析-1082/-819单倍型在各组中的分布, 以健康组和自愈组为对照, AC单倍型对肝硬化和肝癌组的风险度均降低, 且与自愈组对照时降低程度更大; 以自愈组为对照, GC单倍型对慢乙型肝炎和肝硬化组的风险度降低. 在不同临床特征的组别之间比较发现, 对于-819位点, DNA<103拷贝/mL的患者C等位基因频率显著高于DNA≥103拷贝/mL患者(P = 0.025). 而在肝癌和肝硬化组的211例不同Child-Pugh肝功能分级、277例不同AFP水平以及所有患者组的389例不同HBsAg表达之间的比较无统计学差异.
结论: IL-10-1082 AG基因型和G等位基因, -819 TC、CC基因型和C等位基因, -1082/-819 AC、GC单倍型对HBV感染后疾病进展可能有一定的保护作用, -819位点C等位基因更有利于HBV感染患者病毒的清除.
引文著录: 刘娟娟, 高英堂, 杜智, 杨斌, 经翔, 王毅军, 王凤梅, 刘彤. IL-10基因启动子区单核苷酸多态性与乙型肝炎病毒感染后疾病转归的关系. 世界华人消化杂志 2010; 18(16): 1656-1663
Revised: April 30, 2010
Accepted: May 10, 2010
Published online: June 8, 2010
AIM: To investigate the relationship between interleukin-10 (IL-10) gene promoter polymorphisms at positions -1082 and -819 and the outcome of hepatitis B virus (HBV) infection.
METHODS: TaqMan SNP genotyping and sequencing assays were employed to determine the distribution of genotypes and alleles of the IL-10 gene at positions -1082 and -819 in 187 healthy control subjects, 94 self-limited HBV patients, 130 patients with chronic hepatitis B, 119 patients with HBV-induced liver cirrhosis and 170 patients with HBV-related hepatocellular carcinoma (HCC).
RESULTS: Of the 700 subjects, 82.29% had the AA genotype, 16.00% had the AG genotype, and 1.71% had the GG genotype in the IL-10 gene promoter at position -1082. The frequencies of A and T alleles were 90.29% and 9.71%, respectively. The TT, TC and CC genotypes in the IL-10 gene promoter at position -819 accounted for 41.43%, 48.14%, and 10.43%, respectively. The frequencies of T and C alleles were 65.64% and 34.36%, respectively. There were no significant differences in genotype or allele distribution at the two positions among different groups. IL-10-1082 AG genotype and G allele decreased the risk of chronic hepatitis B and cirrhosis. IL-10-819 TC genotype was associated with a decreased risk of HCC. IL-10-819 CC genotype and C allele decreased the risk of chronic hepatitis B, cirrhosis and HCC. AC haplotype decreased the risk of cirrhosis and HCC, while GC haplotype decreased the risk of chronic hepatitis B and cirrhosis. The frequency of IL-10-819 C was significantly higher in patients with HBV DNA levels < 103 copies/mL than those with HBV DNA levels ≥ 103 copies/mL (P = 0.025). No significant differences were noted in genotypes or allele frequencies among HCC or cirrhosis subjects with different grades of Child-Pugh classification, different levels of alpha-fetoprotein or different HBsAg status.
CONCLUSION: IL-10-1082 AG genotype and G allele, IL-10-819 TC and CC genotype and C allele, as well as IL-10-1082/-819 AC and GC haplotypes may play a protective role in the disease progression after HBV infection. IL-10-819 C allele may contribute to virus elimination in HBV-infected patients.
- Citation: Liu JJ, Gao YT, Du Z, Yang B, Jing X, Wang YJ, Wang FM, Liu T. Relationship between IL-10 gene promoter polymorphisms and outcome of hepatitis B virus infection. Shijie Huaren Xiaohua Zazhi 2010; 18(16): 1656-1663
- URL: https://www.wjgnet.com/1009-3079/full/v18/i16/1656.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i16.1656
乙型肝炎病毒(hepatitis B virus, HBV)感染可引起多种临床表现, 包括病毒清除后自愈、无症状病毒携带和持续感染进展为慢性肝炎、肝硬化或原发性肝癌等严重肝病[1,2]. 近年研究表明, 宿主基因白介素-10(interleukin-10, IL-10)启动子区的单核苷酸多态性(single nucleotide polymorphism, SNP)能够影响HBV感染后的疾病进程[3,4]. 有些学者的研究发现, IL-10低表达的等位基因或单倍型(-592A, -1082/-592 AA等)不利于HBV感染后病程好转[5,6]; 有些研究则表明IL-10中、高表达等位基因、基因型或单倍型(-1082G, -819C, -592CC, -1082/-819/-592 ACC等)可以加剧HBV感染后疾病进展[3,4,7-9]; 而Li等[10]的研究则未发现IL-10基因多态性与HBV感染有任何关联. 可见目前IL-10启动子区SNP对HBV感染后疾病进展的影响尚存在争议. 因此, 本研究通过检测其在各类HBV感染者中的分布特点, 进一步探讨其与HBV感染后疾病转归的关系.
参照2005年中华医学会《慢性乙型肝炎防治指南》和2009年《原发性肝癌规范化诊治专家共识》的诊断标准, 收集天津市第三中心医院2003-12/2009-06慢性乙型肝炎患者130例、乙型肝炎肝硬化患者119例和乙型肝炎病毒相关性肝癌患者170例. 健康对照组187例和乙型肝炎自愈组94例来自天津市第三中心医院健康查体, 健康对照组HBV免疫学标志物均为阴性, 且血常规、生化指标均正常; 自愈者均无肝病史, 具有曾感染HBV证据(HBsAb、HBeAb和HBcAb三种抗体至少两种阳性, HBsAg、HBeAg和HBV-DNA均为阴性)且未经治疗而健康者. 各研究对象均抽取空腹外周静脉血3 mL, 血清用于检测HBV免疫学指标、肝功能、HBV DNA和AFP等临床指标, 血凝块用于提取白细胞DNA. 以上所有对象均为天津地区汉族人. 本研究经天津市第三中心医院伦理委员会批准, 所有患者均签署知情同意书.
1.2.1 DNA提取: 用牙签将血凝块捣碎, 吸取100 μL置于离心管中, 加入800 μL溶血试剂[0.3 mol/L蔗糖, 1 mmol/L Tris-HCl(pH8.0), 5 mmol/L MgCl2, 20 mmol/L Triton X-100], 震荡后8 000 r/min离心5 min; 吸出上清, 将沉淀捣碎并加入500 μL消化液[0.1 mol/L NaCl, 10 mmol/L Tris-HCl(pH 8.0), 30 mmol/L EDTA, 20 mmol/L SDS]和13.5 μL蛋白酶K(5.4 μmol/L), 震荡后60 ℃水浴3 h; 再加500 μL Tris饱和酚混匀, 12 000 r/min离心5 min; 吸取上层水相, 加入300 μL氯仿: 异戊醇(24:1), 12 000 r/min离心10 min; 吸取上清, 加入1/10体积NaAC(3 mol/L, pH5.2)和等体积预冷的异丙醇, 12 000 r/min离心15 min, 弃上清, 沉淀加入250 μL乙醇(700 mL/L)洗涤; 12 000 r/min离心5 min弃上清, DNA沉淀溶于50 μL TE(pH 8.0), 取2 μL进行1.5%琼脂糖凝胶电泳检测其质量, 紫外分光光度计测定DNA浓度.
1.2.2 PCR扩增: 参照TaqMan® SNP Genotyping Assays Protocol(美国应用生物系统公司), PCR扩增体系为10 µL, 含DNA模板1.5 µL、2×TaqMan Genotyping Master Mix 5 µL、40×TaqMan SNP Genotyping Assay Mix(-1082A/G: rs1800896, C_1747360_10; -819T/C: rs1800871, C_1747362_10)0.25 µL和dH2O 3.25 µL. 采用ABI PRISM® 7000 SDS进行PCR扩增, 每96孔板设置三个空白对照(NTC). 反应条件为: 95 ℃热变性10 min; 92 ℃变性15 s, 60 ℃退火1 min, 共40个循环.
1.2.3 IL-10-1082、-819基因型检测: 在上述PCR基础上, 利用ABI PRISM® 7000 SDS进行等位基因识别分析, 两个位点均设置FAM、VIC探针和样品孔、3个NTC空白对照, 点击Post-Read按钮, 读取荧光信号, 通过X, Y轴散点图分析各样本的基因型.
1.2.4 DNA测序分析: 为验证TaqMan® SNP基因分型方法的准确性, 随机挑取不同基因型和单倍型的25个样品进行测序, 对4个杂合子样品进行克隆, 每个样品再挑取5个克隆测序. 参照人IL-10序列(GenBank Accession No. NG_012088)设计引物, 正向引物: 5'-AATCCAAGACAACACTACTAAGG-3'(3 909-3 931 nt); 反向引物: 5'-AGGCAGTCACCTTAGGTCTCTGG-3'(4 556-4 534 nt). PCR扩增体系为100 µL, 包括2×PCR Master Mix 50 µL、上下游引物各2 µL、酶4 µL(1 U/µL)、DNA模板2 µL和dH2O 40 µL. PCR扩增条件: 94 ℃预变性2 min; 94 ℃变性45 s, 58 ℃退火45 s, 72 ℃延伸45 s, 共35个循环; 72 ℃延伸5 min. 扩增产物长度为648 bp, 取5 µL进行2.5%琼脂糖凝胶电泳, 剩余产物由上海博亚公司测序.
统计学处理 采用SPSS13.0软件进行统计学分析, 总体和各组样本基因型分布均经Hardy-Weinberg平衡检验. 以行×列或连续校正卡方(χ2)检验分析基因型和等位基因在不同组别之间的差异, 双侧检验P<0.05表示差异显著, P<0.01表示差异极显著.
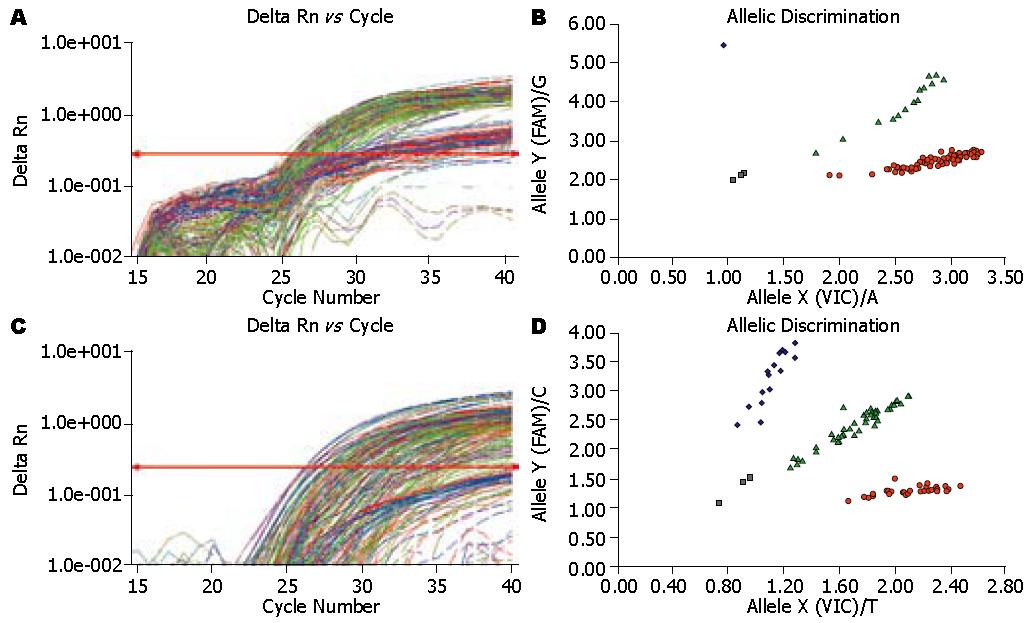
基因组DNA的IL-10- 1082、-819双探针PCR扩增曲线良好(图1A和图1C). SNP基因分型见图1B和图1D, 根据美国应用生物系统公司提供的信息, 等位基因分型图谱中X轴代表VIC探针(-1082A或-819T), Y轴代表FAM探针(-1082G或-819C), 样品集中分布在4个区域, 3个NTC空白对照在接近原点的对角线上, 靠近X轴的代表-1082AA或-819TT纯合子, 靠近Y轴的代表-1082GG或-819CC纯合子, 对角线上的一组为杂合子(-1082AG或-819TC). 参照空白对照, 各样本均能明确分析基因型.
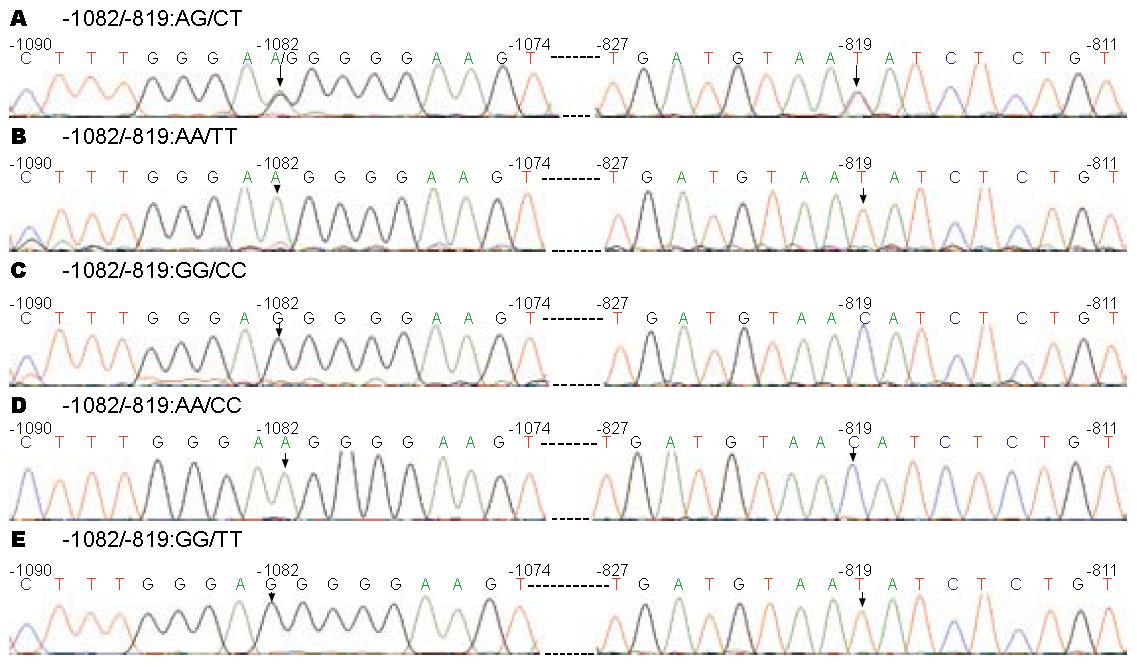
为验证TaqMan® SNP基因分型方法的准确性, 随机挑取两位点各种基因型的25个样品进行PCR产物直接测序和4个杂合子样品的20个克隆测序, 测序结果与TaqMan SNP基因分型结果完全一致(图2).
全部700例研究对象的基因型和等位基因分布: -1082位点的基因型分布为AA基因型占82.29%、AG为16.00%、GG占1.71%; 等位基因频率: A为90.29%, G为9.71%. -819位点的基因型分布为TT占41.43%、TC占48.14%、CC占10.43%; 等位基因频率: T为65.64%, C为34.36%. 各组IL-10-1082、-819基因型和等位基因分布情况见表1, 表2, 各组样本均符合Hardy-Weinberg平衡, 两位点等位基因和基因型分布在各组无统计学差异. 以健康人群为对照计算其他各组的OR值, 未发现风险度的明显升高或降低, 当以自愈组为对照计算其他各组的OR值时, -1082 AG基因型和G等位基因对慢乙型肝炎和肝硬化组的风险度降低, -819 TC对肝癌组风险度降低, CC基因型和C等位基因对三个肝病组的风险度均有降低的趋势. 如表3所示, 分析-1082/-819单倍型在各组中的分布, 以健康和自愈组为对照, AC单倍型对肝硬化和肝癌组的风险度均降低, 且与自愈组对照时降低程度更大; 以自愈组为对照, GC单倍型对慢乙型肝炎和肝硬化组的风险度降低.
| 基因型/等位基因 | 分组 | ||||
| 健康对照组n(%) | 自愈组n(%) | 慢乙型肝炎组n(%) | 肝硬化组n(%) | 肝癌组n(%) | |
| IL-10-1082 | |||||
| AA | 160(85.56) | 73(77.66) | 112(86.15) | 100(84.03) | 131(77.06) |
| AG | 24(12.83) | 20(21.28) | 16(12.31) | 17(14.29) | 35(20.59) |
| GG | 3(1.61) | 1(1.06) | 2(1.54) | 2(1.68) | 4(2.35) |
| A | 344(91.98) | 166(88.30) | 240(92.31) | 217(91.18) | 297(87.35) |
| G | 30(8.02) | 22(11.70) | 20(7.69) | 21(8.82) | 43(12.65) |
| 与健康对照比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | OR(95%CI) | |
| AA | 1.0(-) | 1.0(-) | 1.0(-) | 1.0(-) | |
| AG | 1.826(0.949-3.516) | 0.952(0.484-1.874) | 1.133(0.580-2.214) | 1.781(1.009-3.145) | |
| GG | 0.731(0.075-7.143) | 0.952(0.157-5.793) | 1.067(0.175-6.495) | 1.628(0.358-7.406) | |
| A | 1.0(-) | 1.0(-) | 1.0(-) | 1.0(-) | |
| G | 1.520(0.850-2.716) | 0.956(0.530-1.723) | 1.110(0.619-1.988) | 1.660(1.016-2.714) | |
| 与自愈组比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | ||
| AA | 1.0(-) | 1.0(-) | 1.0(-) | ||
| AG | 0.521(0.254-1.072) | 0.621(0.304-1.267) | 0.975(0.525-1.812) | ||
| GG | 1.304(0.116-14.638) | 1.460(0.130-16.408) | 2.229(0.245-20.317) | ||
| A | 1.0(-) | 1.0(-) | 1.0(-) | ||
| G | 0.629(0.333-1.189) | 0.730(0.388-1.373) | 1.092(0.632-1.889) | ||
| 基因型/等位基因 | 分组 | ||||
| 健康对照组n(%) | 自愈组n(%) | 慢乙型肝炎组n(%) | 肝硬化组n(%) | 肝癌组n(%) | |
| IL-10-819 | |||||
| TT | 75(40.11) | 32(34.04) | 51(39.23) | 53(44.54) | 79(46.47) |
| TC | 92(49.20) | 45(47.87) | 68(52.31) | 59(49.58) | 73(42.94) |
| CC | 20(10.69) | 17(18.09) | 11(8.46) | 7(5.88) | 18(10.59) |
| T | 242(64.71) | 109(57.98) | 170(65.38) | 165(69.33) | 231(67.94) |
| C | 132(35.29) | 79(42.02) | 90(34.62) | 73(30.67) | 109(32.06) |
| 与健康对照比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | OR(95%CI) | |
| TT | 1.0(-) | 1.0(-) | 1.0(-) | 1.0(-) | |
| TC | 1.146(0.664-1.980) | 1.087(0.677-1.746) | 0.908(0.561-1.467) | 0.753(0.485-1.170) | |
| CC | 1.992(0.925-4.293) | 0.809(0.357-1.831) | 0.495(0.195-1.255) | 0.854(0.420-1.739) | |
| T | 1.0(-) | 1.0(-) | 1.0(-) | 1.0(-) | |
| C | 1.329(0.928-1.903) | 0.971(0.696-1.353) | 0.811(0.573-1.148) | 0.865(0.634-1.181) | |
| 与自愈组比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | ||
| TT | 1.0(-) | 1.0(-) | 1.0(-) | ||
| TC | 0.948(0.530-1.695) | 0.792(0.441-1.422) | 0.657(0.378-1.143) | ||
| CC | 0.406(0.169-0.977) | 0.249(0.093-0.665) | 0.429(0.197-0.935) | ||
| T | 1.0(-) | 1.0(-) | 1.0(-) | ||
| C | 0.730(0.496-1.075) | 0.610(0.409-0.910) | 0.651(0.450-0.941) | ||
| 单倍型 | 分组 | ||||
| 健康对照组n(%) | 自愈组n(%) | 慢乙型肝炎组n(%) | 肝硬化组n(%) | 肝癌组n(%) | |
| -1082/-819 | |||||
| AT | 242(64.71) | 109(57.98) | 169(65.00) | 166(69.75) | 231(67.94) |
| AC | 102(27.27) | 57(30.32) | 71(27.31) | 51(21.43) | 66(19.41) |
| GC | 30(8.02) | 22(11.70) | 19(7.31) | 21(8.82) | 43(12.65) |
| GT | 0(0.00) | 0(0.00) | 1(0.38) | 0(0.00) | 0(0.00) |
| 与健康对照比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | OR(95%CI) | |
| AT | 1.0(-) | 1.0(-) | 1.0(-) | 1.0(-) | |
| AC | 1.241(0.836-1.842) | 0.997(0.695-1.430) | 0.729(0.494-1.076) | 0.678(0.474-0.970) | |
| GC | 1.628(0.898-2.951) | 0.907(0.494-1.665) | 1.020(0.565-1.844) | 1.502(0.911-2.475) | |
| 与自愈组比较 | OR(95%CI) | OR(95%CI) | OR(95%CI) | ||
| AT | 1.0(-) | 1.0(-) | 1.0(-) | ||
| AC | 0.803(0.526-1.227) | 0.588(0.375-0.920) | 0.546(0.359-0.833) | ||
| GC | 0.557(0.288-1.077) | 0.627(0.329-1.195) | 0.922(0.526-1.618) | ||
三种基因型在男/女性别中的分布: -1082位点AA为81.46%/83.20%、AG为16.44%/15.23%、GG为1.80%/1.57%; -819位点TT为44.14%/36.72%、TC为45.05%/53.91%、CC为10.81%/9.37%; 均无统计学差异. 如表4, 表5所示, 在已知HBV DNA载量的379例肝病患者中, IL-10-819位点等位基因分布在DNA<103拷贝/mL/≥103拷贝/mL患者中具有统计学差异, 前者C等位基因频率显著高于后者(P = 0.025). 另外, 两个位点的基因型和等位基因在肝癌和肝硬化组的211例不同Child-Pugh肝功能分级、277例不同AFP水平以及所有患者组的389例不同HBsAg表达之间的比较无统计学差异.
| 按临床特征分组 | AA | AG | GG | A | G |
| DNA(拷贝/mL) | |||||
| <103 | 158(80.20) | 33(16.75) | 6(3.05) | 349(88.58) | 45(11.42) |
| ≥103 | 152(83.52) | 28(15.38) | 2(1.10) | 332(91.21) | 32(8.79) |
| OR(95%CI) | 1.0(-) | 0.882(0.509-1.531) | 0.346(0.069-1.743) | 1.0(-) | 0.748(0.464-1.205) |
| P值 | 0.363 | 0.231 | |||
| Child-Pugh分级 | |||||
| A | 65(74.71) | 19(21.84) | 3(3.45) | 149(85.64) | 25(37.36) |
| B+C | 100(80.65) | 22(17.74) | 2(1.61) | 222(89.52) | 26(10.48) |
| OR(95%CI) | 1.0(-) | 0.753(0.378-1.499) | 0.433(0.070-2.664) | 1.0(-) | 0.698(0.388-1.255) |
| P值 | 0.502 | 0.228 | |||
| AFP(ng/mL) | |||||
| <400 | 164(78.10) | 42(20.00) | 4(1.90) | 370(88.10) | 50(11.90) |
| >400 | 55(82.09) | 10(14.93) | 2(2.98) | 120(89.55) | 14(10.45) |
| OR(95%CI) | 1.0(-) | 0.710(0.334-1.509) | 1.491(0.266-8.365) | 1.0(-) | 0.863(0.461-1.617) |
| P值 | 0.58 | 0.646 | |||
| HBsAg | |||||
| (-) | 28(77.78) | 8(22.22) | 0(0) | 64(88.89) | 8(11.11) |
| (+) | 289(81.87) | 56(15.86) | 8(2.27) | 634(89.80) | 72(10.20) |
| OR(95%CI) | 1.0(-) | 0.678(0.294-1.565) | 无 | 1.0(-) | 0.909(0.419-1.971) |
| P值 | 0.308 | 0.808 |
| 按临床特征分组 | TT | TC | CC | T | C |
| DNA(拷贝/mL) | |||||
| <103 | 79(40.10) | 98(49.75) | 20(10.15) | 256(64.97) | 138(35.03) |
| ≥103 | 92(50.55) | 80(43.96) | 10(5.49) | 264(72.53) | 100(27.47) |
| OR(95%CI) | 1.0(-) | 0.701(0.460-1.068) | 0.429(0.190-0.971) | 1.0(-) | 0.703(0.516-0.958) |
| P值 | 0.062 | 0.025 | |||
| Child-Pugh分级 | |||||
| A | 41(47.13) | 35(40.23) | 11(12.64) | 117(67.24) | 57(32.76) |
| B+C | 61(49.19) | 56(45.16) | 7(5.65) | 178(71.77) | 70(28.23) |
| OR(95%CI) | 1.0(-) | 1.075(0.603-1.918) | 0.428(0.153-1.194) | 1.0(-) | 0.807(0.530-1.229) |
| P值 | 0.195 | 0.318 | |||
| AFP(ng/mL) | |||||
| <400 | 92(43.81) | 99(47.14) | 19(9.05) | 283(67.38) | 137(32.62) |
| >400 | 33(49.25) | 29(43.28) | 5(7.47) | 95(70.90) | 39(29.10) |
| OR(95%CI) | 1.0(-) | 0.817(0.460-1.450) | 0.734(0.254-2.123) | 1.0(-) | 0.848(0.554-1.297) |
| P值 | 0.724 | 0.447 | |||
| HBsAg | |||||
| (-) | 12(33.33) | 22(61.11) | 2(5.56) | 46(63.89) | 26(36.11) |
| (+) | 156(44.19) | 164(46.46) | 33(9.35) | 476(47.42) | 230(32.58) |
| OR(95%CI) | 1.0(-) | 0.573(0.274-1.198) | 1.269(0.271-5.940) | 1.0(-) | 0.855(0.515-1.418) |
| P值 | 0.232 | 0.543 |
现有资料表明, 由于相关遗传因素、环境因素和生活习惯等选择压力, 导致IL-10的等位基因和基因型分布具有较强的地域和人种特征[11]. 以-1082位点为例, 本文分析中国天津市187例健康汉族人的A等位基因频率为91.98%, AA、AG和GG基因型频率分别为85.56%、12.83%和1.61%; 同样, 全部700例汉族人的等位基因和基因型分布与此相近(无统计学差异), IL-10-1082 GG基因型频率与其他汉族人[8,10]、韩国人[3]、日本人[9]相似(0%-2%), 而明显低于美洲人群(约10%)[4]和欧洲人群(10%-25%)[12,13]. 启动子多态性与IL-10的表达水平密切相关, -1082G、-819C、-592C相对于-1082A、-819T、-592A为高表达型等位基因[14], 而高表达的IL-10可能与自限性HBV感染呈正相关[15]. 以上因素可能会导致亚洲人群IL-10的平均水平低于欧美人群, 成为中国患肝病者比欧美国家明显较多的原因之一.
人体感染HBV后病程如何演化是宿主和病毒等多种因素共同作用的结果, IL-10作为影响HBV感染的细胞因子之一, 越来越得到重视. 本文研究结果显示, IL-10-1082、-819基因型、等位基因频率在健康对照组、自愈组、慢乙型肝炎、肝硬化和肝癌组之间无统计学差异, 与Li等[10]的研究结果相似, 其通过检测IL-10-1082、-819、-592三个位点的多态性在HBV感染后自愈组、无症状携带和慢乙型肝炎组中的分布, 未发现任何差异. 考虑到并不清楚健康人将来若感染HBV会如何进展, 因此, 以感染HBV后自愈者作为对照组分析各肝病组可能更合理, 更能准确说明在感染HBV这一共同环境因子时不同基因型对肝病进展的风险. 本研究以自愈组为对照, 发现IL-10-1082/-819 AC单倍型对肝硬化和肝癌组的风险度降低, GC单倍型对慢乙型肝炎组和肝硬化组的风险度降低, 说明中、高水平的IL-10对持续性肝病的发生有一定的保护作用. AC、GC单倍型可以导致IL-10中、高表达[16], 在肝脏通过抑制胶原基因的转录, 发挥抗纤维化的作用[17]; 而且, IL-10作为一种重要的抗炎因子, 可以通过减少肝组织局部中性粒细胞的浸润和活化而降低中性粒细胞与肝窦内皮细胞的黏附以减轻肝细胞的损伤, 还可以降低局部炎性细胞因子的活性, 负向调节细胞因子的表达, 加强对肝细胞的保护[18]. 因此, 从IL-10的作用机制分析也支持我们的研究结果.
进一步研究IL-10基因多态性与临床特征的关系, 发现不同DNA拷贝数的肝病患者IL-10-819 C等位基因频率在DNA<103拷贝/mL组明显高于DNA≥103拷贝/mL组, 说明中、高水平的IL-10可能有利于HBV清除. 与本结果相似的是, Cheong等[5]在研究-592位点的多态性时发现, -592CC基因型(与-819C紧密连锁)在HBV清除组高于病毒持续组. 另外也有文献报道高水平IL-10的产生在HBV感染时可能会增强病毒复制[8]. 对于此方面的研究, 我们正在收集更多各类肝病患者进行IL-10多态性分析并检测相应标本血清中IL-10的水平.
总之, IL-10-1082、-819多态性显示在天津市汉族人群中具有较多的IL-10低表达的基因型(-1082AA), 而高表达基因型(-1082GG)较少, 与其他报道的亚洲人群分布特征相似而明显不同于美洲和欧洲地区人群. IL-10中、高表达的等位基因、基因型或单倍型(-1082G、-819C等位基因, -1082AG、-819TC、CC基因型, -1082/-819AC、GC单倍型)对HBV感染后疾病进展有一定的保护作用; 同时, 通过分析其多态性在不同临床特征组别中的分布特点, 发现-819C等位基因更有利于HBV清除. 为进一步验证其准确性, 还需要多中心、大样本的临床观察和血清IL-10含量测定等深入研究.
HBV感染是肝病发生的最主要原因之一, 部分感染者最终发展为慢性肝炎、肝硬化或原发性肝癌. 随着人类SNP研究工作的进展, 基因多态性的研究有望为筛选HBV感染高危人群以及肝病的防治工作提供可靠依据.
任浩, 副教授, 中国人民解放军第二军医大学微生物学教研室
SNP与HBV感染研究一直是国内外学者研究的热点和重点, SNP分布具有较强的地域和种族特征, 因此对二者进行研究可能会为HBV感染后疾病发展的机制提供一定线索.
Turner等通过对IL-10启动子SNP与血清中IL-10表达水平的关系进行分析表明, IL-10-1082G、-819C、-592C相对于-1082A、-819T、-592A为高表达型等位基因.
IL-10中、高表达的等位基因、基因型或单倍型对HBV感染后疾病进展有一定的保护作用; IL-10-819C等位基因更有利于HBV清除.
分析IL-10基因多态性对天津地区汉族人感染HBV后疾病进展的影响, 有助于对本地区肝病发生的病因学进行深入研究, 在筛选HBV易感人群并采取有效的保护和干预措施等方面都具有潜在的应用前景.
本研究科学性较好, 说服力强, 具有一定的临床意义.
编辑: 李军亮 电编: 何基才
| 1. | Rizzetto M, Ciancio A. Chronic HBV-related liver disease. Mol Aspects Med. 2008;29:72-84. [PubMed] [DOI] |
| 2. | Shi YH, Shi CH. Molecular characteristics and stages of chronic hepatitis B virus infection. World J Gastroenterol. 2009;15:3099-3105. [PubMed] [DOI] |
| 3. | Shin HD, Park BL, Kim LH, Jung JH, Kim JY, Yoon JH, Kim YJ, Lee HS. Interleukin 10 haplotype associated with increased risk of hepatocellular carcinoma. Hum Mol Genet. 2003;12:901-906. [PubMed] [DOI] |
| 4. | Truelove AL, Oleksyk TK, Shrestha S, Thio CL, Goedert JJ, Donfield SM, Kirk GD, Thomas DL, O'Brien SJ, Smith MW. Evaluation of IL10, IL19 and IL20 gene polymorphisms and chronic hepatitis B infection outcome. Int J Immunogenet. 2008;35:255-264. [PubMed] [DOI] |
| 5. | Cheong JY, Cho SW, Hwang IL, Yoon SK, Lee JH, Park CS, Lee JE, Hahm KB, Kim JH. Association between chronic hepatitis B virus infection and interleukin-10, tumor necrosis factor-alpha gene promoter polymorphisms. J Gastroenterol Hepatol. 2006;21:1163-1169. [PubMed] [DOI] |
| 6. | Chan HL, Tse AM, Chim AM, Wong VW, Choi PC, Yu J, Zhang M, Sung JJ. Association of cytokine gene polymorphisms and liver fibrosis in chronic hepatitis B. J Gastroenterol Hepatol. 2008;23:783-789. [PubMed] [DOI] |
| 7. | Tseng LH, Lin MT, Shau WY, Lin WC, Chang FY, Chien KL, Hansen JA, Chen DS, Chen PJ. Correlation of interleukin-10 gene haplotype with hepatocellular carcinoma in Taiwan. Tissue Antigens. 2006;67:127-133. [PubMed] [DOI] |
| 8. | Peng XM, Huang YS, Ma HH, Gu L, Xie QF, Gao ZL. Interleukin-10 promoter polymorphisms are associated with the mode and sequel of HBeAg seroconversion in patients with chronic hepatitis B virus infection. Liver Int. 2006;26:326-333. [PubMed] [DOI] |
| 9. | Miyazoe S, Hamasaki K, Nakata K, Kajiya Y, Kitajima K, Nakao K, Daikoku M, Yatsuhashi H, Koga M, Yano M. Influence of interleukin-10 gene promoter polymorphisms on disease progression in patients chronically infected with hepatitis B virus. Am J Gastroenterol. 2002;97:2086-2092. [PubMed] [DOI] |
| 10. | Li C, Zhi-Xin C, Li-Juan Z, Chen P, Xiao-Zhong W. The association between cytokine gene polymorphisms and the outcomes of chronic HBV infection. Hepatol Res. 2006;36:158-166. [PubMed] [DOI] |
| 11. | Le Souëf PN, Candelaria P, Goldblatt J. Evolution and respiratory genetics. Eur Respir J. 2006;28:1258-1263. [PubMed] [DOI] |
| 12. | Trajkov D, Arsov T, Petlichkovski A, Strezova A, Efinska-Mladenovska O, Spiroski M. Cytokine gene polymorphisms in population of ethnic Macedonians. Croat Med J. 2005;46:685-692. [PubMed] |
| 13. | Forte GI, Scola L, Misiano G, Milano S, Mansueto P, Vitale G, Bellanca F, Sanacore M, Vaccarino L, Rini GB. Relevance of gamma interferon, tumor necrosis factor alpha, and interleukin-10 gene polymorphisms to susceptibility to Mediterranean spotted fever. Clin Vaccine Immunol. 2009;16:811-815. [PubMed] [DOI] |
| 14. | Turner DM, Williams DM, Sankaran D, Lazarus M, Sinnott PJ, Hutchinson IV. An investigation of polymorphism in the interleukin-10 gene promoter. Eur J Immunogenet. 1997;24:1-8. [PubMed] [DOI] |
| 15. | Thursz M. Genetic susceptibility in chronic viral hepatitis. Antiviral Res. 2001;52:113-116. [PubMed] [DOI] |
| 16. | Edwards-Smith CJ, Jonsson JR, Purdie DM, Bansal A, Shorthouse C, Powell EE. Interleukin-10 promoter polymorphism predicts initial response of chronic hepatitis C to interferon alfa. Hepatology. 1999;30:526-530. [PubMed] [DOI] |
| 17. | Thompson K, Maltby J, Fallowfield J, McAulay M, Millward-Sadler H, Sheron N. Interleukin-10 expression and function in experimental murine liver inflammation and fibrosis. Hepatology. 1998;28:1597-1606. [PubMed] [DOI] |
| 18. | Ben-Ari Z, Mor E, Papo O, Kfir B, Sulkes J, Tambur AR, Tur-Kaspa R, Klein T. Cytokine gene polymorphisms in patients infected with hepatitis B virus. Am J Gastroenterol. 2003;98:144-150. [PubMed] [DOI] |