修回日期: 2010-04-24
接受日期: 2010-04-27
在线出版日期: 2010-05-18
microRNAs(miRNAs)是一种长度为19-24个核苷酸的单链非编码RNAs, 他们通过下调基因表达广泛参与各种重要的生命进程, 如细胞凋亡、分化、增殖及个体发育等. 最近研究表明, miRNAs可调控许多参与大肠癌(colorectal cancer, CRC)发生发展的癌基因和抑癌基因通路, 如Wnt/β-连环蛋白, KRAS, 磷脂酰肌醇(3)-激酶(PI3-K)和P53信号通路等. 此外, 单核苷酸多态性(SNPs)对CRC中miRNAs表达的影响及miRNAs在CRC表观遗传学改变中的作用也日益受到人们的关注. 本文将就miRNAs调控网络在CRC发生发展中的作用作一综述.
引文著录: 杨建军, 马延磊, 秦环龙. microRNAs调控网络在大肠癌发病机制中的研究进展. 世界华人消化杂志 2010; 18(14): 1478-1484
Revised: April 24, 2010
Accepted: April 27, 2010
Published online: May 18, 2010
MicroRNAs (miRNAs) are single-stranded non-coding RNAs, typically 19-24 nucleotides in length. By down-regulating gene expression, they widely participate in a variety of important life processes, such as apoptosis, differentiation, proliferation and development. Recent studies have shown that miRNAs can control many oncogene and tumor suppressor pathways that are involved in the development and progression of colorectal cancer (CRC), such as the Wnt/β-catenin, K-ras, phosphatidylinositol-3-kinase (PI3-K), and P53 signaling pathways. In addition, the research on the effects of single nucleotide polymorphisms (SNPs) on miRNA expression and the epigenetic regulation of microRNAs in CRC has attracted much attention from researchers. This paper will review the role of microRNA regulatory network in the pathogenesis of CRC.
- Citation: Yang JJ, Ma YL, Qin HL. Advances in understanding the role of microRNA regulatory network in the pathogenesis of colorectal cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(14): 1478-1484
- URL: https://www.wjgnet.com/1009-3079/full/v18/i14/1478.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i14.1478
大肠癌(colorectal cancer, CRC)是临床最常见的恶性肿瘤之一, 在西方发达国家其发病率处于第3位[1], 而在我国其发病率呈上升态势, 排在恶性肿瘤和致死因素的第4位[2]. CRC的发生是一个多因素、多阶段和多基因改变协同作用的过程, 癌基因和抑癌基因的表达失调是CRC发生的分子基础. 近年来研究认为miRNAs通过多种信号通路广泛参与恶性肿瘤(包括CRC)的发生和发展, 进一步明确miRNAs在CRC发病机制中的作用有助于提高CRC的诊治水平.
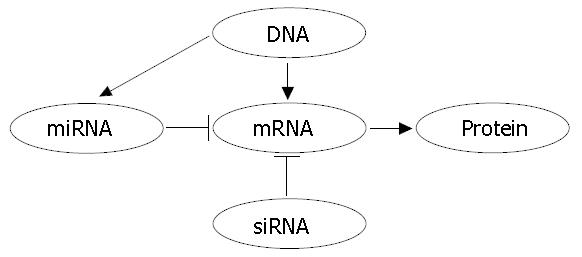
miRNAs源于细胞核内, 其基因数占人类基因总数的2%-5%, 表达分布于整个基因组. miRNAs常簇生于蛋白编码基因的内含子中. 首先, RNA聚合酶Ⅱ依赖的转录产生较长的初级转录物(pri-miRNAs), 随后核糖核酸酶Drosha对pri-miRNAs进行加工处理并形成更短的发夹结构, 即前体miRNAs(pre-miRNAs). Pre-miRNAs被转运至细胞质中接受核酸内切酶Dicer的进一步深加工, 形成一个成熟的双链miRNAs, 随后其中一条单链被整合入RNA诱导沉默复合体中(RNA induced silencing complex, RISC), 并通过部分序列互补与靶点mRNAs的3'端非翻译区(3'UTR) 相互作用, 从而抑制翻译过程或促使mRNA降解[3]. 其作用方式在某种程度上类似于短干扰RNA(siRNAs)(图1). 因此, 成熟miRNAs的最终效应是通过阻止靶点蛋白的生成而下调其靶基因表达.
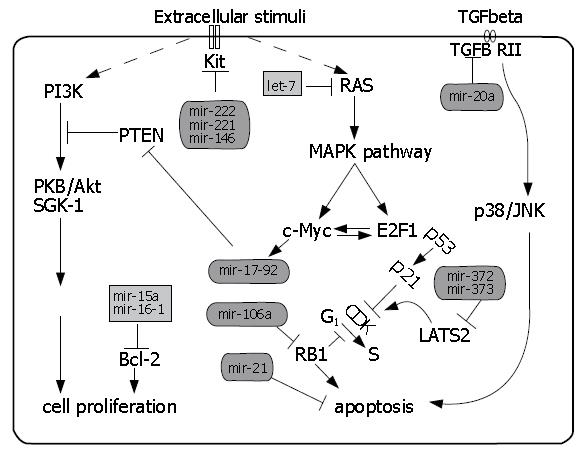
当miRNAs的mRNAs靶点受抑癌基因或癌基因编码时, miRNAs的表达失调可影响肿瘤发生. 在CRC发病机制中常存在特异miRNAs的过表达和表达沉默. 通常miRNAs编码基因的转录激活或扩增可致成熟miRNAs的表达上调, 而特定染色体区的缺失或miRNAs生物合成缺陷可导致其表达沉默或下调[4]. 利用生物信息学工具(如miRBase targets, Targetscan, PicTar, microrna.org, TargetRank, DIANAMicroT, rna22, TarBase等软件[5]), 人们已经预测了上千种miRNAs的靶基因. 值得关注的是主要的miRNAs靶点均为转录因子或激酶[6]. 许多研究者试图明确miRNAs基因靶点与其信号通路在癌症发生发展中的关系, 其中部分通路在CRC发展中的作用已得到证实[7](图2).
目前常用两种方法研究miRNAs和CRC之间的关系, 即功能性研究和表达谱研究. 功能性研究方面, miRNAs可调控许多参与CRC发病机制的癌基因和抑癌基因通路. 参与CRC主要信号通路的蛋白(如Wnt/β-连环蛋白和磷脂酰肌醇(3)激酶(PI3-K)通路的成员, KRAS, P53, 胰岛素样生长因子(insulin-like growth factor, IGF), 转录因子E2F家族和环氧化酶2(COX-2)等[8]的表达水平均有改变, 且与miRNAs的调控相关联. 针对这些miRNAs机制的研究有助于进一步明确CRC的发病机制, 从而最终鉴定出新的治疗靶点[9,10]. 表达谱研究方面, 诸多研究证实, miRNAs存在于CRC肿瘤组织和血液中, 并在CRC的发病机制中起重要作用. 这为如何提高CRC的早期筛查, 诊断和治疗效果及准确判断预后提供了新的研究方向. 本文将对miRNAs在CRC功能性研究方面作重点阐述.
Wnt/β-连环蛋白在早期CRC发生发展中起核心作用. 结肠多发性腺瘤样息肉病(adenomatous polyposis coli, APC)基因失活是CRC发生的主要起始事件之一, 他存在于超过60%的大肠腺瘤和CRC中并通过游离的β-连环蛋白刺激Wnt通路的活性[8]. 最近, Nagel等[11]研究发现miRNAs可能是CRC中一种调控APC发病的新机制. miR-135a和miR-135b在大肠腺瘤和CRC中表达上调, 体外实验进一步证实他们不仅可减少APC转录产物的翻译, 还与APC低表达水平相关联. 这提示miR-135家族的改变可能是CRC分子发病机制的早期事件.
EGFR信号通路存在于大量实体肿瘤的发生发展中, 因此是一个有希望的抗癌治疗通路. 当EGFR受刺激后, KRAS信号通路可导致许多信号转导分子活化并引发下游效应因子的级联反应. 这些级联反应介导了肿瘤的生长、生存、血管发生和转移等[12]. 已有研究发现癌基因KRAS是let-7 miRNAs家族的直接靶点[13]. 低表达let-7 miRNAs的结肠癌细胞系DLD-1中转染let-7a-1前体后, 可显著抑制DLD-1的生长; 同时KRAS蛋白表达水平也明显降低, 而其mRNA表达则无改变[14]. Chen等[15]发现在体外经miR-143前体处理后的KRAS表达水平显著降低. miR-143通过抑制KRAS的表达可阻断促分裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)的磷酸化. 另外一个与CRC中KRAS调控相关的miRNA是miR-18a; 后者在结肠癌细胞系HT-29中可直接调节KRAS的表达水平[16].
EGFR下游的另一个重要信号通路是PI3-K通路. 现已证实p85β调节亚基(参与稳定传导PI3-K信号)是miR-126的直接靶点. 在CRC细胞中, 由miR-126介导的p85β调节亚基表达水平与磷酸化AKT表达水平同时下调, 提示miR-126可间接介导PI3-K信号通路的受损. 在一组配对的结肠癌与癌旁正常组织标本中, miR-126在肿瘤组织中表达下调, 而p85β蛋白表达上调[17]. miR-21在肝细胞癌中可显著抑制抑癌基因PTEN(PI3-K通路中的另一个重要调节成分)的表达[18]; 而miR-21是CRC中最常见的表达上调的miRNAs[19-21]. 据此推测, miR-21也可通过参与抑制PTEN的表达来促进PI3-K信号通路的传导, 从而与CRC发生发展相关联.
抑癌基因P53突变存在于50%-75%的CRC和其他一些肿瘤中. P53通过诱导细胞周期检测点、细胞凋亡或衰老, 产生对DNA损伤或有丝分裂基因失调的反应[8]. P53既是转录激活因子, 也可直接或间接抑制特异基因的表达[22], 其发生机制尚不清楚, 推测可能与其转录和转录后抑制有关. 已有miRNAs调控网络研究表明, 通过调控miRNAs表达水平, P53可在转录后水平抑制其靶基因表达, 即保守的miRNAs是P53的直接转录靶点[23]. 野生型P53+/+和突变型P53-/-结肠癌细胞系HCT-116经DNA损伤因子处理后, 发现一些miRNAs只在野生型HCT-116中被诱导表达, 提示在CRC中存在一种由P53介导的miRNAs表达模式. Chang等[24]研究发现通过P53依赖和非依赖机制, miR-34a的表达可诱导细胞凋亡, 提示miR-34a可调控由P53起始的基因表达程序. 在包含P53等位基因[25]与P53+/+和P53-/-小鼠胚胎成纤维细胞[26]的肿瘤细胞模型中, P53与miR-34a相关性研究证实了上述结论, 且miR-34a参与诱导基因表达重排. 在miR-34家族的下调靶点中, 与P53相关的靶点如CDK4/6, 细胞周期蛋白E2(cyclin E2), E2F5, BIRC3和Bcl-2等变化最为显著[24-26]. Yamakuchi等[27]研究证实SIRT1[一种高度保守的基因和凋亡负性调节因子, 可编码NAD(+)依赖的脱乙酰酶]是miR-34a的另一个靶点. miR-34a对SIRT1的抑制仅在野生型结肠癌细胞系中诱导细胞凋亡, 因此认为在P53和miRNAs之间存在一种正反馈环.
胰岛素受体底物1(insulin receptor substrate-1, IRS-1)在细胞生长和细胞增殖中扮演重要作用. 当IRS-1被1型胰岛素样生长因子受体(type 1 insulin-like growth factor receptor, IGF-IR)激活后, 可发出一个明确的促有丝分裂, 抗凋亡和抗分化信号. IRS-1的表达水平在肿瘤中常增高, 而在分化细胞中降低甚至缺失[28]. 已有研究发现miR-145可作为抑癌基因起作用, 当他靶向作用于IRS-1的3'UTR时, 可显著抑制结肠癌细胞系的生长[29]. 现已证实IGF-IR是miR-145的一个直接靶点. 缺乏3'UTR的IRS-1不再被miR-145下调, 并可解除miR-145对结肠癌细胞系的生长抑制. 消除IGF-IR的3'UTR可导致对miR-145的耐受, 即miR-145既不能下调IGF-IR的表达, 也不能恢复miR-145对结肠癌细胞系的生长抑制. 这些结果提示miR-145在肿瘤发生的IGF信号通路中起重要作用[30].
在一些肿瘤中, 编码miR-17-92簇miRNAs的基因组位点发生扩增. miR-17-92簇可能与转录因子E2F家族(调节细胞周期和细胞凋亡)的功能密切相关. 最近已证实E2F1, E2F2和E2F3等活化型E2Fs是miR-17-92簇的作用靶点[31]. miR-17-92簇的致瘤活性可参与细胞的非自主功能包括诱导实体肿瘤内的血管发生等. Dews等[32]利用小鼠结肠癌模型证实miR-17-92簇可部分激活c-Myc的血管生成活性. c-Myc可显著诱导这些miRNAs负性调节抗血管生成因子凝血酶敏感蛋白1(anti-angiogenic factors thrombospondin-1, TSP1)和结缔组织生长因子(connective tissue growth factor, CTGF). 此外, O'Donnell等[33]研究发现不仅转录因子c-Myc可调节许多miRNAs, 反过来, 特定miRNAs(如miR-17-5p和miR-20a)又可调节E2F的表达.
COX-2可强烈促进CRC肿瘤细胞的生长和侵袭[34]. COX-2过表达依赖于各种参与转录和转录后调节的细胞通路. 在CRC细胞系中COX-2和miR-101的表达负相关, miR-101可直接介导对COX-2 mRNA翻译的抑制. 此外, 针对CRC及其肝转移组织的分析也证实了两者之间的相互关系. miR-101表达的缺失可能是CRC细胞系中COX-2过表达的主要原因之一[34].
缺氧细胞可表现出抗凋亡和细胞周期停滞特性, 因此缺氧常被认为是肿瘤的一个生物学特征. 许多研究证实缺氧不仅对肿瘤生长起关键的调节作用, 还与肿瘤较强的侵袭力和较差的治疗效果相关联. 最近, Kulshreshtha等[35]发现缺氧可诱导结肠癌细胞系HT29和HCT116产生特异miRNAs表达谱(包括miR-23, -24, -26, -27, -103, -107, -181, -210和-213), 且其中一些是通过缺氧诱导因子(hypoxia induced factors, HIF)依赖机制产生的. 此外, 在缺氧环境中miR-26, -107和-210可减少促凋亡信号的传导, 提示特异miRNAs可促进肿瘤形成. 大多数缺氧诱导的miRNAs在肿瘤中过表达; 低氧环境引起HIF的释放, 而HIF又可通过miRNAs基因上的HIF结合位点诱导特异miRNAs的表达. 这些缺氧调节的miRNAs(hypoxia-regulated miRNAs, HRM)可抑制细胞死亡或细胞周期进程, 即HRM不仅可抑制caspases级联反应和其他凋亡组分, 如miR-23, miR-26和miR-181分别抑制caspase-7, BAK1和BIM; 还可影响细胞周期蛋白cyclin的表达, 如miR-26, miR-23和miR-21分别对cyclin D2/cyclin E1/cdk6, cyclin H和cdc25A表达的抑制. 最近还有研究发现, 缺氧还可诱导hsa-miR-210在乳腺癌组织中过表达, 且其表达水平是乳腺癌的一个独立的预后因素[36].
细胞外基质(extracellular matrix, ECM)及其重塑对肿瘤细胞的血供与其周围间质的相互作用起重要调控作用. ECM重塑是肿瘤生长, 生存, 侵袭和转移的必要条件. 参与ECM破坏的关键酶多为蛋白酶, 且其中一些是尿激酶纤维蛋白溶酶原激活剂和基质金属蛋白酶(matrix metalloproteinases, MMPs)[37]. 研究发现, miR-21可调节与细胞运动和ECM重塑相关的多种基因, 如抑癌基因RECK, TIMP3及MMPs抑制因子[38], 且其表达水平在CRC和其他不同起源肿瘤中显著升高[19]. 应用反义寡核苷酸特异性抑制miR-21的表达可致RECK和TIMP3表达上调, 从而降低MMPs的活性. miR-21表达上调也可增强CRC细胞的迁移和侵袭力, 其作用机制与在胶质母细胞瘤模型中的相类似[39]. 已知抑癌基因PDCD4是CRC肿瘤组织中一个独立的预后因素和新的血管内渗调节物. miR-21通过侵袭相关的尿激酶受体可作用于PDCD4. 在CRC细胞系中, miR-21的表达沉默可上调PDCD4表达水平并降低细胞侵袭力. 相比于正常组织, miR-21在22例CRC组织中表达增高, 而PDCD4蛋白水平降低, mRNA表达水平并未变化[39].
通常将上皮细胞向间质细胞的转化简称为上皮间质转化(epithelial-mesenchymal transition, EMT). 因EMT在形态学上常具有E-钙黏蛋白减少, 细胞黏附缺失和细胞运动性增强等特点, 所以他可促进癌细胞迁移[40]. 转录抑制因子锌指E-盒结合同源异形盒(zinc-finger E-box binding homeobox 1, ZEB1)是许多肿瘤(包括CRC)EMT的重要诱导物[41]. 最近研究发现ZEB1可以促进肿瘤细胞的侵袭和转移. miR-200家族(miR-200a, miR-200b, miR-200c, miR-141和miR-429)与EMT的功能相关联. ZEB1可直接抑制miR-200家族成员miR-141和miR-200c(两者可强烈激活包括CRC在内的一些肿瘤上皮细胞的分化)的转录[42]. 另外, EMT激活物转化生长因子β2和ZEB1是miR-200家族下调的主要靶点. 这些结果提示ZEB1触发了一个可稳定EMT并促进癌细胞侵袭的miRNAs介导的前反馈环. 此环可转换或诱导上皮细胞分化, 这可能是肿瘤内异质性的一个原因.
包括miRNAs在内的非编码RNAs诱导产生的基因组修饰的改变称为表观遗传改变. 最近的研究显示, 在肿瘤生成中DNA甲基化和组蛋白修饰可调节miRNAs表达. 通过比较结肠癌细胞系HCT116与DNA甲基转移酶1和3b(DNMT1和DNMT3b)双敲除细胞系的miRNAs表达谱, Han等[43]发现约有10%的miRNAs受DNA甲基化调控. 在CRC中, hsa-miR-342宿主基因EVL上游的CpG岛甲基化导致此miRNAs表达沉默[44]; 在不同细胞类型的肿瘤中, CpG岛超甲基化可致miR-124a的表达失活[45]; 此外, CpG岛甲基化对CRC中miR-34b/c的表观沉默起关键作用[46]. Bandres等[47]研究发现, 位于CpG 岛的miRNAs(hsa-miR-9, hsa-miR-124, hsa-miR-129, hsa-miR-137和hsa-miR-149)在CRC组织标本中表达下调. 而当使用DNA甲基转移酶抑制剂和组蛋白脱乙酰基酶抑制剂处理3种CRC细胞系后, 其中3个miRNAs(hsa-miR-9, hsa-miR-129 andhsa-miR-137)表达水平上调. 这提示异常的DNA甲基化和组蛋白修饰可诱导CRC中的miRNAs表达沉默. 当然并非所有的miRNAs均受表观基因组调控, 例如当给予CRC细胞系Colo320和SW1116染色质重塑药物处理后, 可改变细胞系中抑癌基因的表达水平[48]. 在B细胞淋巴瘤系中并未发现miR-143和miR-145表达上调, 提示表观遗传改变并未对两者起作用[49]. DNA甲基化对miRNAs的调控相当复杂, 有关在CRC中miRNAs的甲基化模式仍需进一步的研究.
现已发现miR-34家族在许多肿瘤(包括CRC)中低表达[22]. Tazawa等[50]报道miR-34a在36%(9/25)的CRC中表达下调, 并认为CpG岛超甲基化可能是导致miR-34家族表达水平减少的主要原因. miR-34a启动子区甲基化存在于13%(3/23)结肠癌组织标本中[51]; 而miR-34b/c的表达在100%(9/9)结肠癌细胞系和90%(101/111)原发CRC肿瘤中发生表观沉默, 但在正常结肠上皮中则表达正常. 当给予结肠癌细胞系去甲基化因子处理后, miR-34b/c的表达水平又可恢复至正常水平, 并抑制肿瘤细胞转移[46]. miR-34家族在CRC中的高频甲基化及其在P53基因转录网络中的作用提示, miR-34家族成员也是一种与P53作用类似的抑癌基因, 即其表达失活导致了癌症的发生.
miRNAs与mRNAs的结合对调节靶基因表达至关重要. 然而, 这种结合可受miRNAs靶基因位点内单核苷酸多态性(single-nucleotide polymorphisms, SNPs)的影响, SNPs既可消除现已存在的结合位点, 也可产生不合理的结合位点. 因此, miRNAs结合位点内的SNPs对基因和蛋白表达均有影响, 这代表了另一种可影响人类疾病(包括CRC)危险因素的遗传变异类型. 已有许多方法可预测和鉴定功能性miRNAs结合位点内的SNPs, 并在大量的病例对照研究中评估其生物学意义[52]. 就CRC而言, 在8个计算机模拟预测的候选基因中, 淋巴细胞共同刺激性配体CD86和胰岛素受体基因均携带一个SNPs, 这与散发CRC的危险性显著相关(比值比分别为2.74和1.94)[53]. 然而这些SNPs的生物学意义需要进一步在体外功能性研究中明确.
CRC的发生发展是一个多步骤、多途径、多基因参与的复杂渐进过程, 近年来越来越多有关miRNAs参与细胞信号转导, 肿瘤微环境改变及SNPs调控的研究进一步补充和丰富了CRC形成的分子生物学与遗传学机制. 此外, 相比于传统的遗传学改变, 表观基因组遗传修饰改变对CRC的发生也具有重要意义. 总之, 深入阐明miRNAs调控网络在CRC发病机制中的作用对CRC临床诊治和预后判断具有重要的指导意义.
大肠癌(CRC)是一个多基因、多阶段、长期形成的复杂的病变过程, 近年来研究认为miRNAs通过多种信号通路广泛参与恶性肿瘤(包括CRC)的发生和发展.
戴冬秋, 教授, 中国医科大学附属第一医院肿瘤外科
进一步明确miRNAs参与细胞信号转导, 肿瘤微环境改变及SNPs调控机制及CRC形成的分子生物学与遗传学机制, 对CRC临床诊治和预后判断具有重要的指导意义.
Dews等利用小鼠结肠癌模型证实miR-17-92簇可部分激活c-Myc的血管生成活性. c-Myc可显著诱导这些miRNAs负性调节抗血管生成因子凝血酶敏感蛋白1(TSP1)和结缔组织生长因子(CTGF).
本文系统综述了microRNAs调控网络在大肠癌发病机制中作用, 重点总结了CRC中的miRNAs调控网络(包括各种信号通路, 如Wnt/β-连环蛋白, KRAS和PI3-K及P53信号通路等); 其他文献并未对近年来miRNAs参与调控CRC发病的信号通路进行系统总结, 并明确其重要意义.
本文综述了大肠癌基础研究的前沿内容之一, 对国内同行有较好的参考价值.
编辑: 李军亮 电编: 何基才
| 1. | Jemal A, Siegel R, Ward E, Hao Y, Xu J, Thun MJ. Cancer statistics, 2009. CA Cancer J Clin. 2009;59:225-249. [PubMed] [DOI] |
| 2. | Yang L, Parkin DM, Li L, Chen Y. Time trends in cancer mortality in China: 1987-1999. Int J Cancer. 2003;106:771-783. [PubMed] [DOI] |
| 3. | Mirnezami AH, Pickard K, Zhang L, Primrose JN, Packham G. MicroRNAs: key players in carcinogenesis and novel therapeutic targets. Eur J Surg Oncol. 2009;35:339-347. [PubMed] [DOI] |
| 4. | Rossi S, Kopetz S, Davuluri R, Hamilton SR, Calin GA. MicroRNAs, ultraconserved genes and colorectal cancers. Int J Biochem Cell Biol. 2010;42:1291-1297. [PubMed] |
| 5. | Li M, Marin-Muller C, Bharadwaj U, Chow KH, Yao Q, Chen C. MicroRNAs: control and loss of control in human physiology and disease. World J Surg. 2009;33:667-684. [PubMed] [DOI] |
| 6. | Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell. 2003;115:787-798. [PubMed] [DOI] |
| 7. | Wu W, Sun M, Zou GM, Chen J. MicroRNA and cancer: Current status and prospective. Int J Cancer. 2007;120:953-960. [PubMed] [DOI] |
| 8. | Fearon ER, Vogelstein B. A genetic model for colorectal tumorigenesis. Cell. 1990;61:759-767. [PubMed] [DOI] |
| 9. | Faber C, Kirchner T, Hlubek F. The impact of microRNAs on colorectal cancer. Virchows Arch. 2009;454:359-367. [PubMed] [DOI] |
| 10. | Aslam MI, Taylor K, Pringle JH, Jameson JS. MicroRNAs are novel biomarkers of colorectal cancer. Br J Surg. 2009;96:702-710. [PubMed] [DOI] |
| 11. | Nagel R, le Sage C, Diosdado B, van der Waal M, Oude Vrielink JA, Bolijn A, Meijer GA, Agami R. Regulation of the adenomatous polyposis coli gene by the miR-135 family in colorectal cancer. Cancer Res. 2008;68:5795-5802. [PubMed] [DOI] |
| 12. | Ciardiello F, Tortora G. EGFR antagonists in cancer treatment. N Engl J Med. 2008;358:1160-1174. [PubMed] [DOI] |
| 13. | Johnson SM, Grosshans H, Shingara J, Byrom M, Jarvis R, Cheng A, Labourier E, Reinert KL, Brown D, Slack FJ. RAS is regulated by the let-7 microRNA family. Cell. 2005;120:635-647. [PubMed] [DOI] |
| 14. | Akao Y, Nakagawa Y, Naoe T. let-7 microRNA functions as a potential growth suppressor in human colon cancer cells. Biol Pharm Bull. 2006;29:903-906. [PubMed] [DOI] |
| 15. | Chen X, Guo X, Zhang H, Xiang Y, Chen J, Yin Y, Cai X, Wang K, Wang G, Ba Y. Role of miR-143 targeting KRAS in colorectal tumorigenesis. Oncogene. 2009;28:1385-1392. [PubMed] [DOI] |
| 16. | Tsang WP, Kwok TT. The miR-18a* microRNA functions as a potential tumor suppressor by targeting on K-Ras. Carcinogenesis. 2009;30:953-959. [PubMed] [DOI] |
| 17. | Guo C, Sah JF, Beard L, Willson JK, Markowitz SD, Guda K. The noncoding RNA, miR-126, suppresses the growth of neoplastic cells by targeting phosphatidylinositol 3-kinase signaling and is frequently lost in colon cancers. Genes Chromosomes Cancer. 2008;47:939-946. [PubMed] [DOI] |
| 18. | Meng F, Henson R, Wehbe-Janek H, Ghoshal K, Jacob ST, Patel T. MicroRNA-21 regulates expression of the PTEN tumor suppressor gene in human hepatocellular cancer. Gastroenterology. 2007;133:647-658. [PubMed] [DOI] |
| 19. | Krichevsky AM, Gabriely G. miR-21: a small multi-faceted RNA. J Cell Mol Med. 2009;13:39-53. [PubMed] [DOI] |
| 20. | Slaby O, Svoboda M, Fabian P, Smerdova T, Knoflickova D, Bednarikova M, Nenutil R, Vyzula R. Altered expression of miR-21, miR-31, miR-143 and miR-145 is related to clinicopathologic features of colorectal cancer. Oncology. 2007;72:397-402. [PubMed] [DOI] |
| 21. | Schetter AJ, Leung SY, Sohn JJ, Zanetti KA, Bowman ED, Yanaihara N, Yuen ST, Chan TL, Kwong DL, Au GK. MicroRNA expression profiles associated with prognosis and therapeutic outcome in colon adenocarcinoma. JAMA. 2008;299:425-436. [PubMed] [DOI] |
| 22. | He L, He X, Lowe SW, Hannon GJ. microRNAs join the p53 network--another piece in the tumour-suppression puzzle. Nat Rev Cancer. 2007;7:819-822. [PubMed] [DOI] |
| 23. | Hermeking H. p53 enters the microRNA world. Cancer Cell. 2007;12:414-418. [PubMed] [DOI] |
| 24. | Chang TC, Wentzel EA, Kent OA, Ramachandran K, Mullendore M, Lee KH, Feldmann G, Yamakuchi M, Ferlito M, Lowenstein CJ. Transactivation of miR-34a by p53 broadly influences gene expression and promotes apoptosis. Mol Cell. 2007;26:745-752. [PubMed] [DOI] |
| 25. | Bommer GT, Gerin I, Feng Y, Kaczorowski AJ, Kuick R, Love RE, Zhai Y, Giordano TJ, Qin ZS, Moore BB. p53-mediated activation of miRNA34 candidate tumor-suppressor genes. Curr Biol. 2007;17:1298-1307. [PubMed] [DOI] |
| 26. | Corney DC, Flesken-Nikitin A, Godwin AK, Wang W, Nikitin AY. MicroRNA-34b and MicroRNA-34c are targets of p53 and cooperate in control of cell proliferation and adhesion-independent growth. Cancer Res. 2007;67:8433-8438. [PubMed] [DOI] |
| 27. | Yamakuchi M, Ferlito M, Lowenstein CJ. miR-34a repression of SIRT1 regulates apoptosis. Proc Natl Acad Sci U S A. 2008;105:13421-13426. [PubMed] [DOI] |
| 28. | Pechlivanis S, Pardini B, Bermejo JL, Wagner K, Naccarati A, Vodickova L, Novotny J, Hemminki K, Vodicka P, Försti A. Insulin pathway related genes and risk of colorectal cancer: INSR promoter polymorphism shows a protective effect. Endocr Relat Cancer. 2007;14:733-740. [PubMed] [DOI] |
| 29. | Shi B, Sepp-Lorenzino L, Prisco M, Linsley P, deAngelis T, Baserga R. Micro RNA 145 targets the insulin receptor substrate-1 and inhibits the growth of colon cancer cells. J Biol Chem. 2007;282:32582-32590. [PubMed] [DOI] |
| 30. | La Rocca G, Badin M, Shi B, Xu SQ, Deangelis T, Sepp-Lorenzinoi L, Baserga R. Mechanism of growth inhibition by MicroRNA 145: the role of the IGF-I receptor signaling pathway. J Cell Physiol. 2009;220:485-491. [PubMed] [DOI] |
| 31. | Woods K, Thomson JM, Hammond SM. Direct regulation of an oncogenic micro-RNA cluster by E2F transcription factors. J Biol Chem. 2007;282:2130-2134. [PubMed] [DOI] |
| 32. | Dews M, Homayouni A, Yu D, Murphy D, Sevignani C, Wentzel E, Furth EE, Lee WM, Enders GH, Mendell JT. Augmentation of tumor angiogenesis by a Myc-activated microRNA cluster. Nat Genet. 2006;38:1060-1065. [PubMed] [DOI] |
| 33. | O'Donnell KA, Wentzel EA, Zeller KI, Dang CV, Mendell JT. c-Myc-regulated microRNAs modulate E2F1 expression. Nature. 2005;435:839-843. [PubMed] [DOI] |
| 34. | Strillacci A, Griffoni C, Sansone P, Paterini P, Piazzi G, Lazzarini G, Spisni E, Pantaleo MA, Biasco G, Tomasi V. MiR-101 downregulation is involved in cyclooxygenase-2 overexpression in human colon cancer cells. Exp Cell Res. 2009;315:1439-1447. [PubMed] [DOI] |
| 35. | Kulshreshtha R, Ferracin M, Wojcik SE, Garzon R, Alder H, Agosto-Perez FJ, Davuluri R, Liu CG, Croce CM, Negrini M. A microRNA signature of hypoxia. Mol Cell Biol. 2007;27:1859-1867. [PubMed] [DOI] |
| 36. | Camps C, Buffa FM, Colella S, Moore J, Sotiriou C, Sheldon H, Harris AL, Gleadle JM, Ragoussis J. hsa-miR-210 Is induced by hypoxia and is an independent prognostic factor in breast cancer. Clin Cancer Res. 2008;14:1340-1348. [PubMed] [DOI] |
| 37. | Takayama T, Miyanishi K, Hayashi T, Sato Y, Niitsu Y. Colorectal cancer: genetics of development and metastasis. J Gastroenterol. 2006;41:185-192. [PubMed] [DOI] |
| 38. | Gabriely G, Wurdinger T, Kesari S, Esau CC, Burchard J, Linsley PS, Krichevsky AM. MicroRNA 21 promotes glioma invasion by targeting matrix metalloproteinase regulators. Mol Cell Biol. 2008;28:5369-5380. [PubMed] [DOI] |
| 39. | Asangani IA, Rasheed SA, Nikolova DA, Leupold JH, Colburn NH, Post S, Allgayer H. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer. Oncogene. 2008;27:2128-2136. [PubMed] [DOI] |
| 40. | Natalwala A, Spychal R, Tselepis C. Epithelial-mesenchymal transition mediated tumourigenesis in the gastrointestinal tract. World J Gastroenterol. 2008;14:3792-3797. [PubMed] [DOI] |
| 41. | Spaderna S, Schmalhofer O, Hlubek F, Berx G, Eger A, Merkel S, Jung A, Kirchner T, Brabletz T. A transient, EMT-linked loss of basement membranes indicates metastasis and poor survival in colorectal cancer. Gastroenterology. 2006;131:830-840. [PubMed] [DOI] |
| 42. | Burk U, Schubert J, Wellner U, Schmalhofer O, Vincan E, Spaderna S, Brabletz T. A reciprocal repression between ZEB1 and members of the miR-200 family promotes EMT and invasion in cancer cells. EMBO Rep. 2008;9:582-589. [PubMed] [DOI] |
| 43. | Han L, Witmer PD, Casey E, Valle D, Sukumar S. DNA methylation regulates MicroRNA expression. Cancer Biol Ther. 2007;6:1284-1288. [PubMed] |
| 44. | Grady WM, Parkin RK, Mitchell PS, Lee JH, Kim YH, Tsuchiya KD, Washington MK, Paraskeva C, Willson JK, Kaz AM. Epigenetic silencing of the intronic microRNA hsa-miR-342 and its host gene EVL in colorectal cancer. Oncogene. 2008;27:3880-3888. [PubMed] [DOI] |
| 45. | Lujambio A, Ropero S, Ballestar E, Fraga MF, Cerrato C, Setién F, Casado S, Suarez-Gauthier A, Sanchez-Cespedes M, Git A. Genetic unmasking of an epigenetically silenced microRNA in human cancer cells. Cancer Res. 2007;67:1424-1429. [PubMed] [DOI] |
| 46. | Toyota M, Suzuki H, Sasaki Y, Maruyama R, Imai K, Shinomura Y, Tokino T. Epigenetic silencing of microRNA-34b/c and B-cell translocation gene 4 is associated with CpG island methylation in colorectal cancer. Cancer Res. 2008;68:4123-4132. [PubMed] [DOI] |
| 47. | Bandres E, Agirre X, Bitarte N, Ramirez N, Zarate R, Roman-Gomez J, Prosper F, Garcia-Foncillas J. Epigenetic regulation of microRNA expression in colorectal cancer. Int J Cancer. 2009;125:2737-2743. [PubMed] [DOI] |
| 48. | Fang JY, Chen YX, Lu J, Lu R, Yang L, Zhu HY, Gu WQ, Lu LG. Epigenetic modification regulates both expression of tumor-associated genes and cell cycle progressing in human colon cancer cell lines: Colo-320 and SW1116. Cell Res. 2004;14:217-226. [PubMed] [DOI] |
| 49. | Akao Y, Nakagawa Y, Kitade Y, Kinoshita T, Naoe T. Downregulation of microRNAs-143 and -145 in B-cell malignancies. Cancer Sci. 2007;98:1914-1920. [PubMed] [DOI] |
| 50. | Tazawa H, Tsuchiya N, Izumiya M, Nakagama H. Tumor-suppressive miR-34a induces senescence-like growth arrest through modulation of the E2F pathway in human colon cancer cells. Proc Natl Acad Sci U S A. 2007;104:15472-15477. [PubMed] [DOI] |
| 51. | Lodygin D, Tarasov V, Epanchintsev A, Berking C, Knyazeva T, Körner H, Knyazev P, Diebold J, Hermeking H. Inactivation of miR-34a by aberrant CpG methylation in multiple types of cancer. Cell Cycle. 2008;7:2591-2600. [PubMed] [DOI] |
| 52. | Chen K, Song F, Calin GA, Wei Q, Hao X, Zhang W. Polymorphisms in microRNA targets: a gold mine for molecular epidemiology. Carcinogenesis. 2008;29:1306-1311. [PubMed] [DOI] |
| 53. | Landi D, Gemignani F, Naccarati A, Pardini B, Vodicka P, Vodickova L, Novotny J, Försti A, Hemminki K, Canzian F. Polymorphisms within micro-RNA-binding sites and risk of sporadic colorectal cancer. Carcinogenesis. 2008;29:579-584. [PubMed] [DOI] |