修回日期: 2010-03-19
接受日期: 2010-03-23
在线出版日期: 2010-04-18
目的: 探讨肿瘤细胞中zeb1基因的表达量与肿瘤细胞的迁移能力之间的关系.
方法: 实时定量PCR方法检测正常胃黏膜上皮细胞GES及四种肿瘤细胞BGC823、SGC7901、A549和HeLa细胞中zeb1基因的表达量; Transwell小室检测五种细胞的迁移能力.
结果: 在五种细胞中, zeb1在HeLa细胞中表达量最高, BGC823及SGC7901次之, 在A549及GES中表达量最低; 发生迁移的细胞数目在HeLa细胞中最多, BGC823及SGC7901其次, 在A549及GES细胞中最少; 线性相关分析表明, zeb1基因的表达量与细胞迁移能力呈正相关(r = 0.961, P<0.01).
结论: zeb1基因可能促进肿瘤细胞的迁移能力.
引文著录: 宋永站, 张尤历, 乌慧玲, 孔梅, 陈鑫, 邵长江. zeb1基因与肿瘤细胞迁移能力的关系. 世界华人消化杂志 2010; 18(11): 1099-1103
Revised: March 19, 2010
Accepted: March 23, 2010
Published online: April 18, 2010
AIM: To explore the relationship between the expression of zeb1 gene and the migration ability of tumor cells.
METHODS: Real-time PCR was used to detect the expression of zeb1 gene in gastric epithelial GES-1 cells and four types of tumor cells: BGC823, SGC7901, A549 and HeLa cells. Cell migration was measured by Transwell assay.
RESULTS: The expression level of zeb1 gene was highest in HeLa cells, followed by SGC7901, BGC823, A549 and GES-1 cells. HeLa cells had highest migration ability , followed by SGC7901, BGC823, A549 and GES-1 cells. A significant correlation was noted between the expression of zeb1 gene and cell migration ability (r = 0.961, P < 0.01).
CONCLUSION: zeb1 may promote the migration of tumor cells.
- Citation: Song YZ, Zhang YL, Wu HL, Kong M, Chen X, Shao CJ. zeb1 may promote the migration of tumor cells. Shijie Huaren Xiaohua Zazhi 2010; 18(11): 1099-1103
- URL: https://www.wjgnet.com/1009-3079/full/v18/i11/1099.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i11.1099
细胞的迁移运动不仅是细胞进行很多重要生理活动的基础, 同时也在炎症反应和肿瘤发生发展等病理过程中的发挥关键作用[1-3]. zeb1(zinc-finger E-box binding homeobox 1), 又称为TCF8或δEF1, 位于人类10号染色体短臂上, 编码序列长3 327 bp, 编码1 108个氨基酸, 含有两个锌指结构簇及一个同源结构域, 其作为转录因子被发现可以与鸡的δ1-晶体蛋白基因的增强子结合[4]. 研究发现zeb1在多种细胞系中可以抑制多种基因的表达, 如在造血细胞中zeb1可以负性调控IL-2、免疫球蛋白μ重链、CD4和GATA-3等的表达[5-8]; 在间充质细胞中, zeb1可以抑制p73基因的表达[9]; 在格根包尔氏细胞中, zeb1可以抑制Ⅰ型和Ⅱ型胶原的表达[10,11]. 新近的研究发现zeb1在肿瘤的发生发展及肿瘤细胞的侵袭及转移过程中也起到十分重要的作
用[12-15]. 但其在胃癌细胞中的表达情况及与肿瘤细胞的迁移能力是否有相关性研究较少. 本研究观察了几种常见恶性肿瘤细胞中zeb1基因的表达量与肿瘤细胞的迁移能力, 以探讨zeb1基因与肿瘤细胞的迁移能力之间的关系.
正常胃黏膜上皮GES细胞, 胃癌BGC823、SGC7901细胞, 宫颈癌HeLa细胞, 肺癌A549细胞均由本实验室保存; 高糖DMEM培养基购自Gibco公司; 胎牛血清购自杭州四季青公司; TRIzol reagent购自Invitrogen公司; 逆转录试剂盒购自TaKaRa公司; 引物序列由上海生工生物工程技术服务有限公司合成; 荧光定量试剂盒购自Takara公司; Transwell小室购自Costar公司(USA); 多聚甲醛、结晶紫均为国产分析级产品.
1.2.1 细胞培养: GES、BGC823、SGC7901、A549及HeLa细胞均于含100 mL/L FBS的高糖DMEM培养液37 ℃、50 mL/L CO2饱和湿度条件下培养. 隔2 d换液, 细胞融合达80%时按1:3比例传代培养, 取对数生长期细胞进行实验.
1.2.2 提取RNA及逆转录合成cDNA: RNA提取参照TRIzol提取试剂盒的操作说明书进行. 提取的RNA纯度A260/A280均在1.80以上. 照逆转录试剂盒说明书将细胞的总RNA逆转录成cDNA, 合成的cDNA于-20 ℃保存.
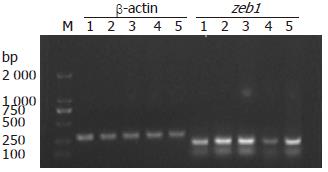
1.2.3 PCR验证zeb1基因在各细胞系中的表达: 反应体系如下(20 μL): ddH2O 13.6 μL, 10×Buffer 2 μL, Mg2+ 2 μL, dNTP 0.5 μL, Taq 0.3 μL, 引物P1、P2各0.3 μL, 模板cDNA 1 μL. 反应条件: 94 ℃ 5 min, 95 ℃ 30 s, 56 ℃ 30 s, 72 ℃ 40 s, 35个循环, 72 ℃ 10 min. zeb1引物序列P1: 5'-GCACAACCAAGTGCAGAAGA-3', P2: 5'-CATTTGCAGATTGAGGCTGA-3', (GenBank登录号: NM_001128128.1, 扩增片段大小为190 bp).
1.2.4 荧光定量PCR检测zeb1在各细胞系中的表达量: 反应体系: SYBR 10 μL, P1 0.4 μL, P2 0.4 μL, ROXⅡ 0.4 μL, 模板cDNA 1.6 μL, ddH2O 7.2 μL. 反应条件为: 95 ℃ 10 s, 95 ℃ 5 s, 56 ℃ 20 s, 40个循环. 引物序列同上. 以β-actin为内参, 校正每个样品的CT值, 其引物序列为P1: 5'-AGCGAGCATCCCCCAAAGTT-3', P2: 5'-GGGCACGAAGGCTCATCATT-3', 每种细胞重复3次. GenBank登录号: BC002409, 扩增片段大小为285 bp. 采用相对定量法分析数据, 将正常胃黏膜上皮细胞GES中的zeb1表达量设定为1, 根据2-ΔΔCt公式计算基因相对拷贝数, 利用SPSS13.0软件对获得的数据进行分析.
1.2.5 Transwell小室迁移实验: 取对数生长期细胞, 常规消化离心, 用无血清的DMEM的培养液重悬细胞, 调整细胞浓度至5×107/L, 小室内加入200 μL细胞悬液, 小室下部24孔板内加入600 μL含100 mL/L FBS的DMEM培养液, 常规培养24 h后取出小室, 棉签擦去小室内未迁移的细胞, 4%多聚甲醛固定15 min, 1%结晶紫染色15 min, 单蒸水洗掉染色液后风干, 放倒置显微镜下观察, 100倍视野下取上中下左右5个视野计数穿过小室的细胞数目, 以穿过膜的细胞数目的多少来评估其迁移能力, 结果采用mean±SD来表示, 每组细胞重复3次实验.
统计学处理 数据以mean±SD, 采用SPSS13.0统计软件对所得数据进行单因素方差分析, 两两比较采用LSD法, 以P<0.05为差异有统计学意义. 对定量结果和迁移结果之间进行相关性分析.
经PCR电泳跑胶表明在五种细胞中都有zeb1基因的表达, 扩增片段大小与预期大小一致均为190 bp(图1).
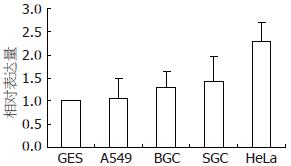
以各细胞的cDNA为模板进行Real time PCR检测zeb1基因的表达量, 可见到4种肿瘤细胞中肺癌A549细胞中的zeb1表达量最低, 胃癌细胞BGC823及SGC7901的zeb1表达量其次, 宫颈癌HeLa细胞中的zeb1表达量最高. 其中HeLa细胞与其他四组细胞间的zeb1基因的表达量有明显差异, 具有统计学意义(P<0.05), 而其他各组细胞间zeb1基因的表达量无明显差异(图2).
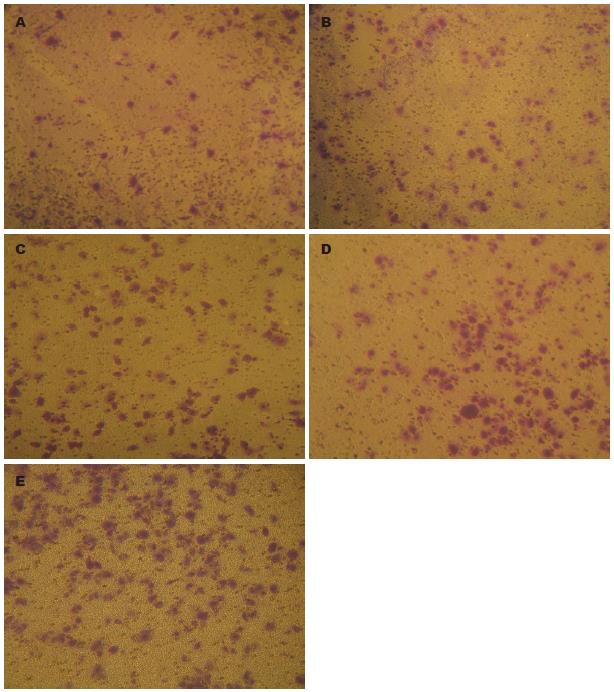
正常胃黏膜上皮细胞GES的迁移细胞数目最少, 而HeLa细胞迁移细胞数目最多. 各细胞系的迁移数目除GES与A549、A549与BGC823、SGC7901与HeLa之间无明显差异外, 其余各组细胞间迁移细胞数目有明显统计学意义(P<0.05, 表1, 图3).
| 细胞 | 迁移细胞数目 |
| GES | 79.13±17.12 |
| A549 | 97.60±25.21 |
| BGC823 | 108.93±39.16 |
| SGC7901 | 132.73±45.02 |
| HeLa | 169.07±32.11 |
zeb1表达量与细胞迁移能力之间存在线性关系(r = 0.961, P<0.01), zeb1的表达量越高, 细胞的迁移数目越多.
侵袭性与转移性是恶性肿瘤最重要的生物学特征, 也是肿瘤患者致死的主要原因[16]. 肿瘤的侵袭和转移包括细胞黏附、蛋白酶水解组织基质以及细胞迁移等进程. 其中细胞迁移运动普遍存在于组织发育、伤口愈合、血管发生等生理过程中, 同时又存在于肿瘤发生发展这样的病理过程中[1-3]. 在迁移过程中, 细胞内细胞骨架蛋白的动态组装, 细胞和细胞外基质间黏着的动态变化、周围基质的重塑等以及这些反应在迁移过程中的协调涉及复杂的信号调节.
上皮间质转化(epithelial-mesenchymal transitions, EMT)参与人体许多的生理病理过程, 包括胚胎发生, 器官分化, 组织炎症, 创伤愈合等, 越来越多的研究表明异常活化的EMT还可以促进肿瘤细胞的侵袭及转移[17,18]. 在EMT过程中, 上皮细胞获得成纤维样细胞的特征: 细胞间黏附减弱、运动性增强, 且细胞间紧密连接及细胞极性均被破坏. EMT的一个重要标志是E-钙黏蛋白(E-cadherin)表达的下调, E-钙黏蛋白的表达水平与肿瘤发生的阶段相关[19,20]. 许多转录因子抑制E-钙黏蛋白的表达, 如: Snail/Slug家族蛋白、Twist、δEF1/zeb1、SIP1和E12/E47. 其中关于zeb1促进肿瘤细胞的侵袭和转移的研究主要集中在他的对E-钙黏蛋白及T-钙黏蛋白的抑制作用[12,21]. 同时zeb1还被发现有促进TGF-β介导的抗增殖信号的作用[22]. Drake等[23]的研究发现某些前列腺癌细胞系的迁移能力与zeb1基因的表达有关, 但不具有普遍性, 其机制还有待进一步的研究.
本实验采用实时定量PCR的方法检测了GES、BGC823、SGC7901、A549及HeLa细胞中zeb1基因的相对表达量, 同时用Transwell小室法检测了这五种细胞的迁移能力, 对两者之间的关系进行线性相关分析, 结果表明在zeb1表达量的多少与细胞的迁移能力之间呈正相关, 说明zeb1可能促进肿瘤细胞的迁移能力, 对研究肿瘤细胞迁移的机制提供了新的方向与靶点. 但其究竟是通过什么途径达到促进其迁移能力等有待进一步研究. 近来的研究认为黏着斑(focal adhension)[24], 整合素(integrin)[25], 黏着斑激酶(focal adhension kinase, FAK)[26], RHO家族蛋白[27]等在细胞的迁移中发挥重要作用. 进一步的关于zeb1促迁移机制的研究我们可以从以上几个方面来进行, 后续相关试验正在进行中.
恶性肿瘤细胞的侵袭转移能力是肿瘤患者死亡的主要原因, 也是临床治疗面临的巨大困难之一. 早期发现肿瘤并抑制其侵袭转移成为治疗的关键. 寻找肿瘤迁移侵袭相关基因, 阐明其作用机制, 从而为临床治疗提供分子理论基础.
丁士刚, 主任医师, 北京大学第三医院消化科.
zeb1基因已经被证明在肿瘤细胞的侵袭机制中发挥了重要作用, 但其与迁移能力之间的关系尚不明确, 对于zeb1基因与迁移能力之间的关系成为新的研究方向.
Drake等的研究表明在某些肿瘤细胞系中zeb1基因的表达与肿瘤细胞的迁移能力有相关性, 但并不是在所有的肿瘤细胞系中都存在这种关系, 其机制有待进一步的研究.
本文首次在多种肿瘤细胞系中对zeb1基因的表达与肿瘤细胞的迁移能力进行分析, 数据更具说服力.
本研究为抑制恶性肿瘤细胞的侵袭转移提供了新的研究方向, zeb1基因有可能成为新的基因治疗靶点.
本文内容新颖, 值得进一步研究证实.
编辑: 李军亮 电编:吴鹏朕
| 1. | Ausprunk DH, Folkman J. Migration and proliferation of endothelial cells in preformed and newly formed blood vessels during tumor angiogenesis. Microvasc Res. 1977;14:53-65. [PubMed] |
| 2. | Han VK, D'Ercole AJ, Lund PK. Cellular localization of somatomedin (insulin-like growth factor) messenger RNA in the human fetus. Science. 1987;236:193-197. [PubMed] |
| 3. | Guvakova MA. Insulin-like growth factors control cell migration in health and disease. Int J Biochem Cell Biol. 2007;39:890-909. [PubMed] |
| 4. | Funahashi J, Sekido R, Murai K, Kamachi Y, Kondoh H. Delta-crystallin enhancer binding protein delta EF1 is a zinc finger-homeodomain protein implicated in postgastrulation embryogenesis. Development. 1993;119:433-446. [PubMed] |
| 5. | Williams TM, Moolten D, Burlein J, Romano J, Bhaerman R, Godillot A, Mellon M, Rauscher FJ 3rd, Kant JA. Identification of a zinc finger protein that inhibits IL-2 gene expression. Science. 1991;254:1791-1794. [PubMed] |
| 6. | Genetta T, Ruezinsky D, Kadesch T. Displacement of an E-box-binding repressor by basic helix-loop-helix proteins: implications for B-cell specificity of the immunoglobulin heavy-chain enhancer. Mol Cell Biol. 1994;14:6153-6163. [PubMed] |
| 7. | Brabletz T, Jung A, Hlubek F, Löhberg C, Meiler J, Suchy U, Kirchner T. Negative regulation of CD4 expression in T cells by the transcriptional repressor ZEB. Int Immunol. 1999;11:1701-1708. [PubMed] |
| 8. | Grégoire JM, Roméo PH. T-cell expression of the human GATA-3 gene is regulated by a non-lineage-specific silencer. J Biol Chem. 1999;274:6567-6578. [PubMed] |
| 9. | Fontemaggi G, Gurtner A, Strano S, Higashi Y, Sacchi A, Piaggio G, Blandino G. The transcriptional repressor ZEB regulates p73 expression at the crossroad between proliferation and differentiation. Mol Cell Biol. 2001;21:8461-8470. [PubMed] |
| 10. | Murray D, Precht P, Balakir R, Horton WE Jr. The transcription factor deltaEF1 is inversely expressed with type II collagen mRNA and can repress Col2a1 promoter activity in transfected chondrocytes. J Biol Chem. 2000;275:3610-3618. [PubMed] |
| 11. | Terraz C, Toman D, Delauche M, Ronco P, Rossert J. delta Ef1 binds to a far upstream sequence of the mouse pro-alpha 1(I) collagen gene and represses its expression in osteoblasts. J Biol Chem. 2001;276:37011-37019. [PubMed] |
| 12. | Eger A, Aigner K, Sonderegger S, Dampier B, Oehler S, Schreiber M, Berx G, Cano A, Beug H, Foisner R. DeltaEF1 is a transcriptional repressor of E-cadherin and regulates epithelial plasticity in breast cancer cells. Oncogene. 2005;24:2375-2385. [PubMed] |
| 13. | Spaderna S, Schmalhofer O, Hlubek F, Berx G, Eger A, Merkel S, Jung A, Kirchner T, Brabletz T. A transient, EMT-linked loss of basement membranes indicates metastasis and poor survival in colorectal cancer. Gastroenterology. 2006;131:830-840. [PubMed] |
| 14. | Aigner K, Dampier B, Descovich L, Mikula M, Sultan A, Schreiber M, Mikulits W, Brabletz T, Strand D, Obrist P. The transcription factor ZEB1 (deltaEF1) promotes tumour cell dedifferentiation by repressing master regulators of epithelial polarity. Oncogene. 2007;26:6979-6988. [PubMed] |
| 15. | Yang S, Du J, Wang Z, Yuan W, Qiao Y, Zhang M, Zhang J, Gao S, Yin J, Sun B. BMP-6 promotes E-cadherin expression through repressing deltaEF1 in breast cancer cells. BMC Cancer. 2007;7:211. [PubMed] |
| 16. | Müller A, Homey B, Soto H, Ge N, Catron D, Buchanan ME, McClanahan T, Murphy E, Yuan W, Wagner SN. Involvement of chemokine receptors in breast cancer metastasis. Nature. 2001;410:50-56. [PubMed] |
| 17. | Thompson EW, Newgreen DF, Tarin D. Carcinoma invasion and metastasis: a role for epithelial-mesenchymal transition? Cancer Res. 2005;65:5991-5995; discussion 5995. [PubMed] |
| 18. | Berx G, Raspé E, Christofori G, Thiery JP, Sleeman JP. Pre-EMTing metastasis? Recapitulation of morphogenetic processes in cancer. Clin Exp Metastasis. 2007;24:587-597. [PubMed] |
| 19. | Cowin P, Rowlands TM, Hatsell SJ. Cadherins and catenins in breast cancer. Curr Opin Cell Biol. 2005;17:499-508. [PubMed] |
| 20. | Junghans D, Haas IG, Kemler R. Mammalian cadherins and protocadherins: about cell death, synapses and processing. Curr Opin Cell Biol. 2005;17:446-452. [PubMed] |
| 21. | Adachi Y, Takeuchi T, Nagayama T, Ohtsuki Y, Furihata M. Zeb1-mediated T-cadherin repression increases the invasive potential of gallbladder cancer. FEBS Lett. 2009;583:430-436. [PubMed] |
| 22. | Postigo AA. Opposing functions of ZEB proteins in the regulation of the TGFbeta/BMP signaling pathway. EMBO J. 2003;22:2443-2452. [PubMed] |
| 23. | Drake JM, Strohbehn G, Bair TB, Moreland JG, Henry MD. ZEB1 enhances transendothelial migration and represses the epithelial phenotype of prostate cancer cells. Mol Biol Cell. 2009;20:2207-2217. [PubMed] |
| 24. | Zamir E, Geiger B. Molecular complexity and dynamics of cell-matrix adhesions. J Cell Sci. 2001;114:3583-3590. [PubMed] |
| 25. | Hynes RO. Integrins: bidirectional, allosteric signaling machines. Cell. 2002;110:673-687. [PubMed] |
| 26. | Parsons JT, Martin KH, Slack JK, Taylor JM, Weed SA. Focal adhesion kinase: a regulator of focal adhesion dynamics and cell movement. Oncogene. 2000;19:5606-5613. [PubMed] |
| 27. | Yan X, Shen Y, Zhu X. Live show of Rho GTPases in cell migration. J Mol Cell Biol. 2010;2:68-69. [PubMed] |