修回日期: 2009-02-23
接受日期: 2009-03-02
在线出版日期: 2009-03-28
目的: 通过结直肠癌与其他常见恶性肿瘤血清低分子差异蛋白的分析, 探讨其对结直肠癌的诊断特异性.
方法: 收集结直肠癌(68例)、其他恶性肿瘤(乳腺癌、胃癌、食管癌、肝癌、肺癌及肾癌各25例)血清标本共218份, 应用表面增强激光解吸/电离-飞行时间-质谱检测其蛋白质指纹谱. 用Biomarker Wizard软件分析结直肠癌与其他恶性肿瘤患者血清中的低分子差异蛋白后, 分别作出受试者工作特征(ROC)诊断曲线. 再选取ROC曲线下面积(AUC)>0.8的蛋白, 建立Fisher判别模型.
结果: 结直肠癌与其他恶性肿瘤相比, 血清中有12种蛋白相对高表达, 102种蛋白相对低表达. ROC曲线显示, 有75种蛋白AUC>0.5, 其中8911、8919、8964、11726、14049、14139 M/Z的蛋白AUC>0.8. 用这6种蛋白建立的Fisher判别模型, 鉴别结直肠癌的敏感性88.2%, 特异性93.3%, 准确率91.7%.
结论: 结直肠癌与其他恶性肿瘤相比, 血清中存在明显差异表达的低分子蛋白, 对结直肠癌具有诊断特异性, 值得进一步研究.
引文著录: 盛新华, 高春芳, 王秀丽, 李冬晖, 郑国宝. 结直肠癌与其他恶性肿瘤血清低分子差异蛋白的比较. 世界华人消化杂志 2009; 17(9): 945-949
Revised: February 23, 2009
Accepted: March 2, 2009
Published online: March 28, 2009
AIM: To evaluate the diagnostic specificity of differentially expressed serum low molecular proteins for colorectal cancer by comparative analysis between the patients with colorectal cancer and with other malignant tumors.
METHODS: A total of 218 serum samples from patients with colorectal cancer (n = 68) and other malignant tumors (breast, gastric, esophagus, liver, lung and kidney, 25 cases each) were collected. Their serum protein fingerprinting was read by surface-enhanced laser desorption/ionization-time of flight-mass spectrometry (SELDI-TOF-MS). Biomarker Wizard software was used to analyze the distinct proteins between colorectal cancer and other malignant tumors and to create receiver operating characteristic (ROC) curves. Then Fisher's discriminant model was established using the distinct proteins with the area under the curve (AUC) > 0.8.
RESULTS: Relatively 12 proteins were highly expressed and 102 proteins were lowly expressed in the sera of colorectal cancer compared with that of the other malignant tumors. Of all the 75 proteins with AUC > 0.5 by ROC analysis, six proteins (AUC > 0.8) were selected to establish the Fisher's discriminant model. The accuracy of distinguishing colorectal cancer from other malignant tumors was 91.7%, and the sensitivity and specificity were 88.2% and 93.3%, respectively.
CONCLUSION: The significantly differentially expressed serum low molecular proteins have a high diagnostic specificity for distinguishing colorectal cancer from other malignant tumors, which deserves further research.
- Citation: Sheng XH, Gao CF, Wang XL, Li DH, Zheng GB. Comparative analysis of differential serum low molecular weight proteins between colorectal cancer and other malignant tumors. Shijie Huaren Xiaohua Zazhi 2009; 17(9): 945-949
- URL: https://www.wjgnet.com/1009-3079/full/v17/i9/945.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i9.945
结直肠癌(colorectal carcinoma, CRC)是威胁人类健康与生命的恶性肿瘤之一. 在我国, 随着人们生活水平的提高、饮食结构及生活方式的改变, CRC的发病率呈上升趋势. 近年我们从血清蛋白质指纹谱研究入手, 应用表面增强激光解吸/电离-飞行时间-质谱(surface enhanced laser desorption/ionization-time of flight-mass spectrometry, SELDI-TOF-MS)技术, 建立了CRC早期诊断与临床分期诊断模型, 具有较高的敏感性与特异性[1-3]. 本实验我们仍采用血清差异蛋白质组研究方法, 从CRC与其他恶性肿瘤患者血清中筛选出了明显差异表达的低分子蛋白.
CRC与其他恶性肿瘤患者血清标本取自2006-06/2007-12中国人民解放军第150中心医院住院的手术患者, 均经术后病理确诊. CRC组68例, 男36例, 女32例, 年龄<40岁11例, 40-60岁32例, ≥60岁25例, 其中Dukes A期6例、B期28例、C期23例、D期11例. 其他恶性肿瘤组包括乳腺癌、胃癌、食管癌、肝癌、肺癌及肾癌各25例, 男78例, 女72例, 年龄<40岁31例, 40-60岁67例, ≥60岁52例. 正常人对照组67例, 男35例, 女32例, 年龄<40岁14例, 40-60岁29例, ≥60岁24例, 为同期本院健康体检人群. 以上三组人群在性别构成、年龄分布上均无统计学差异. 所有病例均排除能影响血清中蛋白质含量的其他相关疾病的干扰. 受检者均获知情同意. 尿素、乙腈(acetonitrile, ACN)、三氟乙酸(trifluoro-acetic, TFA)、芥子酸(sinapinic acid, SPA)、三羟基甲基氨基甲烷-盐酸缓冲液(Tris-HCl)、3-环乙胺-1-丙磺酸(CHAPS)及羟乙基哌嗪乙磺酸 (HEPES)等均购自美国Sigma公司. SELDI蛋白质芯片系统(PBSⅡ-C型)及其配套的金属亲和表面(immobilized metal affinity capture, IMAC30)铜离子芯片均购自美国Ciphergen公司.
1.2.1 血清标本的采集: 清晨空腹抽取静脉血5 mL于普通干燥管, 常温下放置不超过4 h或4℃冷藏不超过12 h, 析出血清后, 1000 r/min离心20 min, 取上清液分装, 每管100 μL, 置-80℃冰箱或液氮中保存. 用于质控的血清取自同一个健康人, 一次留取100 mL, 分装于Eppendorf管中, 每管50 μL, 置于-80℃低温冰箱中保存, 作为每次实验的质控血清.
1.2.2 样品的准备: 4℃融解血清, 20 000 g离心10 min; 取20 μL血清, 置于1.5 mL离心管中; 加40 μL U9缓冲液(含9 mol/L尿素、2% CHAPS、50 mmol/L Tris-HCl, pH9.0); 4℃振荡30 min, 使蛋白质变性; 取10 μL变性后的样品, 每管加入120 μL结合/洗脱缓冲液(含100 mmol/L磷酸钠、500 mmol/L氯化钠, pH7.0); 4℃混匀. 使最后上样的血清稀释约40倍.
1.2.3 芯片的预处理及与血清蛋白的结合: 芯片每孔加100 mmol/L硫酸铜50 μL, 室温下200 r/min振荡5 min, 倒除硫酸铜; 用去离子水冲洗5次后甩干, 每孔中加入100 mmol/L醋酸钠(pH4.0)50 μL, 室温下200 r/min振荡5 min; 去离子水冲洗5次后芯片每孔加入结合/洗脱缓冲液150 μL, 置于振荡器上, 室温孵育5 min后除去缓冲液, 重复1次, 后每孔加入50 μL稀释好的血清样品, 置于振荡器上, 4℃孵育60 min后除去缓冲液; 接着用150 μL结合/洗脱缓冲液冲洗2次, 每次振荡5 min, 最后1次用1 mmol/L HEPES (pH7.0)快速冲洗. 取出芯片, 待芯池即将风干时, 加入饱和SPA(SPA 1 mg溶于15 μL 50% ACN、15 μL 1% TFA中)2次, 每次0.5 μL. 风干后上机检测.
1.2.4 芯片检测与数据收集处理: SELDI蛋白芯片质谱仪在检测样品蛋白质指纹谱前用已知分子质量的标准蛋白质芯片校正, 使误差<0.1%. 检测时设定SELDI-TOF-MS的激光强度为175, 灵敏度为8, 收集数据的质荷比 (M/Z)范围为2000-20 000, 收集位置20-80. 用Ciphergen ProteinChip 3.2.0软件自动采集数据, 用Biomarker Wizard 3.2.0软件对芯片检测得到的蛋白质相对含量及蛋白质M/Z数据进行分析处理, Biomarker Patterns软件建立用于区分3组人群的决策分类树.
统计学处理 采用芯片系统自带的软件和SPSS13.0统计软件. 计数资料的比较用χ2检验. 应用受试者工作特征(receiver operating characteristic, ROC)曲线对差异蛋白进行评价, 并计算各自的ROC曲线下面积(area under the curve, AUC). 选取AUC>0.8的标志蛋白, 建立Fisher判别函数.
用质控标本进行SELDI-TOF-MS技术的稳定性测试, 将质控血清分别在同一芯片的8个点上同时检测以观察其批内的差异, 然后在不同实验日的三条芯片上再加以检测, 以观察其批间的差异. 结果显示, 该芯片检测到的同一个蛋白峰位置的批内和批间变异系数分别为0.3%和0.5%; 蛋白峰标准化强度(峰高)的批内和批间变异系数分别为9%和14%.
利用Biomarker Patterns软件对检测得到的3组人群的蛋白质指纹谱数据进行数据挖掘, 建立的决策分类树鉴别CRC、其他恶性肿瘤及正常人的准确率分别为97.1%(66/68)、93.3%(140/150)、95.5%(64/67), 总准确率94.7%.
用Biomarker Wizard软件对两组患者血清中的低分子蛋白质指纹谱数据进行对比分析, 共发现差异蛋白质峰114个(P<0.05), 其中在CRC中高表达的有12种, 低表达的有102种.
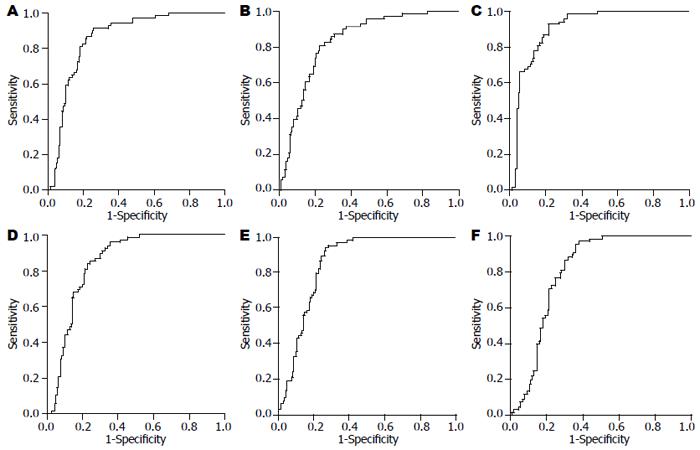
对114个差异蛋白质分别用ROC曲线进行评价, AUC>0.5且其95%可信限下限>0.5的蛋白共有75个, 其中AUC>0.8以上的有6个, M/Z分别为8911、8919、8964、11726、14049、14139(表1, 图1).
| 标志蛋白(M/Z) | 相对含量 | P值 | AUC (95%CI) | |
| 其他肿瘤 | 结直肠癌 | |||
| 8911 | 0.648 | 4.577 | 0.000 | 0.859 (0.808, 0.909) |
| 8919 | 1.423 | 7.896 | 0.000 | 0.829 (0.773, 0.884) |
| 8964 | 0.441 | 2.867 | 0.000 | 0.909 (0.870, 0.948) |
| 11726 | 0.717 | 3.456 | 0.000 | 0.849 (0.798, 0.899) |
| 14049 | 0.225 | 1.500 | 0.000 | 0.853 (0.804, 0.902) |
| 14139 | 0.173 | 1.033 | 0.000 | 0.804 (0.747, 0.861) |
以AUC>0.8的蛋白建立的Fisher判别函数如下: CRC = -1.244-0.01X1+0.054X2+0.005X3+0.062X4-0.002X5+0.198X6; 其他恶性肿瘤 = -2.16+0.015X1+0.188X2+0.132X3+0.225X4+0.468X4+0.46X5. 其中X1、X2、X3、X4、X5、X6分别代表8911、8919、8964、11726、14049、14139 M/Z蛋白的相对含量. 进行判别分析时, 将每种蛋白的相对含量分别代入上述2个判别函数中求值, 哪一个判别函数值较大, 则该观察单位就属于哪一类. 本实验中判别CRC的敏感性88.2%(60/68), 特异性93.3%(140/150), 准确率91.7%(200/218).
在全球范围内, CRC的发病率在男、女性中分别位居第4和第3位, 死亡率分别位居第4和第5位[4]. 我国CRC的发病亦不容乐观[5], 严重威胁着人们的身心健康. 患者如获得早期诊断和治疗, 5年生存率将达到90%[6]. 遗憾的是, 大部分患者在确诊时已处于进展期或已有转移, 错过了极佳的治疗窗口期. CRC早期诊断方法如直肠指诊、粪潜血试验、肠黏膜脱落细胞学检查及影像学检查等均不同程度地存在敏感性与特异性不高的问题, 而合适的肿瘤标志物将有利于癌症的检出. 目前临床应用较多的CRC相关性肿瘤标志物有CEA、CA19-9和CA242等, 用于手术前后的对比、估计预后, 其中CEA作为CRC的标志物应用最广泛, 有较高的特异性(93.5%), 但敏感性仅为47.3%[7].
研究发现, 肿瘤在发生、发展过程中伴随了一系列细胞及亚细胞水平的变化, 包括蛋白质结构和功能的改变. 人血清中的蛋白质相当丰富并不断变化, 单一的血清肿瘤标志物难以真正反溯到相关的细胞、组织, 而发现一组与特定组织癌变相关的血清蛋白质则是更有效的途径, 这也就是所谓的"癌症指纹". 因此, 研究人员正从过去的用"单一标志物"诊断肿瘤的模式向"多种标志物联合应用"的诊断模式过渡, 这不仅更加适合蛋白质组学高通量的特点, 而且也为肿瘤的诊断提供了更多的思路和机会[8]. 高通量SELDI蛋白质芯片技术的出现加速了开展发现"癌症指纹"的研究[9]. 该技术是蛋白质芯片技术与质谱技术相结合的产物, 自Adam et al[10]、Petricoin et al[11]将其应用于前列腺癌、卵巢癌的研究以来, 已被广泛应用于临床常见恶性肿瘤[12]的研究. 我们也利用该技术, 从血清蛋白质组学分析入手, 建立了CRC早期诊断模型及对中晚期CRC进行Dukes分期的分类树模型[1-3]. 这几种诊断模型的联合应用可将术前CRC患者进行准确分期, 为临床治疗提供决策依据.
目前广泛开展的肿瘤血清蛋白质组学研究, 大多将与某种肿瘤相关的良性病和正常人作对照, 而将其他恶性肿瘤纳入对照进行的研究不多. Engwegen et al[6]应用CM10芯片分析了77例CRC患者与80例正常人的血清蛋白质谱, 同时选取38例其他系统的恶性肿瘤(包括乳腺癌、卵巢癌、前列腺癌及非小细胞肺癌)标本作对照, 建立的分类树模型, 预测CRC的敏感性与特异性为65%-90%. 本研究我们在前期工作的基础上, 将临床上其他常见的恶性肿瘤(乳腺癌、食管癌、肝癌、胃癌、肺癌、肾癌)作为对照, 并以健康人作正常对照, 建立的决策分类树鉴别CRC、其他恶性肿瘤及正常人的准确率分别为97.1%、93.3%、95.5%. 进而筛选出CRC与其他恶性肿瘤的血清低分子差异表达蛋白, 应用ROC曲线分析筛选出的AUC>0.8的蛋白8911、8919、8964、11726、14049、14139 M/Z, 以此6种蛋白建立Fisher判别模型. 最终结果显示, 建立的模型鉴别CRC与其他恶性肿瘤的敏感性88.2%, 特异性93.3%, 准确率为91.7%.
由于SELDI技术本身具有较强的操作依赖性, 实验结果可因样品来源和处理条件的不一致或操作过程的非标准化, 造成实验结果批次之间或同类样本在不同检测单位之间的平行性与可重复性较差, 使实验结果的可信度遭到质疑[13-14]. 我们在实验中严格规定了实验操作流程, 对样品的存储条件、保存时间、冻融的次数、芯片和基质的选择以及检测参数的设置等各个环节都进行了规范, 建立了SELDI技术的标准化平台. 本实验中我们用同一个正常人的血清作为质控血清, 对SELDI-TOF-MS技术的稳定性进行评价, 结果表明, 我们建立的芯片检测方法具有较好的稳定性.
总之, 本实验通过蛋白质芯片技术发现了CRC与其他恶性肿瘤患者血清中的低分子差异表达蛋白, 利用这些蛋白建立的判别分析模型具有较好的鉴别诊断能力, 今后还需要用大样本量的研究来证实. 同时, 这些标志性差异蛋白可能在CRC的发生发展中起到了重要作用, 对这些差异表达蛋白的鉴定也正在进行中.
近年来结直肠癌的诊断、治疗取得了一些长足的进展, 但总体疗效并无明显改善, 术后5年生存率在50%左右, 早期诊断率还很低, 缺乏灵敏度高、特异性强的早期诊断方法. SELDI-TOF-MS将蛋白质芯片与质谱技术相结合, 具有高通量、高灵敏度等优势, 能检测出传统方法检测不到的蛋白质或多肽, 已成为肿瘤蛋白质组学主要技术之一, 为肿瘤的早期诊断与鉴别研究提供了理想的平台.
何超, 教授, 浙江大学医学院附属邵逸夫医院肛肠外科; 刘连新, 教授, 哈尔滨医科大学第一临床医学院普通外科三病房
应用SELDI-TOF-MS技术进行比较蛋白质组学研究已成为疾病蛋白质组学研究的热点, 不同的研究小组建立了多种疾病的血清或组织蛋白质指纹谱诊断模型, 发现了一些极富意义的低分子量蛋白或多肽, 由于其在血清中的含量极微, 对这些蛋白的鉴定还存在相当的困难.
Engwegen et al分析了结直肠癌患者与正常人的血清蛋白质谱, 并以其他恶性肿瘤(乳腺癌、卵巢癌、前列腺癌及非小细胞肺癌)作对照, 建立的分类树模型预测结直肠癌的敏感性与特异性为65%-90%, 鉴定出了载脂蛋白C-I、载脂蛋白A-I及白蛋白氨基末端片段.
SELDI-TOF-MS检测并经ROC曲线分析得到的6个血清低分子差异表达蛋白建立的判别分析模型具有较好的鉴别诊断能力, 这些标志性差异蛋白可能在结直肠癌的发生发展中起到了重要作用.
本文目的及结论明确, 实验方法较为先进, 统计分析结果较可靠, 参考文献引用恰当而充分, 具有较好的学术价值.
编辑: 李军亮 电编:何基才
| 1. | Zhao G, Gao CF, Song GY, Li DH, Wang XL. [Identification of colorectal cancer using proteomic patterns in serum]. Ai Zheng. 2004;23:614-618. [PubMed] |
| 4. | Parkin DM, Bray F, Ferlay J, Pisani P. Global cancer statistics, 2002. CA Cancer J Clin. 2005;55:74-108. [PubMed] [DOI] |
| 6. | Engwegen JY, Helgason HH, Cats A, Harris N, Bonfrer JM, Schellens JH, Beijnen JH. Identification of serum proteins discriminating colorectal cancer patients and healthy controls using surface-enhanced laser desorption ionisation-time of flight mass spectrometry. World J Gastroenterol. 2006;12:1536-1544. [PubMed] [DOI] |
| 7. | Chen YD, Zheng S, Yu JK, Hu X. Artificial neural networks analysis of surface-enhanced laser desorption/ionization mass spectra of serum protein pattern distinguishes colorectal cancer from healthy population. Clin Cancer Res. 2004;10:8380-8385. [PubMed] [DOI] |
| 8. | Zolg W. The proteomic search for diagnostic biomarkers: lost in translation? Mol Cell Proteomics. 2006;5:1720-1726. [PubMed] [DOI] |
| 9. | Powell K. Proteomics delivers on promise of cancer biomarkers. Nat Med. 2003;9:980. [PubMed] [DOI] |
| 10. | Adam BL, Qu Y, Davis JW, Ward MD, Clements MA, Cazares LH, Semmes OJ, Schellhammer PF, Yasui Y, Feng Z. Serum protein fingerprinting coupled with a pattern-matching algorithm distinguishes prostate cancer from benign prostate hyperplasia and healthy men. Cancer Res. 2002;62:3609-3614. [PubMed] |
| 11. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. |
| 12. | Azad NS, Rasool N, Annunziata CM, Minasian L, Whiteley G, Kohn EC. Proteomics in clinical trials and practice: present uses and future promise. Mol Cell Proteomics. 2006;5:1819-1829. [PubMed] [DOI] |
| 13. | Engwegen JY, Gast MC, Schellens JH, Beijnen JH. Clinical proteomics: searching for better tumour markers with SELDI-TOF mass spectrometry. Trends Pharmacol Sci. 2006;27:251-259. [PubMed] [DOI] |
| 14. | Poon TC. Opportunities and limitations of SELDI-TOF-MS in biomedical research: practical advices. Expert Rev Proteomics. 2007;4:51-65. [PubMed] [DOI] |