修回日期: 2009-01-04
接受日期: 2009-01-05
在线出版日期: 2009-02-18
目的: 应用酵母双杂交技术筛选人胰腺cDNA文库中与HCV 1b亚型非结构蛋白NS3相互作用的结合蛋白的编码基因, 为进一步研究HCV影响糖、脂代谢机制奠定实验基础.
方法: 扩增人胰腺cDNA文库并纯化鉴定, 纯化后的文库质粒转化酵母菌Y187; 构建诱饵质粒PGBKT7-NS3并转化酵母菌AH109, 在色氨酸缺陷型培养基(SD/-Trp)上筛选阳性菌落. 应用酵母双杂交系统3将阳性重组AH109菌株与重组酵母菌株Y187进行配合, 在四缺培养基(SD/-Trp/-Leu/-His/-Ade)和铺有X-α-gal的四缺培养基上进行筛选, 提取蓝色酵母菌落质粒, 电转化大肠埃希菌DH5α后提取质粒测序, 对测序结果进行序列比对和分析.
结果: 成功构建人胰腺cDNA文库和pGBKT7-NS3重组质粒; 将pGBKT7-NS3质粒转化AH109酵母菌株后, 并从人胰腺cDNA文库中筛选出11种与HCV NS3蛋白相结合的蛋白基因.
结论: 筛选出的与HCV NS3蛋白结合的胰腺蛋白基因中, 部分与2型糖尿病、肝脏脂肪变性密切相关.
引文著录: 胡传翠, 张锦前, 王琦, 李国力, 李朝品, 成军. 酵母双杂交技术筛选人胰腺细胞中HCV 1b NS3结合蛋白基因. 世界华人消化杂志 2009; 17(5): 454-458
Revised: January 4, 2009
Accepted: January 5, 2009
Published online: February 18, 2009
AIM: To screen proteins in pancreas cDNA library interacting with HCV 1b nonstructural protein NS3 and to study the biological functions of binding proteins.
METHODS: The pancreas cDNA library was amplified, purified and evaluated, and then the purified library plasmids were transformed into yeast strain Y187. The reconstructed plasmid pGBKT7-E1 was transformed into yeast strain AH109 and screened on the nutrient deficiency medium SD/-Trp. The transformed AH109 was mated with Y187 containing the library plasmid. The diploid yeast cells were plated on nutrient deficiency medium SD/-Trp/-Leu/-His/-Ade and SD/-Trp/-Leu/-His/-Ade containing X-α-gal for selection. The plasmids in diploid yeast cells were extracted and electrotransformed into E.coli DH5α. The plasmids in DH5α were extracted, sequenced and analyzed using bioinformatic methods.
RESULTS: The human pancreas cDNA library and bait plasmid (pGBKT7-NS3) were constructed successfully. The reconstructed bait plasmid was transformed into yeast cells AH109 successfully. 11 proteins interacting with HCV NS3 were screened.
CONCLUSION: These results show that HCV NS3 protein may be related with metabolism of glucose and lipid.
- Citation: Hu CC, Zhang JQ, Wang Q, Li GL, Li CP, Cheng J. Screening of proteins binding to HCV 1b NS3 protein from human pancreas cDNA library by yeast two-hybrid system. Shijie Huaren Xiaohua Zazhi 2009; 17(5): 454-458
- URL: https://www.wjgnet.com/1009-3079/full/v17/i5/454.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i5.454
丙型肝炎病毒(hepatitis C virus, HCV)的感染已呈全球流行趋势, 椐WHO报道, 全球约有1.7亿感染者(感染率约3%), 在我国约有4100万感染者(感染率为3.2%), 其中主要以1b亚型感染为主[1-2]. HCV患者易转变为慢性感染过程, 可出现肝脏慢性炎症坏死、脂肪病变及纤维化, 部分患者可发展为肝硬化甚至肝细胞癌. 目前已有研究表明, 肝脏脂肪变性是慢性丙型肝炎的发病机制之一, 这充分提示我们, 慢性丙型肝炎也是一种代谢性疾病[3], 代谢异常是HCV致病机制的重要组成部分. 而且近年来, 流行病学调查表明HCV慢性感染常伴有糖、脂肪代谢性疾病, 并已经证实HCV慢性感染与糖尿病和脂肪肝等代谢性疾病的发生密切相关[4-5], 但确切机制尚不明确.
HCV基因组全长约9.6 kb, 编码一个多聚蛋白, 根据不同功能可分为结构蛋白和非结构蛋白. 其中, 非结构蛋白3(non-structure protein 3, NS3)主要具有丝氨酸蛋白酶功能, 而丝氨酸蛋白酶在人体代谢方面起着极其重要的作用. 因此本文中, 我们应用酵母双杂交技术筛选人胰腺细胞cDNA文库中的HCV 1b亚型NS3的结合蛋白基因, 以便为进一步研究HCV慢性感染影响糖、脂类代谢的分子生物学机制提供一定的思路和方向.
酵母双杂交系统试剂盒包括人类胰腺cDNA文库、pGADT7-AD克隆载体、pGBKT7-BD克隆载体、pGADT7-T对照质粒、pGBKT7-53对照质粒、pGBKT7诱饵质粒、酵母菌株AH109、酵母菌株Y187, 购自Clontech公司. 限制性核酸内切酶购自TaKaRa(大连)公司. Taq酶、T4 DNA连接酶购于Promega公司. 质粒pCon1 SG-Neo和大肠埃希杆菌(DH5α)为本室保存. 胰蛋白胨、酵母提取物为OXOID公司产品, X-α-Gal、酵母YPD培养基、酵母YPDAgar培养基、SD/-Trp、SD/-Leu、SD/-Trp/-Leu、SD/-Trp/-Leu/-His以及SD/-Trp/-Leu/-His/-Ade培养基均购自Clontech公司. 鲑精DNA(DNA, from herring sperm)、半硫酸腺苷、醋酸锂均购自Sigma公司. 引物合成及DNA测序由奥科公司完成.
1.2.1 酵母胰腺cDNA文库的构建: 按Clontech公司的说明书对文库进行扩增, 用Qiagen公司的操作手册(Qiagen plasmid Maxi kit)对文库进行纯化. 取纯化的文库质粒转化E.coli DH5α感受态细胞, 随机挑取6个单克隆提取质粒, EcoRⅠ、XhoⅠ双酶切鉴定cDNA文库的多样性. 然后将该文库转化酵母, 制备酵母胰腺cDNA文库. 提取新鲜生长的Y187酵母菌落(直径>2 mm)接种于150 mL YPDA液体培养基中, 30℃振摇至A600>1.5时, 把该培养物转摇至850 mL YPDA液体培养基中, 30℃振摇至A600 = 0.5左右. 以醋酸锂法将文库质粒(0.25 mg)转化入酵母细胞Y187进行文库转化, 然后将转化物铺SD/-Leu板50块, 30℃倒置孵育7 d. 同时将转化产物按1∶10, 1∶102, 1∶1×104, 1∶1×106, 1∶1×108分别铺于SD/-Leu培养基上检验转化效率.
1.2.2 诱饵质粒的构建及鉴定: 根据GenBank中HCV 1b NS3基因序列(GI: 20 384 690), 在编码区的上游和下游设计合成一对特异性引物. 正链引物为5'-CCGGAATTCGCGCCCATCACGGCCTACGCCCAACAGACG-3', 负链引物为5'-CGCGGATCCTGTGACGACCTCCAGGTCAGCCGACATGC-3', 引物两端分别引入EcoRⅠ、BamHⅠ酶切位点. 以质粒pCon1 SG-Neo为模板, 用上述引物进行PCR扩增. PCR反应参数如下: 94℃预变性5 min; 94℃变性40 s, 63℃退火40 s, 72℃延伸90 s, 共35个循环; 72℃延伸10 min. 10 g/L琼脂糖凝胶电泳鉴定PCR产物, 玻璃奶回收. 将回收的目的基因片段与pGBKT7载体16℃连接过夜, 转化DH5α感受态细胞, 铺于卡那霉素LB琼脂平板上进行抗生素筛选. 12 h后, 挑数个克隆分别进行菌落PCR. 对PCR阳性的克隆进行活化培养, 碱裂解法提取质粒DNA, 用EcoRⅠ、BamHⅠ内切酶进行双酶切验证. 证明目的基因片段已插入pGBKT7载体中, 阳性克隆送奥科生物公司测序. 醋酸锂法将重组质粒pGBKT7-HCV 1b NS3转入酵母菌株AH109. 转化后铺板于SD/-Trp固体培养基进行筛选, 对直径>2 mm的菌落随机挑取进行菌落PCR鉴定.
1.2.3 诱饵与胰腺文库的酵母配合: 挑取新鲜生长的经菌落PCR鉴定正确的AH109酵母菌落(直径>2 mm)接种于SD/-Trp液体培养基中, 30℃振摇至A600为0.8-1.0时, 离心并重悬于50 mL 2×YPDA培养基中, 与4 mL含胰腺文库的酵母细胞于30℃ 30-45 r/min配合24 h. 观察到三叶草状的二倍体细胞后, 将其离心并重悬于10 mL 0.25×YPDA培养基中, 然后铺SD/-Trp/-Leu/-His平板和SD/-Trp/-Leu/-His/-Ade平板各25块, 置30℃孵箱培养. 同时将配合产物按1∶10, 1∶100, 1∶1000分别铺于SD/-Trp/-Leu、SD/-Trp、SD/-Leu培养基上检验配合效率. 生长16 d后把直径>2 mm的菌落划线于铺有X-α-Gal的SD/-Trp/-Leu/-His/-Ade培养基上, 在此培养基上生长且变成蓝色的为阳性菌落.
1.2.4 阳性质粒的分析: 于SD/-Trp/-Leu/-His/-Ade液体培养基上培养挑取的阳性菌落, 30℃ 250 r/min振摇5 d后用酸化玻璃珠法提取酵母质粒. 电穿孔法将提取的质粒转化大肠杆菌并于含有氨苄青霉素的LB平板上培养, 所获得菌落质粒行BglⅡ酶切鉴定后测序, 测序后与GenBank中的生物信息进行对照分析.
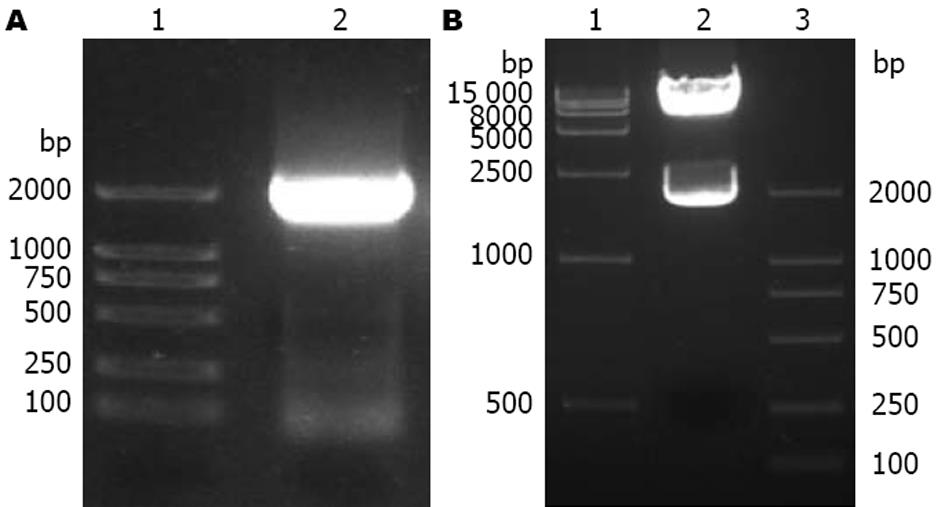
经测定, 待转化的胰腺cDNA文库滴度在4×107左右, 纯化后的的质粒DNA浓度约在0.34 g/L. 用EcoRⅠ, XhoⅠ双酶切鉴定cDNA文库的多样性(图1). 结果显示插入片断大小不一, 说明文库具有良好的多样性. 经转化效率的检验, 确定文库细胞计数大于1×109细胞/L, 证明胰腺cDNA文库转化成功.
以质粒pCon1 SG-Neo为模板, 经过PCR扩增获得预期的1893 bp的基因片段, 且无非特异性扩增现象(图2A). HCV 1b NS3的PCR扩增产物连接至pGBKT7载体相应的酶切位点上, EcoRⅠ、BamHⅠ双酶切鉴定正确后, 经测序鉴定表明诱饵质粒pGBKT7-HCV 1b NS3构建成功(图2B).
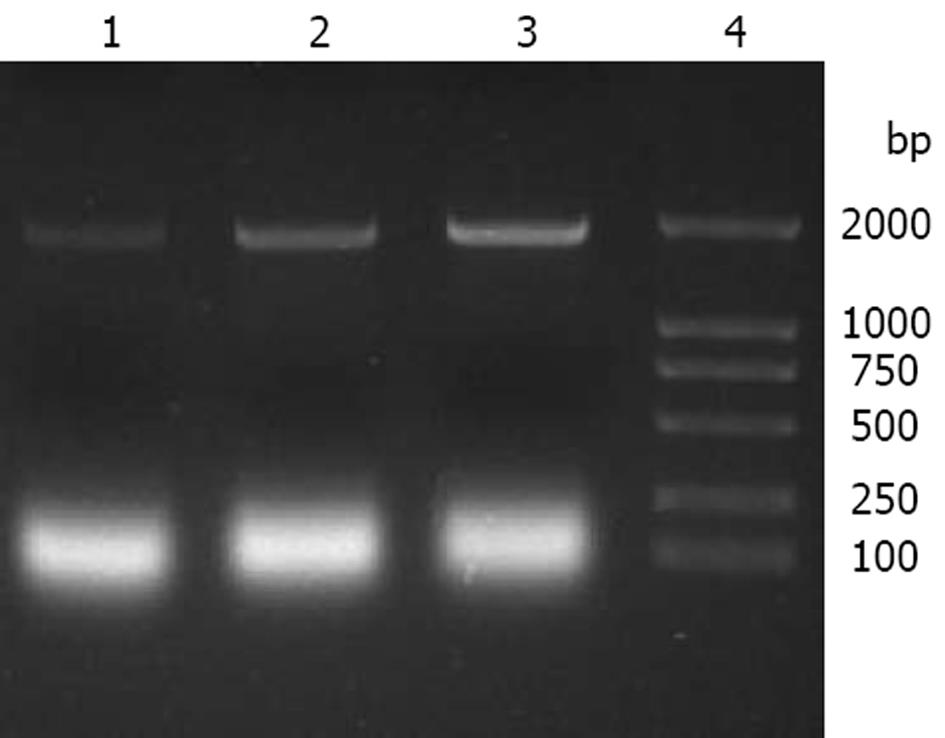
用醋酸锂法转化酵母细胞后在缺陷型SD/-Trp培养基上筛选生长. 培养4 d后, 挑取菌落进行PCR扩增HCV 1b NS3基因, 结果显示转化成功(图3).
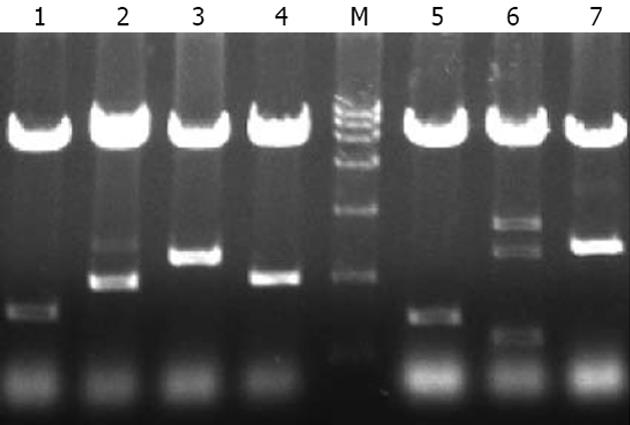
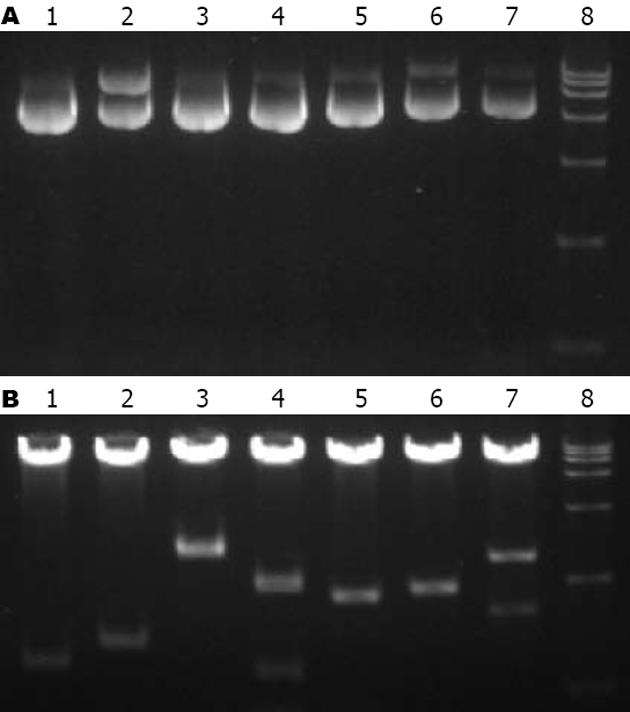
筛选获得的阳性克隆用酸化玻璃珠法提取酵母质粒, 琼脂糖凝胶电泳后可见明显质粒带(图4A). pACT2载体上含有的两个BglⅡ酶切位点分别位于多克隆位点两侧, 故使用该内切酶消化将释放出所筛选到的胰腺文库的基因片段. 图4B所示BglⅡ酶切、琼脂糖凝胶电泳后的不同大小胰腺cDNA文库片段.
挑选23个克隆测序后, 与GenBank数据库进行BLAST同源序列比对, 如表1所示.
| 序号 | 已知的同源序列编码蛋白基因 | GenBank序号 | 相同克隆数 | 同源性(%) |
| 1 | 人类胰凝乳蛋白酶原B1 | NM 001906.4 | 2 | 100 |
| 2 | 人类羧肽酶A1 | NM 001868.1 | 2 | 95 |
| 3 | 人类胰凝乳蛋白酶原B2 | NM 001025200.3 | 1 | 94 |
| 4 | 人类胰蛋白酶2 | NM 002769.2 | 1 | 98 |
| 5 | 人类羧肽酶B1 | NM 001871.2 | 7 | 99 |
| 6 | Homo sapiens chromosome 12 genomic contig alternat assembly | NW 001838052.1 | 1 | 94 |
| 7 | 人类淀粉酶α2B | NM 020978.3 | 1 | 98 |
| 8 | 人类胰脂肪酶 | NM 000936.2 | 1 | 99 |
| 9 | 人类v-Ki-ras2鼠Kirsten肉瘤病毒致癌基因同源物 | NM 033360.2 | 2 | 99 |
| 10 | 人类弹性蛋白酶2A | NM 033440.1 | 4 | 100 |
| 11 | 人类弹性蛋白酶3A | NM 005747.3 | 1 | 98 |
自1989年发现HCV以来, 人们就通过研究发现, HCV感染除引起肝脏损害并与肝细胞癌有相关性外, 还与肝外组织的损害表现密切相关, 其中一个最主要的表现就是代谢性疾病[6-8]. 代谢综合征(metabolic syndrome, MS)是多种病态组合现象的一个名称, 其主要特征是肥胖、糖代谢紊乱和高血压, 主要病理生理基础是胰岛素抵抗(insulin resistance, IR). 国内外的大量研究表明, 慢性丙型肝炎病毒感染患者常伴糖、脂代谢紊乱, 很可能与IR有关, 而糖、脂代谢紊乱又加重IR, 三者关系密切复杂. 因此IR亦可能是肝炎病毒感染相关的代谢性疾病发病机制的中心环节. 研究还发现, IR是肝纤维化进展的相关因素, 并影响HCV抗病毒治疗的疗效[9-10], 另外, 糖尿病可能增加肝炎病毒致癌的作用[11].
HCV与脂蛋白、载脂蛋白等之间的结合和相互作用可能是干扰肝细胞中脂肪代谢正常通路, 引起HCV慢性感染肝脂肪变的机制之一. 早在1992年, Thomssen et al[12]就发现含有HCV RNA的病毒颗粒密度差别显著, 且与血清中的β脂蛋白有关. 1995年人们[13]注意到慢性丙型肝炎患者及感染了HCV的大猩猩肝活检标本中, 脂肪变性是其一大特征. 从而认识到丙型肝炎病毒可能干扰了肝脏的脂肪代谢. 王琳 et al[14]运用酵母双杂交技术证实HCV核心蛋白可以与肝细胞中的载脂蛋白Apo-A1结合, 他们的结合可能会影响Apo-A1的结构以及其脂质转运的功能, 进而影响脂类的代谢过程. 同时建立了HCV结构蛋白的转基因小鼠模型, 发现小鼠肝脏有典型的脂肪变病理改变, 再次证实了HCV感染与代谢性疾病的密切关系[15]. 但是, 关于HCV引起代谢性疾病的分子生物学机制还有许多环节没有阐明. 考虑到HCV NS3的丝氨酸蛋白酶功能, 同时介于我国HCV感染以1b型为多的实际情况, 我们选择以HCV 1b NS3为诱饵蛋白, 应用酵母双杂交系统3从正常人胰腺细胞cDNA文库中筛选与其存在相互作用的蛋白基因, 寻找其中与糖、脂类代谢相关的蛋白基因, 为相关的机制研究奠定基础.
本实验中将胰腺细胞cDNA文库质粒扩增后成功转化酵母菌株Y187, 构建成功酵母胰腺细胞cDNA文库, 以备进一步更广泛的酵母双杂交筛选工作使用. 本研究所成功构建了pGBKT7-HCV NS3诱饵质粒并在酵母菌株AH109中表达出HCV NS3蛋白, 并进一步与前述构建好的酵母胰腺细胞cDNA文库进行配合后, 筛选出可能与HCV NS3蛋白存在相互作用的已知胰腺细胞蛋白基因11种. 这些蛋白以参与三大物质消化、吸收的酶类为主, 其中参与糖、脂类代谢的酶主要有弹性蛋白酶、胰蛋白酶(原)、胰脂肪酶和胰淀粉酶等. 弹性蛋白酶可以影响脂质代谢、降低胆固醇、增强肝中脂肪分解并能抑制动脉硬化, 此外还可抑制糖尿病发展[16]; 胰蛋白酶可以引起胰腺其他酶类物质的分泌; 胰脂肪酶主要通过分解食物中的甘油三酯参与对食物脂肪的消化和吸收; 胰淀粉酶通过对淀粉的分解消化影响糖代谢.
另外在本实验中, 我们还发现人类v-Ki-ras2鼠Kirsten肉瘤病毒致癌基因同源物(KRAS)蛋白也可以与HCV NS3蛋白相互作用, KRAS在人类正常组织的增殖、分化和衰亡中有着非常重要的作用. 研究表明, KRAS是活性最强的癌基因之一, 其突变株与17%-25%的人类肿瘤有关. 可能是由于这些激活的RAS突变株可以通过降低内在GTP酶活性和增强对GTP酶激活蛋白的抗性, 导致RAS大量聚集在活化GTP结合的部位[17]. 这可能为HCV感染导致肝细胞肿瘤的发病机制提供了研究方向.
通过前述的实验研究, 我们推测HCV NS3可能与胰腺细胞中的蛋白结合后进一步导致IR和糖、脂类代谢异常, 最终发展为2型糖尿病、脂肪肝、MS等代谢性疾病. 我们此次应用经典的验证蛋白质-蛋白质相互作用的酵母双杂交方法, 首次在胰腺细胞cDNA文库中筛选出HCV NS3结合蛋白, 为研究慢性丙型肝炎患者易出现糖、脂代谢异常及伴发代谢性疾病的机制提供了研究依据和方向, 为临床工作中相关疾病的预防及诊治提供了新的思路.
慢性丙型肝炎也是一种代谢性疾病, 代谢异常是HCV致病机制的重要组成部分. HCV基因组全长约9.6 kb, 编码一个多聚蛋白, 根据不同功能可分为结构蛋白和非结构蛋白. 其中, 非结构蛋白3(NS3)主要具有丝氨酸蛋白酶功能, 而丝氨酸蛋白酶在人体代谢方面起着极其重要的作用.
管冬元, 副教授, 上海中医药大学基础医学院实验中医学教研室; 李淑德, 主任医师, 中国人民解放军第二军医大学长海医院消化内科.
研发前沿 流行病学调查表明HCV慢性感染常伴有糖、脂肪代谢性疾病, 并已经证实HCV慢性感染与糖尿病和脂肪肝等代谢性疾病的发生密切相关, 但确切机制尚不明确.
王琳 et al运用酵母双杂交技术证实HCV核心蛋白可以与肝细胞中的载脂蛋白Apo-A1结合, 他们的结合可能会影响Apo-A1的结构以及其脂质转运的功能, 进而影响脂类的代谢过程.
应用经典的验证蛋白质-蛋白质相互作用的酵母双杂交方法, 首次在胰腺细胞cDNA文库中筛选出HCV NS3结合蛋白.
本研究设计严整, 思路清晰, 文字表达合理, 具有较强的创新性、较高的学术价值与较强的临床指导意义.
编辑: 史景红 电编:何基才
| 1. | 陈 云茹, 刘 敏, 陈 天艳, 张 树林. 丙型肝炎病毒核心蛋白引发肝脂肪变性及其分子学机制. 国际流行病学传染病学杂志. 2006;33:327-329. |
| 2. | Demetriou VL, van de Vijver DA, Kostrikis LG. Molecular epidemiology of hepatitis C infection in Cyprus: evidence of polyphyletic infection. J Med Virol. 2009;81:238-248. [PubMed] [DOI] |
| 3. | Koike K. Hepatitis C as a metabolic disease: Implication for the pathogenesis of NASH. Hepatol Res. 2005;33:145-150. [PubMed] [DOI] |
| 4. | Jármay K, Karácsony G, Nagy A, Schaff Z. Changes in lipid metabolism in chronic hepatitis C. World J Gastroenterol. 2005;11:6422-6428. [PubMed] |
| 5. | Wang CS, Wang ST, Yao WJ, Chang TT, Chou P. Hepatitis C virus infection and the development of type 2 diabetes in a community-based longitudinal study. Am J Epidemiol. 2007;166:196-203. [PubMed] [DOI] |
| 6. | Aksõz K, Unsal B, Kirci A, Alper E, Buyraç Z, Aslan F, Cekiç C, Cengiz O, Ozcan Ari F, Akpinar Z. The relationship between chronic HCV infection and the level of plasma adiponectin. Turk J Gastroenterol. 2008;19:254-257. [PubMed] |
| 7. | Rafiq N, Younossi ZM. Interaction of metabolic syndrome, nonalcoholic fatty liver disease and chronic hepatitis C. Expert Rev Gastroenterol Hepatol. 2008;2:207-215. [PubMed] [DOI] |
| 8. | Slama L, Le Camus C, Serfaty L, Pialoux G, Capeau J, Gharakhanian S. Metabolic disorders and chronic viral disease: The case of HIV and HCV. Diabetes Metab. 2008; Nov 28. [Epub ahead of print]. [PubMed] |
| 9. | Reaven GM. Banting lecture 1988. Role of insulin resistance in human disease. Diabetes. 1988;37:1595-1607. [PubMed] [DOI] |
| 11. | Locarnini S. Molecular pathogenesis of viral hepatitis. J Gastroenterol Hepatol. 2000;15:546-548. [PubMed] [DOI] |
| 12. | Thomssen R, Bonk S, Propfe C, Heermann KH, Köchel HG, Uy A. Association of hepatitis C virus in human sera with beta-lipoprotein. Med Microbiol Immunol. 1992;181:293-300. [PubMed] [DOI] |
| 13. | Goodman ZD, Ishak KG. Histopathology of hepatitis C virus infection. Semin Liver Dis. 1995;15:70-81. [PubMed] [DOI] |
| 15. | 成 军, 任 进余, 李 莉, 陆 志檬, 李 克, 洪 源, 陆 荫英, 王 刚, 刘 妍, 张 玲霞. 丙型肝炎病毒结构基因转基因小鼠引起肝脏脂肪变. 世界华人消化杂志. 2002;10:1022-1026. [DOI] |
| 16. | Thongboonkerd V, Barati MT, McLeish KR, Benarafa C, Remold-O'Donnell E, Zheng S, Rovin BH, Pierce WM, Epstein PN, Klein JB. Alterations in the renal elastin-elastase system in type 1 diabetic nephropathy identified by proteomic analysis. J Am Soc Nephrol. 2004;15:650-662. [PubMed] [DOI] |
| 17. | Kranenburg O. The KRAS oncogene: past, present, and future. Biochim Biophys Acta. 2005;1756:81-82. [PubMed] |