修回日期: 2008-12-06
接受日期: 2008-12-08
在线出版日期: 2009-01-08
目的: 分析结直肠癌期患者血清蛋白质组变化, 初步建立结直肠癌期分类树模型.
方法: 将335例血清样本(其中结直肠癌患者169例, 健康人166例)随机分为训练组和测试组, 将血清样本加至IMAC30-Cu2+蛋白芯片, 利用SELDI-TOF-MS得到血清蛋白质谱, 利用Biomarker Wizard软件进行蛋白峰值鉴定和聚类. 利用Biomarker Pattern以训练组建立由1个差异蛋白组成的结直肠癌期分类树模型, 以测试组进行独立样本的双盲验证. 另外, 应用电化学发光免疫测定法检测测试组血清样本CEA.
结果: 软件识别了59个质峰, 其中由质荷比为5765的蛋白构成的分类树模型可以有效鉴别结直肠癌患者与正常人, 灵敏度和特异度分别是98.81%及100.00%, 经双盲验证其灵敏度, 特异度及阳性预测值分别是97.65%, 98.80%及98.81%. CEA的灵敏度及特异度低于SELDI分类树模型(P<0.05).
结论: SELDI-TOF-MS检测得到的血清蛋白质组分类树模型可以准确的鉴别结直肠癌患者与正常人, 对结直肠癌的筛查有重要的意义.
引文著录: 范乃军, 高春芳, 王秀丽, 盛新华, 李冬晖, 郑国宝. 应用SELDI-TOF-MS技术初步建立结直肠癌分类树模型. 世界华人消化杂志 2009; 17(1): 53-58
Revised: December 6, 2008
Accepted: December 8, 2008
Published online: January 8, 2009
AIM: To establish a serum protein fingerprinting technique coupled with a pattern-matching algorithm to distinguish colorectal cancer from healthy individuals.
METHODS: Serum samples were applied to metal affinity protein chips to generate mass spectra by surface-enhanced laser desorption/ionization time-of-flight mass spectrometry (SELDI-TOF-MS). Protein peak identification and clustering were performed using the Biomarker Wizard software. Proteomic spectra of serum samples from 84 colorectal cancer patients and age- and sex-matched 83 healthy individuals were used as a training set and a classification tree with 1 distinct protein masses was generated by using Biomarker Pattern software. The validity of the classification tree was then challenged with a blind test set including another 85 colorectal cancer patients and 83 healthy individuals. We additionally determined carcinoembryonic antigen (CEA) in all the serum samples included in the blind test set using an electrochemiluminescent immunoassay.
RESULTS: The software identified an average of 59 mass peaks/spectrum and 1 of the identified peaks at 5765 was used to construct the classification tree. The classification tree separated effectively colorectal cancer from healthy individuals, with a sensitivity of 98.81% and a specificity of 100.00%. The blind test challenged the model with a sensitivity of 97.65% and a specificity of 98.80%, and a positive predictive value of 98.81%, respectively. The specificity and sensitivity provided by CEA were significantly lower than that of the SELDI marker pattern (P < 0.05).
CONCLUSION: SELDI-TOF-MS technique can correctly distinguish colorectal cancer patients from healthy individuals and shows great potential for the development of a screening test for the detection of colorectal cancer.
- Citation: Fan NJ, Gao CF, Wang XL, Sheng XH, Li DH, Zheng GB. Identification of colorectal cancer patients by serum protein profile using surface-enhanced laser desorption/ionization time-of-flight mass spectrometry. Shijie Huaren Xiaohua Zazhi 2009; 17(1): 53-58
- URL: https://www.wjgnet.com/1009-3079/full/v17/i1/53.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i1.53
结直肠癌的发病率和死亡率在欧美及中国均居前列. 目前结直肠癌的诊断及治疗较30年前发生了很大的改进, 但其发病率和死亡率并未发生根本的变化, 主要原因在于缺乏高灵敏度、特异度的早期诊断方法. 表面增强激光解析离子化飞行时间质谱仪(surface-enhanced laser desorption/ionization time-of-flight mass spectrometry, SELDI-TOF-MS)是蛋白质芯片和质谱技术的结合, 在肿瘤标志物的发现和诊断方面已崭露巨大的潜力. 结直肠散发性中分化腺癌是结直肠癌的主要病理类型. 利用SELDI-TOF-MS和铜离子鳌合蛋白芯片(IMAC30-Cu2+)检测了结直肠散发性中分化腺癌患者及正常人的血清样本, 利用相关软件进行蛋白峰值鉴定和聚类, 比较结直肠癌患者与正常人血清蛋白质组构型的变化, 建立了由1个差异蛋白组成的结直肠癌血清蛋白分类树模型, 并进行独立样本的双盲验证, 可以准确的鉴别结直肠癌与正常人, 对结直肠癌的筛查有重要意义, 现报告如下.
结直肠散发性中分化腺癌患者血清标本169例来自中国人民解放军150中心院2006-10/2008-03住院患者, 健康志愿者血清标本166例来自2007年在中国人民解放军150中心院健康体检者. 取样人群有相似的暴露史, 汉族, 年龄范围为18-82岁, 结直肠癌及正常人的性别比(男/女)分别是0.97及0.98, 采用完全随机化的方法分别将样本分为训练组和测试组, 2组中结直肠癌患者及正常人分别是84/83及85/83例, 年龄、性别的差异无显著性. 结直肠癌患者均经术后病理检查, 根据AJCC的TNM分期2004确诊为结直肠中分化腺癌[1], 训练组Ⅰ、Ⅱ、Ⅲ、Ⅳ期患者各13、37、25和9例, 测试组Ⅰ、Ⅱ、Ⅲ、Ⅳ期患者各13、36、26和10例, 无家族史, 正常人经纤维结肠镜、B超、ECG、血清CEA等检查, 除外肿瘤及炎症性、代谢性等影响血清蛋白表达的相关疾病. 告知所有受试者本人或其家属该研究的目的、方法及意义, 经同意后, 签署知情同意书. 尿素、乙腈、三氟乙酸、SPA(sinapinic)、CHAPS、Tris-HCL等均购自Sigma公司, 美国Ciphergen Biosystem 公司的ProteinChip Biology System(PBSⅡ+)及其配套的IMAC30-Cu2+蛋白芯片、SELDI-TOF-MS购自美国Ciphergen Biosystem公司.
1.2.1 样品的收集: 清晨空腹采集肘静脉全血5 mL后立即放入4℃冰箱静置2 h, 4℃、1000 r/min离心30 min, 吸取血清分装成100 μL/管, 共4管, 做好标记, 置于-70℃冰箱保存, 另取正常人血标本30mL进行预实验, 处理方法同上, 吸取血清分装成每管40 μL, 共64管.
1.2.2 样品的准备及分组: 溶解冰冻血清并4℃、20 000 g离心10 min, 每个样品取20 μL血清, 分别加40 μL U9缓冲液(含9 mol/L尿素, 2% CHAPS, 50 mmol/L Tris-HCl pH9.0), 4℃、200 r/min振荡30 min, 使蛋白质变性. 将120 μL结合/洗脱缓冲液(含100 mmol/L磷酸钠, 500 mmol/L氯化钠, pH7.0, 即IMAC缓冲液)加入1.5 mL离心管, 每管加入10 μL变性后的血清样品, 4℃、200 r/min振荡30 min. 将64份预实验样本分成8组, 以8条IMAC30-Cu2+芯片检测, 评价质谱图在芯片之间及同1芯片不同芯池之间的重复性. 采用随机区组法将实验样本分为42组, 每组8例分别加至同一条芯片的8个芯池A-H(每组正常人及结直肠癌患者各4例, 第42组结直肠癌患者5例, 正常人2例).
1.2.3 芯片的预处理: 预处理方法如文献[2]所述, 主要包括: 芯片每个芯池加入100 mmol/L CuSO4 50 μL, 4℃, 200 r/min振荡5 min, 倒除CuSO4残液, 去离子水冲洗5次后甩干, 每个芯池加入100 mmol/L醋酸钠(pH4.0)50 μL, 4℃、200 r/min振荡5 min, 倒除醋酸钠残液, 去离子水冲洗5次后甩干, 每个芯池加入150 μL IMAC缓冲液, 4℃、200 r/min振荡5 min后倒除残液重复1次, 每个芯池加入50 μL稀释后的样品, 4℃、200 r/min振荡60 min后倒除残液, 以150 μL IMAC缓冲液冲洗3次, 每次4℃、200 r/min振荡5 min, 最后以1 mmol/L HEPES(pH7.0)快速冲洗; 取出芯片, 在每个芯池周围以疏水笔圈样品孔并风干; 取2 mg EAM(SPA)置于1.5 mL离心管中, 加入30 μL乙腈和30 μL 1% TFA, 室温1800 r/min振荡5 min使SPA全部溶解, 离心1 min; 每个芯池加SPA 2次, 每次0.5 μL, 2次之间允许各孔风干.
1.2.4 芯片检测: 用加有all-in-one标准蛋白质的NP20芯片校正质谱仪, 设定仪器参数, 激光强度175, 检测灵敏度8, 优化相对分子质量范围1500-20 000, 最佳聚焦中心6000, 数据采集参数范围20-80, 收集点数为100次. 在Ciphergen ProteinChip软件中设定读片程序读取芯片数据, 计算机以1×109 Hz/s的速度获得原始数据并快速精确的绘制出蛋白质谱图, 其横坐标为质荷比(M/Z), 纵坐标为蛋白质相对含量.
1.2.4.1 蛋白质的M/Z: 结合EAM后的蛋白质在SELDI-TOF-MS氦氖激光器的激光轰击下电离, 带有电荷的蛋白质在加速电场的作用下, 不同质荷比的蛋白质在一定的真空管中飞行所需时间不同, 蛋白质M/Z与离子飞行时间的平方成正比. 由E = UZ = 1/2mv2和t = L/A推出M/Z = Kt2 =(2U/L2)t2. 其中Z为离子所带电荷数, U为电压, V为飞行速度, L为加速飞行电场电压, K为常数.
1.2.4.2 蛋白质相对含量: 带有正电荷的蛋白质离子束在到达检测器的一瞬间, 电子倍增器将产生瞬时电流(瞬时电流It = Q/t, 其中Q为t时刻检测器检测到的电荷数)转换成蛋白质相对含量.
1.2.5 血清样本CEA的检测: 应用电化学发光免疫测定法(试剂盒由郑州博赛生物试剂实验研究所提供)检测测试组168例受试者血清样本的CEA, <5 μg/L诊断为正常, ≥5 μg/L诊断为结直肠癌.
统计学处理 用Ciphergen ProteinChip软件和BioMarker Wizard软件对芯片检测得到的蛋白质相对含量及蛋白质M/Z数据按数据库挖掘要求进行处理. 通过BioMarker Wizard软件用方差分析对同一M/Z对应蛋白的相对含量在不同组间的差异进行统计学分析. 用数据挖掘软件Biomarker Pattern(其采用的是决策树方法对目标对象进行分类)对数据组及相关性进行分析. SPSS13.0用于分类树模型和CEA两种诊断方法的比较、分析一般病历资料、计算预试验样本中所选取3个M/Z对应蛋白相对含量的变异系数.
用BioMarker Wizard软件对结直肠癌84例和正常人83例的质谱图进行比较分析, 共检测到有差异的蛋白质峰59个(图1), 其中差异有显著性的有54个(P<0.05, 表1).
| 差异蛋白的M/Z | 结直肠癌组的均数 | 正常人组的均数 | P | 结直肠癌组的表达变化 |
| 3901 | 0.23 | 3.42 | 0 | ↓ |
| 4105 | 4.72 | 0.50 | 0 | ↑ |
| 4224 | -0.16 | 4.11 | 0 | ↓ |
| 4499 | 6.89 | 0.88 | 0 | ↑ |
| 4943 | 6.01 | 0.29 | 0 | ↑ |
| 5765 | 38.30 | 0.39 | 0 | ↑ |
| 5785 | 14.20 | 0.32 | 0 | ↑ |
| 5921 | 0.41 | 10.87 | 0 | ↓ |
| 5970 | 6.52 | 1.93 | 0 | ↑ |
| 6289 | 5.16 | 0.75 | 0 | ↑ |
| 7398 | 3.46 | 0.20 | 0 | ↑ |
| 7596 | 40.34 | 4.82 | 0 | ↑ |
| 7617 | 13.30 | 0.92 | 0 | ↑ |
| 7638 | 4.01 | -0.03 | 0 | ↑ |
| 7662 | 3.72 | 0.74 | 0 | ↑ |
| 7718 | -0.67 | 6.84 | 0 | ↓ |
| 7966 | 10.79 | 4.27 | 0 | ↑ |
| 8761 | 3.53 | 0.67 | 0 | ↑ |
| 9118 | 9.66 | 1.54 | 0 | ↑ |
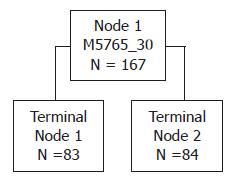
用BioMarker Wizard软件和Biomarker Pattern软件在学习模式下分析84例结直肠癌患者与83例正常人血清中蛋白质的组成, 结果发现M/Z为5765的蛋白组成的分类树模型可将结直肠癌患者与正常人准确的分组. 用此生物标志物分析数据共产生1个节点(图2): 在节点1处, 满足5765≤3.325(终节点1)诊断为正常人, 满足5765>3.325(终节点2)诊断为结直肠癌.
用Biomarker Pattern软件统计学分析结果显示: 训练组样本在学习模式下由M/Z为5765的蛋白组成的分类树模型在学习模式下可以将84例结直肠癌患者及83例健康志愿者完全正确分组, 正确率为100.00%(167/167).
训练组样本在测试模式下对上述分类树模型进行交叉验证, 结果83/84例结直肠癌患者被本方法正确诊断, 83/83例正常人被本法正确诊断, 灵敏度、特异性及约登指数(Youden's index, YI)分别为98.81%、100.00%和0.99.
另取结直肠癌患者血清标本各85例及正常人血清标本83例对分类树模型进行独立样本的双盲验证, 分类树模型可以将83/85例结直肠癌患者、82/83例正常人正确分组, 正确率、灵敏度、特异性、YI及阳性预测值分别为98.21%(165/168)、97.65%(83/85)、98.80%(82/83)、0.97和98.81%(83/84).
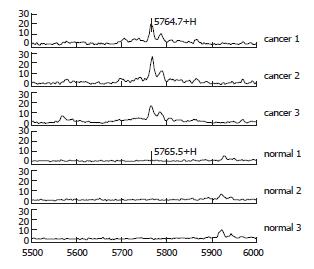
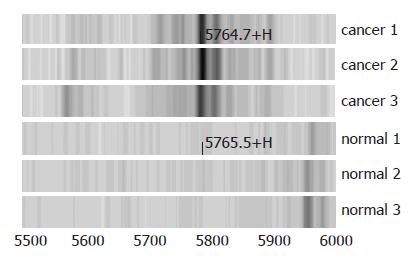
M/Z 5765蛋白峰的相对含量在结直肠组高于正常人组, 经方差分析其差异有显著性(P = 0, 图3-4).
将同一志愿者的血清分成64份, 以8条IMAC30-Cu2+芯片检测, 从质谱图中随机挑选3个波峰, 计算其在不同芯片以及同1条芯片不同芯池之间相对强度的变异系数, 小于3%, 说明在不同芯片以及同1条芯片不同芯池之间M/Z和波峰强度具有较好的重复性.
采用郑州博赛生物试剂实验研究所提供的试剂盒通过电化学发光免疫测定法检测了测试组168例样本的CEA, 正常参考值: <5 μg/L, 34/85例结直肠癌患者、30/83例正常人被正确诊断, 灵敏度、特异性别为40%、36.14%, 阳性检测率低于结直肠癌分类树模型(χ2 = 10.7, P<0.005).
结直肠癌是常见恶性肿瘤之一, 5年生存率随肿瘤分期的增高而显著降低[3-5], 对不具有高危因素的结直肠癌患者在50岁前后切除而具有高危因素的结直肠癌患者在50岁之前切除可以显著降低结直肠癌的新发生率及死亡率[6]. 因此早期诊断、及时治疗是提高结直肠癌预后的主要因素. 目前结直肠癌的诊断主要依赖于对高危人群进行体格检查、大便潜血试验、X线、CT、纤维结肠镜, 但是这些方法对结直肠癌的诊断尤其是对Ⅰ期的诊断较为困难. 发现对结直肠癌不同分期和亚型有意义的标志物对结直肠癌的预防、诊断及治疗有重要意义[7-8]. 但是, 目前常用的血清标志物如CEA、CA19-9、CA242、CA72-4以及组织多肽抗原、组织多肽特意抗原等对结直肠癌的诊断灵敏度、特异度较低[8]. 肿瘤的多因素特性表明多种标志物的联合检测对肿瘤的早期发现和诊断有重要意义[9].
基因组学及蛋白质组学的迅速发展为结直肠癌诊断和标志物鉴定等方面的研究提供了新思路. 基因的功能不仅由排序决定, 他给予生命信息之多, 变异之广使罹病机制成了一个极其复杂的问题[10]. 蛋白质由含特定DNA序列的氨基酸经四级结构装配而成, 不同的蛋白质由于组成的肽链和结构不同, 有着不同的生物学功能, 他直接反映基因给予的信息, 他的功能一旦出现异常就可引发疾病, 复杂的蛋白质组学模型可以反应多种组织的病理状态, 蛋白质组学技术可以鉴别肿瘤细胞的不同亚型, 通过对这些蛋白或肽的定量与定性研究, 可以发现肿瘤特异标志物. 因此, 研究蛋白质的组成及性质是揭示基因和疾病联结链条中的关键一环, 而肿瘤蛋白质组学为肿瘤标志物的发现提供了一个新的方法, 应用这种方法, 已经发现和鉴定了载脂蛋白A1等一些标志物[11-13]. 肿瘤是一个多因素多阶段的慢性疾病, 在疾病发生发展的过程中, 一些肿瘤细胞的蛋白质或多肽可以被分泌到循环系统中, 血液的蛋白质组学可发生相应变化, 反映出机体的疾病状态, 从而使得应用血清进行结直肠癌蛋白质组学研究、发现患者的血清特异标志物成为一种可能. 而血清标志物的检测具有微创、患者易于接受等优点.
由美国Ciphergen Biosystems公司发明的SELDI-TOF-MS是蛋白质芯片和质谱技术的结合, 主要由蛋白质芯片、芯片阅读器和生物信息学三大部分组成, 其表面为可结合蛋白质的固体点表面, 检测的灵敏度高, 可把传统方法检测不到的蛋白和多肽检测出来, 可直接分析患者血清、淋巴液、脑脊液、尿液、细胞分泌液等样品而无需复杂的样品制备, 继Petricoin et al[14]利用SELDI-TOF-MS分析了116例血清标本(其中卵巢癌50例, 正常对照66例)得出可以完全检出卵巢癌的血清蛋白质分类树模型之后, SELDI-TOF-MS广泛用于卵巢癌、前列腺癌、肝癌、食管癌、结直肠癌等多种肿瘤的研究, 已成为肿瘤蛋白质组学主要技术平台之一. 结直肠散发性中分化腺癌是结直肠癌的主要病理类型, 选择该病理类型患者进行研究对结直肠癌的早期诊断有较好的代表性. 预实验表明在SAX2、WCX2和IMAC30-Cu2+这3种芯片之中, IMAC30-Cu2+芯片应用于SELDI-TOF-MS技术可以结合较多的蛋白, 得到稳定的质谱图, 因此我们选择了IMAC30-Cu2+芯片和SELDI-TOF-MS分析了335例血清样本(其中结直肠癌169例, 健康志愿者163例)的蛋白质谱图, 建立了由M/Z为5765的蛋白组成的分类树模型用于鉴别结直肠癌和正常人, 经独立样本的双盲验证其正确率、灵敏度、特异性、YI及阳性预测值分别为98.21%(165/168)、97.65%(83/85)、98.80%(82/83)、0.97和98.81%(83/84), 显著优于血清CEA的检测, 具有较好的诊断意义, 为结直肠癌分子诊断提供了潜在标志物.
今后我们将应用蛋白质组学技术鉴定所发现的差异蛋白及分类树模型中M/Z峰对应蛋白, 以便进一步提高诊断结直肠腺癌不同亚型和TNM临床分期的灵敏度和特异度, 明确这些蛋白的来源, 如宿主器官、肿瘤或代谢产物[15-16]. 此外, 我们将用分类树模型鉴别结直肠癌与胃癌、食管癌等常见肿瘤.
总之, 我们应用SELDI-TOF-MS和IMAC30-Cu2+芯片发现了可以快速识别经病理确诊为结直肠癌患者的血清蛋白标志物. 结果表明应用SELDI-TOF-MS、IMAC30-Cu2+芯片和有效的分析软件可以得到能准确鉴别结直肠癌和正常人的血清分类树模型, 具有较高灵敏度、特异度及阳性预测值, 对提高结直肠癌的早期诊断水平、改善结直肠癌预后具有重要的潜在意义, 为结直肠癌血清标志物的鉴定提供了候选蛋白.
结直肠癌的诊断及治疗发生了很大的改进, 但其发病率和死亡率并未发生根本的变化, 主要原因在于缺乏高灵敏度、特异度的早期诊断方法. SELDI-TOF-MS是蛋白质芯片和质谱技术的结合, 具有高通量、高灵敏度等技术优势, 可检测到传统方法检测不到的蛋白质或多肽, 已成为肿瘤蛋白质组学主要技术平台之一, 为肿瘤早期诊断的研究开拓了新的视野.
何超, 教授, 浙江大学医学院附属邵逸夫医院肛肠外科
应用SELDI-TOF-MS技术进行比较蛋白质组学研究已成为疾病蛋白质组学研究的热点, 不同的研究小组建立了多种疾病的血清或组织蛋白质谱分类树模型, 发现了一些有意义的蛋白质或多肽, 但由于这些标志物多是低丰度、小分子质量蛋白, 其鉴定是一项具有挑战性的工作.
Melle et al利用SELDI-TOF-MS和相关蛋白质组学技术发现和鉴定了S100A11在结直肠癌组织中的表达水平高于其在正常上皮和腺瘤组织中的表达.
利用SELDI-TOF-MS进行结直肠癌的比较蛋白质组学研究, 发现了多个有意义的蛋白, 科研设计严密, 结果符合统计学要求.
SELDI-TOF-MS检测得到的血清蛋白质组分类树模型可用于结直肠癌的初步筛查, 为血清标志物的发现提供了目标蛋白.
SELDI-TOF-MS: 即表面增强激光解析离子化飞行时间质谱, 由美国Ciphergen Biosystems公司发明, 是蛋白质芯片和质谱技术的结合, 主要由蛋白质芯片、芯片阅读器和生物信息学三大部分组成, 其表面为可结合蛋白质的固体点表面, 具有高通量、高灵敏度等技术优势.
文章目的及结论明确, 实验方法较为先进, 统计分析结果较可靠, 参考文献引用恰当而充分, 内容及结论对结直肠癌的早期诊断有一定的临床应用价值.
编辑: 李军亮 电编:吴鹏朕
| 1. | Compton CC, Greene FL. The staging of colorectal cancer: 2004 and beyond. CA Cancer J Clin. 2004;54:295-308. [PubMed] |
| 2. | Zhao G, Gao CF, Song GY, Li DH, Wang XL. [Identification of colorectal cancer using proteomic patterns in serum]. Ai Zheng. 2004;23:614-618. [PubMed] |
| 3. | Jemal A, Siegel R, Ward E, Hao Y, Xu J, Murray T, Thun MJ. Cancer statistics, 2008. CA Cancer J Clin. 2008;58:71-96. [PubMed] [DOI] |
| 5. | O'Connell JB, Maggard MA, Liu JH, Etzioni DA, Ko CY. Are survival rates different for young and older patients with rectal cancer? Dis Colon Rectum. 2004;47:2064-2069. [PubMed] [DOI] |
| 6. | Sarfaty M, Wender R. How to increase colorectal cancer screening rates in practice. CA Cancer J Clin. 2007;57:354-366. [PubMed] [DOI] |
| 7. | Crawford NP, Colliver DW, Galandiuk S. Tumor markers and colorectal cancer: utility in management. J Surg Oncol. 2003;84:239-248. [PubMed] [DOI] |
| 8. | Duffy MJ, van Dalen A, Haglund C, Hansson L, Klapdor R, Lamerz R, Nilsson O, Sturgeon C, Topolcan O. Clinical utility of biochemical markers in colorectal cancer: European Group on Tumour Markers (EGTM) guidelines. Eur J Cancer. 2003;39:718-727. [PubMed] [DOI] |
| 9. | Li J, Zhang Z, Rosenzweig J, Wang YY, Chan DW. Proteomics and bioinformatics approaches for identification of serum biomarkers to detect breast cancer. Clin Chem. 2002;48:1296-1304. [PubMed] |
| 11. | Zhang Z, Bast RC Jr, Yu Y, Li J, Sokoll LJ, Rai AJ, Rosenzweig JM, Cameron B, Wang YY, Meng XY, Berchuck A, Van Haaften-Day C, Hacker NF, de Bruijn HW, van der Zee AG, Jacobs IJ, Fung ET, Chan DW. Three biomarkers identified from serum proteomic analysis for the detection of early stage ovarian cancer. Cancer Res. 2004;64:5882-5890. [PubMed] [DOI] |
| 12. | Albrethsen J, Bogebo R, Gammeltoft S, Olsen J, Winther B, Raskov H. Upregulated expression of human neutrophil peptides 1, 2 and 3 (HNP 1-3) in colon cancer serum and tumours: a biomarker study. BMC Cancer. 2005;5:8. [PubMed] [DOI] |
| 13. | Adam BL, Qu Y, Davis JW, Ward MD, Clements MA, Cazares LH, Semmes OJ, Schellhammer PF, Yasui Y, Feng Z. Serum protein fingerprinting coupled with a pattern-matching algorithm distinguishes prostate cancer from benign prostate hyperplasia and healthy men. Cancer Res. 2002;62:3609-3614. [PubMed] [DOI] |
| 14. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] [DOI] |
| 15. | Jones MB, Krutzsch H, Shu H, Zhao Y, Liotta LA, Kohn EC, Petricoin EF 3rd. Proteomic analysis and identification of new biomarkers and therapeutic targets for invasive ovarian cancer. Proteomics. 2002;2:76-84. [PubMed] [DOI] |
| 16. | Poon TC, Johnson PJ. Proteome analysis and its impact on the discovery of serological tumor markers. Clin Chim Acta. 2001;313:231-239. [PubMed] [DOI] |