修回日期: 2008-01-15
接受日期: 2008-01-27
在线出版日期: 2008-02-28
MicroRNA(miRNA)是一类内源性非编码的小RNA, 广泛存在于动物、植物和病毒中. 通过与目标mRNA互补配对, 调控靶基因的表达或翻译, 在生物体的发育、增殖、分化和凋亡等方面发挥重要的生理作用. 最近研究表明miRNA与肿瘤发生存在密切关系. 利用实验生物学及生物信息学方法对与肿瘤相关的miRNA进行研究是当前研究的热点. 其中, 在肝癌中目前已发现若干miRNA如miRNA-122、-21、-195、-18、-199等的异常表达与之有关. 而在乙型肝炎病毒(HBV)感染相关疾病中的miRNA表达的研究尚不多见. 本文就MicroRNA与肝癌发生发展之间的关系作一综述.
引文著录: 黄金明, 叶进, 陈立波. MicroRNA与HBV相关性肝癌发生发展关系的研究进展. 世界华人消化杂志 2008; 16(6): 645-652
Revised: January 15, 2008
Accepted: January 27, 2008
Published online: February 28, 2008
MicroRNA is a kind of endogenous non-coding small RNA, is universally present in animals, plants and viruses and plays an important role in the development, proliferation, differentiation and apoptosis of organisms through complementary pairing with target mRNA, which can regulate its expression or translation. Recent studies indicate that miRNA is closely associated with tumors. Study on tumor-associated miRNA using techniques of experimental biology and bioinformatics is currently a hot subject of research. Recent studies on hepatocellular carcinoma(HCC) have shown that HCC is correlated with abnormal expression of several miRNAs, such as miRNA-122, -21, -195, -18, -199a. However, few studies on miRNA expression in HBV-infected diseases are available. This paper reviews researches on the association of microRNA with the development of liver cancer.
- Citation: Huang JM, Ye J, Chen LB. Advances in research of microRNA- and HBV-related liver cancer. Shijie Huaren Xiaohua Zazhi 2008; 16(6): 645-652
- URL: https://www.wjgnet.com/1009-3079/full/v16/i6/645.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i6.645
MicroRNA(miRNA)是近年的一个研究热点, 随着研究的深入, 越来越多的miRNA被发现, 其中, miRNA与肿瘤的关系是很多研究的重点, 已经发现若干miRNA通过负调控基因的表达与慢性淋巴细胞性白血病、肺癌、乳腺癌、结肠癌高度相关. miRNA的正调控靶基因现象是最近的发现, 具体机制还不明确. 有学者提出了"癌microRNA"(OncomiRs)[1-3]的观点, 即认为某些miRNA的异常表达在肿瘤的发生发展过程中充当了类似癌基因的角色. 肝癌, 特别是乙型肝炎相关性肝癌在我国的发病率高, 死亡率高, 严重危害人们的健康, 而乙型肝炎相关性肝癌目前尚无满意的治疗. 国内外学者对肝癌开展了广泛而深入的研究, 但乙肝病毒与肝癌发生发展的关系尚未完全阐明. 近来研究表明miRNA可能在肝癌发生与进展中发挥重要作用. 本文就miRNA与肝癌发生发展之间关系的研究作一综述.
miRNA亦称微小RNA, 是一种广泛存在于真核生物中、大小约20-22个核苷酸(nt)的内源性单链小分子RNA, 在生物进化过程中高度保守, 在细胞中具有时空特异性表达模式. miRNA不编码蛋白, 而是通过与靶基因序列特异性相互作用, 在转录或翻译水平调节相关基因表达, 参与发育、增殖、分化、凋亡等多种生物学过程[4-5]. 自1993年第一个miRNA编码基因lin-4由Lee et al[6]在线虫体内发现以来, 人们相继在烟草、果蝇、斑马鱼、哺乳动物与人体内[7]发现了大量miRNA编码基因. 据生物信息学预测, 人类基因组内存在着约1000个miRNA基因, 调控基因组内1/3左右基因的表达[8]. 至2007-12, Sanger最新的miRNA数据库Release10.1已收录了5395个miRNA, 其中人类有851个(http://microrna.sanger.ac.uk/sequences/).
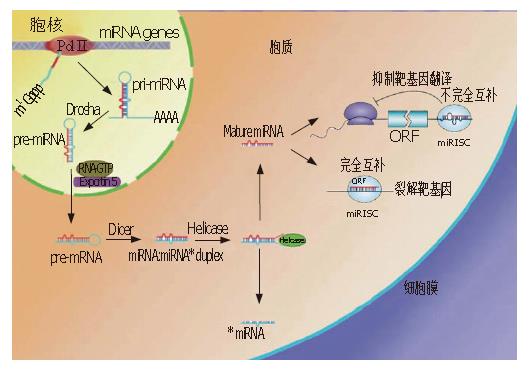
编码miRNA的基因大多位于染色体内含子、基因间区域或非编码mRNA外显子, 一些miRNA在基因组中有多个位点, 也有串联排列的, 由RNA聚合酶Ⅱ转录. 在动物细胞核中, 首先转录出一个很大的初始转录子(pri-miRNA), 有的长达7000 nt. pri-miRNA被Drosha(一种核酸内切酶RNase Ⅲ)处理成约70-80 nt的发夹状茎-环结构, 即miRNA前体(pre-miRNA). pre-miRNA由小分子单体G蛋白Ran-GTP依赖的转运蛋白exportin-5运输到胞质, 被Dicer(另一种核酸内切酶RNase Ⅲ)剪切成约22个核苷酸的双链miRNA[9](图1). 双链miRNA被解旋酶解螺旋后, 一条链被降解, 另一条成为5'端有磷酸基团, 3'端为羟基的成熟miRNA[5].
经典的研究认为, 成熟miRNA进入miRNA诱导的基因沉默复合物(miRNA-induced silencing complex; miRISC)中与目标mRNA配对, 主要通过两种机制来负调控目标基因的表达: (1)当miRNA和目的mRNA完全或几乎完全互补时, 导致目的mRNA降解, 此时的作用类似于小干扰RNA(siRNA)的功能; (2)当miRNA和目的mRNA不完全互补时, 则负调控翻译过程, 阻碍蛋白质翻译, 而对靶基因mRNA没有影响[9-11](图1). 第一种作用以线虫lin-4为代表, 第二种作用方式以拟南芥miR-171为代表, 而let-7则兼有两种机制. miRISC是一种核糖蛋白复合体(ribonucleoprotein complex, miRNP), 蛋白成分主要是Ago家族, 可介导miRNA 5'端第2-8核苷酸与mRNA 3'非翻译区(UTR)部分序列的结合. miRNA靠近5'端有一个与互补链不匹配的突起, 这个突起显著的减弱了双链结构5'端稳定性. 成熟miRNA总是趋向于选择双链中5'端相对更不稳定的序列, 所以只有一条成熟的miRNA最终能结合到RISC中. 另外, miRNA在靶mRNA识别过程中, miRNA 5'端的2-8 nt在与靶mRNA的结合中比3'端序列更为重要, 被称为种子区(seed region)[8], 这也可能与5'端的突起有关. 在极少的情况下, miRNA和互补链具有相似的稳定性, 其分别结合到miRISC上的概率也相似.
miRNA及siRNA的这种沉默靶基因的理论目前受到了挑战. Li et al[12]针对抑癌基因E-cadherin的启动子, 设计出一系列含有21 nt的双链RNA(dsRNA), 其中两种dsRNA(dsEcad-302和dsEcad-215)转染到两种人前列腺癌细胞株PC-3 和DU-145, 48 h后发现E-cadherin的mRNA和蛋白表达开始增加, 转染3 d后细胞的活性逐渐降低; 而相应的空白组及对照组E-cadherin表达及细胞活性均无变化. 在PC-3细胞中单独用dsEcad-215转染, 转然后10 d和13 d E-cadherin的表达仍升高14倍和3.8倍. 同样针对p21、VEGF启动子分别设计的dsRNAs转染至PC-3, HeLa, MCF-7三种肿瘤细胞后, 发现p21、VEGF的表达有着不同程度的增加. 在进一步探究dsRNAs作用机制时, 发现dsRNAs对靶基因的正调控作用需要Ago 2蛋白的参与及H3K9的去甲基化. 最近的研究[13]发现let-7及合成的miRcxcr4在细胞生长停滞时上调靶mRNA的表达, 而在增殖的细胞中抑制靶基因的表达, 认为在细胞周期中, miRNP对靶基因翻译的调控作用往返于抑制和激活之间. 这些研究颠覆了人们以往认为miRNA对基因仅负调控的观点, 为miRNA作用及作用机制的进一步研究开辟了一条新的道路. 但miRNA正调控靶基因的机制远不如负调控清楚, 还需更深入的研究. 有学者认为miRNA调节比蛋白水平的调节节省能量, 效果更快而且可逆, 大多数miRNA对靶基因起着微调作用, 少数miRNA调节可引起表型的明显改变. 不同组织或细胞在不同阶段表达不同的miRNA谱. 一个miRNA可调节数百个靶基因包括转录因子、细胞因子和受体等, miRNA和靶基因组成复杂的调节网络[2].
针对miRNA研究的技术和方法可概括为两类: 即实验生物学方法和生物信息学方法, 前者的代表是发展了检测miRNA前体和成熟miRNA表达的实时荧光定量PCR(RT-PCR), 基因芯片(microarray)、锁定的核苷酸原位杂交(LNA-ISH)、基于细珠的流式细胞术(bead-based flow cytometry)以及小RNA的体外抑制和过表达技术; 后者的代表是发展了数个成熟的miRNA基因预测软件[14-16](如miRseeker、MirScan、PalGrade、ProMiR、miRAlign)和miRNA靶基因预测软件[17-21](如TargetScan、miRand、PicTa、miRNApath、miRNAMap2.0)等. 数个功能得到成功诠释的小RNA分子(如miR-375和miR-223等[22-23])均是两种方法完美结合的研究典范, 两者各有优势, 互为补充. 生物信息学为小RNA研究提供了有益的线索, 可以指导实验的进行, 但仍需通过实验方法加以确认, 而实验生物学虽然在研究对象的选择上受到约束, 不能很快对大量候选者进行逐个验证, 却能提供直接而有力的证据.
目前研究表明动物中miRNA参与生命过程中一系列的重要进程, 包括早期胚胎发育、细胞增殖、分化、凋亡以及在基因表达调控中的作用. 最经典的lin-4在线虫幼虫的第一、二期积累, 通过抑制靶基因lin-14和lin-28的翻译, 引发其进入第三幼虫期[6]; 而存在于线虫幼虫第三、四期的let-7通过抑制lin-41和hbl-1的转录, 引导线虫从幼虫向成虫的转变[24-25]. Brennecke et al[26]在果蝇体内鉴定出第一个与增殖相关的miRNA基因bantam及其靶基因hid, Bantam与hid mRNA的3'UTR互补结合, 阻止了hid的翻译, 抑制蛋白的表达, 最终表现为促进细胞增殖的作用. miR-1与血管生成[27], miR-181与血细胞生成[28], miR-430与神经发生[29]等都得到了证实. 近年来, 发现miRNA与肿瘤的发生、发展密切相关[1-3,30]. 研究表明miR-15、miR-16与慢性淋巴细胞性白血病[31-32], miR-155、let-7与肺腺癌[33-34], miR-10b、miR-27a与乳腺癌[35-37], miR-21与多形性恶性胶质瘤、胃癌、结肠癌、乳腺癌、胰腺癌等[38-39]均存在高度的相关性, 虽然详细机制还有待于进一步阐明, 但强烈提示miRNA在肿瘤研究领域有广阔前景.
在我国乙型肝炎病毒(HBV)相关性肝细胞肝癌(hepatocellular carcinoma, HCC)发病率、死亡率均排名于疾病谱前列, 严重危害人民健康[40-42]. 近年来, 国内外对miRNA在肝癌发生发展中的调控作用进行了初步的研究.
日本学者Murakami et al[43]利用芯片技术对24例肝癌组织(HCC)、22例肝癌周边正常组织(NT)及9例慢性肝炎(CH)细针穿刺肝组织的miRNA表达谱进行分析发现, HCC与NT间表达有差异的miRNA有30个(P<0.01). 其中, 相对于NT, HCC表达明显上调的有3个: miR-18、pre-miR-18、miR-224, HCC表达明显下调的有5种: miR-199a-3p、miR-195、miR-199a、miR-200a、miR-125a(P<0.001). 该研究并指出与肝癌分化程度相关的miRNA有4个: miR-92、miR-20、miR-l8、pre-miR-18, 他们的表达水平在分化差的HCC中最高, 在分化良好的HCC中最低; 而miR-99a的表达水平与分化程度呈正相关. 可见, miR-18与pre-miR-18在肝癌的发生和失分化中均扮演了一定的角色. 另外, 他们对其中的14例肝硬化(LC)和12例CH标本(22例NT中有14例LC, 3例CH, 5例无病理信息)分析发现miRNA表达谱存在显著差异(P<0.01)的有12个(LC>CH: miR-28, miR-342, miR-126, miR-199a, miR-145b, miR-143, miR-368, pre-miR-372; LC<CH: miR-182, pre-miR-199b, miR-224, miR-15b). 但是, 对24例HCC的病因学(6例乙肝, 17例丙肝, 1例不祥)分析发现, HBV与HCV感染的肝癌组织miRNA表达谱无统计学差异.
国内孙凯et al[44]运用含114个miRNA的液相芯片及Luminex100检测系统对20对HCC及NT组织进行分析, 结果miRNA表达谱中表达差异的有28个(上调6个, 下调22个); 全部标本中共存性差异表达miRNA有9个, 其中上调2个: miR-222、miR-224, 下调7个: miR-122a、miR-143、miR-145、miR-195、miR-199a、miR-199a-3p、miR-200a(P<0.01). 并选取表达上调的miR-222及其预测靶基因CTGF(结缔组织生长因子)在2例标本中行RT-PCR及Western blot验证, 发现CTGF的mRNA水平在HCC与NT组织中无差异, 而蛋白水平在HCC中明显下降. 提示miR-222通过转录后基因沉默机制抑制CTGF的蛋白表达, 从而促进癌的浸润和转移. 上述Murakami et al[43]与孙氏的实验有多个miRNA表达一致如: 上调的miR-224, 下调的miR-195、miR-199a、miR-199a-3p、miR-200a. 对这些在不同实验中表达一致的miRNA更应引起研究者的注意, 同时也应认识到miRNA芯片的结果只是一个初步筛查, 对筛选出的miRNA需要通过进一步的实验验证才有意义.
LIN28[6]是线虫发育时序过程中的一个关键基因, Guo et al[45]发现LIN28的同源基因LIN28B高表达于正常睾丸、胎肝、胚胎、临床肝癌组织以及6个肝癌细胞系(Huh7、HepG2、Alex、HLE、Huh6、Hep3B)中, 外源性导入LIN28B可以促进肝癌细胞的增生, 并推断LIN28B可能通过其3'非编码区与let-7互补结合而接受后者的调控, 从而在细胞的生长和肿瘤的形成中发挥重要作用.
美国学者Kutay et al[46]利用含245种miRNAs的芯片分析叶酸、甲基、胆碱缺乏性大鼠肝肿瘤(FMD)模型与正常鼠肝的miRNA差异表达, 发现在癌组织中有23种miRNA表达上调, 3种下调. 其中在3例肿瘤组织中均升高达两倍及以上的有9种: miR-101b-2、-130、-130a、-172a-2、-219-1、-23a、-23b、-24、-328-1; 在3例肿瘤组织中均降低一半的仅有miR-122, 而miR-123与miR-235只在2例中降低. 同时, 对20例人肝癌组织测定表明, 相对于癌周肝组织, 有10例miR-122明显下降. 在人和鼠的多个肝癌细胞系(HepG2、Hep3B、H-7; Hepa、H4)中, miR-122检测不到或表达极低. 有研究表明miR-122作为一个调节肝脏发育的"肝特异性miRNA", 在肝脏的发育、分化及维持肝脏正常功能中发挥重要的作用. 在受精卵置入后12.5 d的小鼠即可检测到miR-122表达, 其表达水平在出生前达到平台期, 出生后以一种相当缓慢的方式升高, 而应用反义寡聚核苷酸使miR-122失活后, 出现肝脏功能受损, 胆固醇合成降低, 提示miR-122不仅参与肝脏分化的调节, 还在维持肝脏正常功能中起着重要作用[47]. miR-122也可以与丙肝病毒基因组相互作用调节丙肝病毒的表达, 当miR-122被灭活, 丙肝病毒复制水平明显降低[48]. 但在乙肝相关肝癌中miR-122的作用尚未见报道.
在Gramantieri et al[49]的研究中, 利用含有381种人类miRNA的芯片检测17例HCC和21例肝硬化肝组织(CL)标本, 发现35种miRNA表达有统计学差异(P<0.05). 相对于CL, miR-221在83%HCC中上调, let-7a-1、miR-122a在70%HCC中下调, Northern blot及RT-PCR进一步验证以上结果. 通过TargetScan和PicTar软件分析Cyclin G1是miR-122a的一个作用靶点; 在肝癌细胞系Hep3B、SNU449中, miR-122a用Northern blot不能检出, Cyclin G1蛋白用Western blot检测高表达. Cyclin G1与基因组的不稳定性相关[50], 在乳腺癌、结肠癌、平滑肌瘤组织中Cyclin G1蛋白高表达[51-53]. 因而miR-122a调控Cyclin G1功能的缺失可能在HCC发生与进展中具有重要作用.
Gastroenterology的一篇文章[54]则阐述了miR-21通过下调抑癌基因PTEN(phosphatase and tensin homolog)促进肝癌的增生、迁移和侵袭; 在肝癌细胞系中抑制miR-21, PTEN表达增加, 则减少肿瘤细胞的增生、迁移和侵袭; 将miR-21前体(pre-miR-21)转染在正常肝细胞中, 可以观察到转染细胞的迁移能力增强. 该研究也利用芯片技术证实了在人肝癌组织中miR-21显著高表达.
Pogribny et al[55]使用致鼠肝癌剂tamoxifen喂食大鼠, 诱导其发生肝癌, 12 wk及24 wk后检测鼠肝中的miRNA表达谱, 发现相对于对照组, 喂食tamoxifen组有33个(20个上调, 13个下调)在人、大鼠、小鼠中序列保守的miRNA表达异常(P<0.05). 一些被认为具有癌基因作用的miRNA如: miR-16、-17-5p、-20a、-20b、-106a、-34等有明显上调(P<0.001), 并确认这些miRNA的相应靶基因如Bcl2、E2F1、RB1、NOTCH1的蛋白表达下调(P<0.05), 而上述蛋白在细胞周期调节、凋亡、DNA复制、染色质修饰以及肿瘤发生过程中发挥作用; 其他检测到上调的miRNA有miR-18、-199b、-193、-31、-345、-365、-375、-424、-92、-25、-362、-93、-324-5p、-15a, 下调的有miR-195、-422b、-152、-28、-191、-350、-361、-22-3p、-27a、-378、-203、-192、-194, 并认为miRNA的异常表达发生于肿瘤形成的早期阶段, 可以作为肿瘤形成过程的生物标志.
国内学者[56]对59个相关miRNA进行分析, 发现在96例肝癌组织中3个miRNA有4处序列变异, 其中miR-106b存在2处已被鉴定的单核苷酸多态性(SNP), miR-192和let-7a-2各存在1处未报道过的变异. 并且在癌旁组织中也发现相同变异, 提示这些变异可能是种系变异或自然的多态现象. 而在8种肝癌细胞株中, 未发现miRNA的序列变异. 故作者认为miRNA的变异在肝癌中少见, 不能认为是肝癌发生的一个主要机制.
国内学者刘妍et al[57]利用人肝母细胞瘤细胞系HepG2及其转染HBV基因组的HepG2.2.15细胞的microRNA的表达谱差异来研究肝细胞中HBV复制的生物学机制, 结果显示HepG2.2.15细胞与HepG2细胞间差异表达的miRNA共27种(占5.3%), 按照HepG2.2.15与HepG2中表达强度排列, 其中表达上调的有7种(miR-181a、-196b、-181c、181d、-483、-183、-429), 表达下调的有20种(miR-129-3p、-15a、-195、-422b、-16、pre-miR-131、-15b、-523、-338、-516-3b、-422a、-520b、-212、-520c、-17-3p、-7、-202、-501、-198、pre-miR-166). 其中, 本实验miR-195的低表达与Murakami et al[43]和孙凯et al[44]的研究中miR-195的低表达提示我们, miR-195的下调可能在HBV相关性肝癌的发生发展中起到重要的作用, 具体机制还需要进一步的研究.
病毒是一种极小的寄生生物, 只能依赖宿主细胞进行复制、转录、翻泽以维持其生存, 但其复制等过程中可能会造成宿主细胞基因表达水平的改变. 考虑到miRNA在基因表达调控中所起到的重要作用, 病毒这种只具有有限基因编码能力的生物同样可能利用miRNA调节宿主或自身某些基因的表达. 2004年Pfeffer et al[58]报道在EB病毒(Epstein-Bar Virus)基因组中发现了5个miRNA, 这是首次发现病毒编码miRNA. 到目前为止, 通过在病毒感染的细胞中利用cDNA克隆和生物信息学方法, 在疱疹病毒(herpesvirus)[59-64]、多瘤病毒(polyomaviruses)[65]、腺病毒(adenoviruses)[66]以及逆转录病毒(retro-viruses)[67]中发现上百个miRNA. 其中, 在HCV和HIV等慢性病毒感染中, miRNA的重要作用已得到证实[48,68-70]. 有学者[71]利用生物信息学技术, 从HBV基因组中仅找到了一个候选的病毒编码pre-miRNA, 推断其成熟miRNA序列为: CAUGUCCUACUGUUCAAGCCUC; 但通过分析, 在人类基因组中没有找到潜在靶基因, 而在HBV病毒基因组中却找到了三个作用靶点. 这提示我们HBV编码的miRNA可能通过调节自身mRNA的表达发挥病理生理作用, 但尚未见相关实验结果证实及报道. 在病毒感染性疾病中, 宿主细胞中的miRNA可能在病毒生命活动周期中起着重要作用, 而病毒自身为了在宿主细胞中成功存活, 也必定会反过来调节宿主细胞miRNA的表达, 从而参与炎症、肿瘤等疾病的发生发展[72].
miRNA作为一类内源性非编码的小RNAs, 通过与靶基因序列特异性相互作用, 在转录或翻译水平调节相关基因表达, 参与发育、增殖、分化、凋亡等多种生物学过程[4-5]. 与肿瘤相关的miRNA的研究是近年的研究热点[1-3], 在白血病[30-32]、肺癌[33-35]、乳腺癌[36-37]、肝癌[38-39]等肿瘤中均证实有特定miRNA的异常表达. 目前对肝细胞性肝癌的研究中已发现若干miRNA, 如miRNA-122[43-44]、-21[45-46]、-195[47-48]、-18[49]、-199a[54-55]等的异常表达与之有关. 在病毒感染性疾病中[58-59], 宿主细胞中的miRNA可能在病毒生命活动周期中起着重要作用[60-62], 而病毒自身为了在宿主细胞中成功存活, 也可能反过来调节宿主细胞miRNA的表达[63-65], 从而参与炎症、肿瘤等疾病的发生发展[66-70,72]. 目前研究表明, 表达HBV的HepG2.2.15细胞株与不表达HBV的肝癌细胞株HepG2相比, 两者的若干miRNA表达水平存在显著差异[57]. 生物信息学提示[71], HBV可以编码miRNA, 并且该miRNA能调节乙肝病毒自身的基因表达, 但尚需实验证实. 乙肝相关性肝癌是我国最常见的恶性肿瘤之一, 发病率、死亡率均排名于疾病谱前列[40-42]. 针对我国乙肝患者多的国情, 对该类人群开展miRNA相关表达谱研究, 将为乙肝相关性肝癌的发生发展机制的研究提供一个新的方向.
自从1993年在线虫中发现第一个miRNA-lin4, 2001年阐明另一个miRNA-let-7在线虫发育中的功能以来, miRNA成为RNA干扰领域中的一大研究热点, 越来越多的证据表明, miRNA在生物的生理及病理过程中发挥着极其重要的作用.
谭德明, 教授, 中南大学湘雅医院感染病科.
在miRNA的研究中, 探讨癌症的发生发展机制及病毒的感染致病中, miRNA所扮演的角色是学界关注的焦点. 作为内源性的小RNA, 寻找并筛选具体疾病中某些miRNA的异常表达, 结合其调节靶基因表达的独特作用, 可为癌症及HBV、HCV、HIV等难治性感染疾病提供新思路.
本文针对我国HBV相关性肝癌发病高, 危害大的状况, 主要介绍了在miRNA研究领域中肝癌及HBV感染的研究. 虽然目前国内外学者在这方面做了大量的工作, 但仍有大量的问题等待更多更深的研究去解答. 将HBV、肝癌与miRNA结合起来考虑是本文的一个创新.
本文在着重介绍miRNA与肝癌的关系之外, 还简要介绍了miRNA的一些基本知识及研究方法, 为读者提供全面完整的信息, 更利于理解具体的研究论文. 同时, 对数个独立研究的总结分析, 提出一些可能在HBV相关性肝癌中具有研究前景的miRNA.
本文反映了MicroRNA与肝癌发生发展间关系的最新研究动态, 文笔流畅, 层次清楚, 是一篇较优秀的综述性文章.
编辑: 李军亮 电编: 吴鹏朕
| 1. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 2. | Hammond SM. MicroRNAs as oncogenes. Curr Opin Genet Dev. 2006;16:4-9. [PubMed] [DOI] |
| 3. | Cho WC. OncomiRs: the discovery and progress of microRNAs in cancers. Mol Cancer. 2007;6:60. [PubMed] [DOI] |
| 4. | Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T. Identification of novel genes coding for small expressed RNAs. Science. 2001;294:853-858. [PubMed] [DOI] |
| 5. | Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 2004;116:281-297. [PubMed] [DOI] |
| 6. | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-854. [PubMed] [DOI] |
| 7. | Bentwich I, Avniel A, Karov Y, Aharonov R, Gilad S, Barad O, Barzilai A, Einat P, Einav U, Meiri E. Identification of hundreds of conserved and nonconserved human microRNAs. Nat Genet. 2005;37:766-770. [PubMed] [DOI] |
| 8. | Lewis BP, Burge CB, Bartel DP. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell. 2005;120:15-20. [PubMed] [DOI] |
| 9. | Liu W, Mao SY, Zhu WY. Impact of tiny miRNAs on cancers. World J Gastroenterol. 2007;13:497-502. [PubMed] [DOI] |
| 10. | He L, Hannon GJ. MicroRNAs: small RNAs with a big role in gene regulation. Nat Rev Genet. 2004;5:522-531. [PubMed] [DOI] |
| 11. | Chen PY, Meister G. microRNA-guided posttranscriptional gene regulation. Biol Chem. 2005;386:1205-1218. [PubMed] [DOI] |
| 12. | Li LC, Okino ST, Zhao H, Pookot D, Place RF, Urakami S, Enokida H, Dahiya R. Small dsRNAs induce transcriptional activation in human cells. Proc Natl Acad Sci USA. 2006;103:17337-17342. [PubMed] [DOI] |
| 13. | Vasudevan S, Tong Y, Steitz JA. Switching from repression to activation: microRNAs can up-regulate translation. Science. 2007;318:1931-1934. [PubMed] [DOI] |
| 14. | Lai EC, Tomancak P, Williams RW, Rubin GM. Computational identification of Drosophila microRNA genes. Genome Biol. 2003;4:R42. [PubMed] [DOI] |
| 15. | Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ. miRBase: microRNA sequences, targets and gene nomenclature. Nucleic Acids Res. 2006;34:D140-D144. [PubMed] [DOI] |
| 16. | Doran J, Strauss WM. Bio-informatic trends for the determination of miRNA-target interactions in mammals. DNA Cell Biol. 2007;26:353-360. [PubMed] [DOI] |
| 17. | Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell. 2003;115:787-798. [PubMed] [DOI] |
| 18. | John B, Enright AJ, Aravin A, Tuschl T, Sander C, Marks DS. Human MicroRNA targets. PLoS Biol. 2004;2:e363. [PubMed] [DOI] |
| 19. | Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M. Combinatorial microRNA target predictions. Nat Genet. 2005;37:495-500. [PubMed] [DOI] |
| 20. | Chiromatzo AO, Oliveira TY, Pereira G, Costa AY, Montesco CA, Gras DE, Yosetake F, Vilar JB, Cervato M, Prado PR. miRNApath: a database of miRNAs, target genes and metabolic pathways. Genet Mol Res. 2007;6:859-865. [PubMed] |
| 21. | Hsu SD, Chu CH, Tsou AP, Chen SJ, Chen HC, Hsu PW, Wong YH, Chen YH, Chen GH, Huang HD. miRNAMap 2.0: genomic maps of microRNAs in metazoan genomes. Nucleic Acids Res. 2008;36:D165-D169. [PubMed] |
| 22. | Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, Macdonald PE, Pfeffer S, Tuschl T, Rajewsky N, Rorsman P. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 2004;432:226-230. [PubMed] [DOI] |
| 23. | Fazi F, Rosa A, Fatica A, Gelmetti V, De Marchis ML, Nervi C, Bozzoni I. A minicircuitry comprised of microRNA-223 and transcription factors NFI-A and C/EBPalpha regulates human granulopoiesis. Cell. 2005;123:819-831. [PubMed] [DOI] |
| 24. | Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature. 2000;403:901-906. [PubMed] [DOI] |
| 25. | Lin SY, Johnson SM, Abraham M, Vella MC, Pasquinelli A, Gamberi C, Gottlieb E, Slack FJ. The C elegans hunchback homolog, hbl-1, controls temporal patterning and is a probable microRNA target. Dev Cell. 2003;4:639-650. [PubMed] [DOI] |
| 26. | Brennecke J, Hipfner DR, Stark A, Russell RB, Cohen SM. bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila. Cell. 2003;113:25-36. [PubMed] [DOI] |
| 27. | Zhao Y, Samal E, Srivastava D. Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis. Nature. 2005;436:214-220. [PubMed] [DOI] |
| 28. | Chen CZ, Li L, Lodish HF, Bartel DP. MicroRNAs modulate hematopoietic lineage differentiation. Science. 2004;303:83-86. [PubMed] [DOI] |
| 29. | Giraldez AJ, Cinalli RM, Glasner ME, Enright AJ, Thomson JM, Baskerville S, Hammond SM, Bartel DP, Schier AF. MicroRNAs regulate brain morphogenesis in zebrafish. Science. 2005;308:833-838. [PubMed] [DOI] |
| 30. | Calin GA, Croce CM. MicroRNA signatures in human cancers. Nat Rev Cancer. 2006;6:857-866. [PubMed] [DOI] |
| 31. | Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci USA. 2004;101:11755-11760. [PubMed] [DOI] |
| 32. | Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K. Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sci USA. 2002;99:15524-15529. [PubMed] [DOI] |
| 33. | Yanaihara N, Caplen N, Bowman E, Seike M, Kumamoto K, Yi M, Stephens RM, Okamoto A, Yokota J, Tanaka T. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell. 2006;9:189-198. [PubMed] [DOI] |
| 34. | Lee YS, Dutta A. The tumor suppressor microRNA let-7 represses the HMGA2 oncogene. Genes Dev. 2007;21:1025-1030. [PubMed] [DOI] |
| 35. | Iorio MV, Ferracin M, Liu CG, Veronese A, Spizzo R, Sabbioni S, Magri E, Pedriali M, Fabbri M, Campiglio M. MicroRNA gene expression deregulation in human breast cancer. Cancer Res. 2005;65:7065-7070. [PubMed] [DOI] |
| 36. | Ma L, Teruya-Feldstein J, Weinberg RA. Tumour invasion and metastasis initiated by microRNA-10b in breast cancer. Nature. 2007;449:682-688. [PubMed] [DOI] |
| 37. | Mertens-Talcott SU, Chintharlapalli S, Li X, Safe S. The oncogenic microRNA-27a targets genes that regulate specificity protein transcription factors and the G2-M checkpoint in MDA-MB-231 breast cancer cells. Cancer Res. 2007;67:11001-11011. [PubMed] [DOI] |
| 38. | Ciafrè SA, Galardi S, Mangiola A, Ferracin M, Liu CG, Sabatino G, Negrini M, Maira G, Croce CM, Farace MG. Extensive modulation of a set of microRNAs in primary glioblastoma. Biochem Biophys Res Commun. 2005;334:1351-1358. [PubMed] [DOI] |
| 39. | Volinia S, Calin GA, Liu CG, Ambs S, Cimmino A, Petrocca F, Visone R, Iorio M, Roldo C, Ferracin M. A microRNA expression signature of human solid tumors defines cancer gene targets. Proc Natl Acad Sci USA. 2006;103:2257-2261. [PubMed] [DOI] |
| 40. | Merican I, Guan R, Amarapuka D, Alexander MJ, Chutaputti A, Chien RN, Hasnian SS, Leung N, Lesmana L, Phiet PH. Chronic hepatitis B virus infection in Asian countries. J Gastroenterol Hepatol. 2000;15:1356-1361. [PubMed] [DOI] |
| 41. | Tang ZY. Hepatocellular carcinoma--cause, treatment and metastasis. World J Gastroenterol. 2001;7:445-454. [PubMed] [DOI] |
| 42. | Luo RH, Zhao ZX, Zhou XY, Gao ZL, Yao JL. Risk factors for primary liver carcinoma in Chinese population. World J Gastroenterol. 2005;11:4431-4434. [PubMed] [DOI] |
| 43. | Murakami Y, Yasuda T, Saigo K, Urashima T, Toyoda H, Okanoue T, Shimotohno K. Comprehensive analysis of microRNA expression patterns in hepatocellular carcinoma and non-tumorous tissues. Oncogene. 2006;25:2537-2545. [PubMed] [DOI] |
| 45. | Guo Y, Chen Y, Ito H, Watanabe A, Ge X, Kodama T, Aburatani H. Identification and characterization of lin-28 homolog B (LIN28B) in human hepatocellular carcinoma. Gene. 2006;384:51-61. [PubMed] [DOI] |
| 46. | Kutay H, Bai S, Datta J, Motiwala T, Pogribny I, Frankel W, Jacob ST, Ghoshal K. Downregulation of miR-122 in the rodent and human hepatocellular carcinomas. J Cell Biochem. 2006;99:671-678. [PubMed] [DOI] |
| 47. | Chang J, Nicolas E, Marks D, Sander C, Lerro A, Buendia MA, Xu C, Mason WS, Moloshok T, Bort R. miR-122, a mammalian liver-specific microRNA, is processed from hcr mRNA and may downregulate the high affinity cationic amino acid transporter CAT-1. RNA Biol. 2004;1:106-113. [PubMed] [DOI] |
| 48. | Jopling CL, Yi M, Lancaster AM, Lemon SM, Sarnow P. Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA. Science. 2005;309:1577-1581. [PubMed] [DOI] |
| 49. | Gramantieri L, Ferracin M, Fornari F, Veronese A, Sabbioni S, Liu CG, Calin GA, Giovannini C, Ferrazzi E, Grazi GL. Cyclin G1 is a target of miR-122a, a microRNA frequently down-regulated in human hepatocellular carcinoma. Cancer Res. 2007;67:6092-6099. [PubMed] [DOI] |
| 50. | Tanaka S, Diffley JF. Deregulated G1-cyclin expression induces genomic instability by preventing efficient pre-RC formation. Genes Dev. 2002;16:2639-2649. [PubMed] [DOI] |
| 51. | Reimer CL, Borras AM, Kurdistani SK, Garreau JR, Chung M, Aaronson SA, Lee SW. Altered regulation of cyclin G in human breast cancer and its specific localization at replication foci in response to DNA damage in p53+/+ cells. J Biol Chem. 1999;274:11022-11029. [PubMed] [DOI] |
| 52. | Perez R, Wu N, Klipfel AA, Beart RW. A better cell cycle target for gene therapy of colorectal cancer: cyclin G. J Gastrointest Surg. 2003;7:884-889. [PubMed] [DOI] |
| 53. | Baek WK, Kim D, Jung N, Yi YW, Kim JM, Cha SD, Bae I, Cho CH. Increased expression of cyclin G1 in leiomyoma compared with normal myometrium. Am J Obstet Gynecol. 2003;188:634-639. [PubMed] [DOI] |
| 54. | Meng F, Henson R, Wehbe-Janek H, Ghoshal K, Jacob ST, Patel T. MicroRNA-21 regulates expression of the PTEN tumor suppressor gene in human hepatocellular cancer. Gastroenterology. 2007;133:647-658. [PubMed] [DOI] |
| 55. | Pogribny IP, Tryndyak VP, Boyko A, Rodriguez-Juarez R, Beland FA, Kovalchuk O. Induction of microRNAome deregulation in rat liver by long-term tamoxifen exposure. Mutat Res. 2007;619:30-37. [PubMed] [DOI] |
| 56. | Yang J, Zhou F, Xu T, Deng H, Ge YY, Zhang C, Li J, Zhuang SM. Analysis of sequence variations in 59 microRNAs in hepatocellular carcinomas. Mutat Res. 2008;638:205-209. [PubMed] [DOI] |
| 58. | Pfeffer S, Zavolan M, Grässer FA, Chien M, Russo JJ, Ju J, John B, Enright AJ, Marks D, Sander C. Identification of virus-encoded microRNAs. Science. 2004;304:734-736. [PubMed] [DOI] |
| 59. | Cui C, Griffiths A, Li G, Silva LM, Kramer MF, Gaasterland T, Wang XJ, Coen DM. Prediction and identification of herpes simplex virus 1-encoded microRNAs. J Virol. 2006;80:5499-5508. [PubMed] [DOI] |
| 60. | Pfeffer S, Sewer A, Lagos-Quintana M, Sheridan R, Sander C, Grässer FA, van Dyk LF, Ho CK, Shuman S, Chien M. Identification of microRNAs of the herpesvirus family. Nat Methods. 2005;2:269-276. [PubMed] [DOI] |
| 61. | Burnside J, Bernberg E, Anderson A, Lu C, Meyers BC, Green PJ, Jain N, Isaacs G, Morgan RW. Marek's disease virus encodes MicroRNAs that map to meq and the latency-associated transcript. J Virol. 2006;80:8778-8786. [PubMed] [DOI] |
| 62. | Grey F, Antoniewicz A, Allen E, Saugstad J, McShea A, Carrington JC, Nelson J. Identification and characterization of human cytomegalovirus-encoded microRNAs. J Virol. 2005;79:12095-12099. [PubMed] [DOI] |
| 63. | Grundhoff A, Sullivan CS, Ganem D. A combined computational and microarray-based approach identifies novel microRNAs encoded by human gamma-herpesviruses. RNA. 2006;12:733-750. [PubMed] [DOI] |
| 64. | Cai X, Lu S, Zhang Z, Gonzalez CM, Damania B, Cullen BR. Kaposi's sarcoma-associated herpesvirus expresses an array of viral microRNAs in latently infected cells. Proc Natl Acad Sci USA. 2005;102:5570-5575. [PubMed] [DOI] |
| 65. | Sullivan CS, Grundhoff AT, Tevethia S, Pipas JM, Ganem D. SV40-encoded microRNAs regulate viral gene expression and reduce susceptibility to cytotoxic T cells. Nature. 2005;435:682-686. [PubMed] [DOI] |
| 66. | Sano M, Kato Y, Taira K. Sequence-specific interference by small RNAs derived from adenovirus VAI RNA. FEBS Lett. 2006;580:1553-1564. [PubMed] [DOI] |
| 67. | Bennasser Y, Le SY, Yeung ML, Jeang KT. HIV-1 encoded candidate micro-RNAs and their cellular targets. Retrovirology. 2004;1:43. [PubMed] [DOI] |
| 68. | Appel N, Bartenschlager R. A novel function for a micro RNA: negative regulators can do positive for the hepatitis C virus. Hepatology. 2006;43:612-615. [PubMed] [DOI] |
| 69. | Couturier JP, Root-Bernstein RS. HIV may produce inhibitory microRNAs (miRNAs) that block production of CD28, CD4 and some interleukins. J Theor Biol. 2005;235:169-184. [PubMed] [DOI] |
| 70. | Huang J, Wang F, Argyris E, Chen K, Liang Z, Tian H, Huang W, Squires K, Verlinghieri G, Zhang H. Cellular microRNAs contribute to HIV-1 latency in resting primary CD4+ T lymphocytes. Nat Med. 2007;13:1241-1247. [PubMed] [DOI] |
| 71. | Jin WB, Wu FL, Kong D, Guo AG. HBV-encoded microRNA candidate and its target. Comput Biol Chem. 2007;31:124-126. [PubMed] [DOI] |
| 72. | Yeung ML, Bennasser Y, Jeang KT. miRNAs in the biology of cancers and viral infections. Curr Med Chem. 2007;14:191-197. [PubMed] [DOI] |