修回日期: 2008-01-17
接受日期: 2008-01-24
在线出版日期: 2008-02-08
目的: 通过序列分析确定抗胃癌抗体重链可变区(VH)的一级结构, 并借助同源模建方法模拟其三级结构.
方法: 从抗人胃癌噬菌体抗体库中筛选出VH基因, 并进行序列测定、翻译和分析. 利用计算机辅助蛋白质空间模拟技术, 采用同源模建、力学优化合理模建VH的三维空间结构.
结果: 序列比对分析表明获得的VH序列符合鼠抗体可变区特征, 通过Kabat分析确定了FR、CDR; 合理搭建了抗体重链可变区的空间构象, 并通过分子力学优化获得了稳定的三维结构.
结论: 所测得的VH一级结构和构建的三维空间结构均有较高的可靠性, 为进一步的生物学实验奠定了基础.
引文著录: 杨丽娟, 侯颖春, 药立波, 苏成芝. 抗胃癌抗体重链可变区序列分析及空间结构模拟. 世界华人消化杂志 2008; 16(4): 413-416
Revised: January 17, 2008
Accepted: January 24, 2008
Published online: February 8, 2008
AIM: To confirm the primary structure of heavy chain variable regions (VH) of antibody against human gastric cancer based on the sequence analysis method and to model its three-dimensional structure using homology modeling method.
METHODS: The VH gene selected from the phage display library of antibodies against human gastric cancer was sequenced and analyzed. Its three-dimensional structure was modeled with computer homology modeling techniques and optimized using molecular mechanism method.
RESULTS: The sequence of VH was in agreement with the characteristics of the mouse antibody variable region. The FR and CDR were determined by Kabat analysis. The spatial structure of the VH was constructed and optimized with molecular mechanism method to obtain the stable 3-D structure.
CONCLUSION: The primary and three-dimensional structures of VH are reasonable and reliable and lay the theoretical foundation for further biological experiments.
- Citation: Yang LJ, Hou YC, Yao LB, Su CZ. Analysis of primary structure and modeling of spatial structure of heavy chain variable region of antibody against human gastric cancer. Shijie Huaren Xiaohua Zazhi 2008; 16(4): 413-416
- URL: https://www.wjgnet.com/1009-3079/full/v16/i4/413.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i4.413
自人源化抗体Herceptin被美国FDA批准上市后, 更多的实践表明, 抗体可能成为较理想的肿瘤诊断和治疗的新型药物[1]. 本研究利用噬菌体抗体库技术, 绕过杂交瘤技术, 获得了鼠抗人胃癌重链可变区片段, 为今后胃癌的显像和导向治疗奠定了一定的基础. 然而, 由于其为鼠源性, 在应用过程中会引发HAMA反应而限制了其应用. 必须先进行人源化改造, 才能更好地应用于临床. 而进行人源化设计必须了解其三维结构. 因此本研究利用同源蛋白结构模建及分子力学优化技术, 采用Biosym公司开发的计算机辅助分子设计系统搭建了VH的三维结构.
细菌菌株E.coli TG1, 克隆及表达载体pCANTAB5E, 扩增VH可变区基因引物RS Primer Mix, S3和S6引物均为Pharmacia公司产品. 其他有关分子生物学试剂系华美公司和Promega公司产品. InsightⅡ(2005)软件包为MSI公司产品.
抗人胃癌单链抗体基因文库及噬菌体表面呈现文库的构建和筛选见文献[2].
1.2.1 VH基因序列的测定及分析: 从4 ℃贮存的阳性克隆菌种取100 μL接种于25 mL含氨苄青霉素(100 mg/L)的SOC培养液中, 30 ℃振荡培养过夜, 碱裂解法大量制备噬菌粒DNA, 经FPLC Hi Trap Q柱纯化, 经EcoRⅠ、HindⅢ双酶切鉴定后, 作为测序模板, 用S3和S6引物从基因两端测序. 将所测得的DNA序列通过http://www.expasy.ch进行氨基酸序列的翻译, 并通过http://www.ncbi.nlm.nih.gov/blast对其序列进行相似性比较.
1.2.2 VH三维结构模建及结构优化: 通过http://www.ncbi.nlm.nih.gov/blastp, 利用BLASTp[3]软件搜索蛋白质晶体结构数据库(PDB), 获得VH的同源蛋白, 利用InsightⅡ(2000)软件, 选择HOMOLOGY[4]程序包, 经过同源蛋白结构叠加以确定结构保守区(structural conserved region, SCR)和Loop区, 以该同源蛋白为模板对VH的主链、侧链进行结构模建. 模建获得的VH初始结构利用DISCOVERY-3[5]程序包, 应用CVFF/Gromos96力场通过最陡下降、共轭梯度、牛顿力学对初始模型进行分子力学优化及常温分子动力学动态模拟, 获得VH的三维结构的能量最低构象. 所有工作均在SGI Indigo 2工作站上完成.
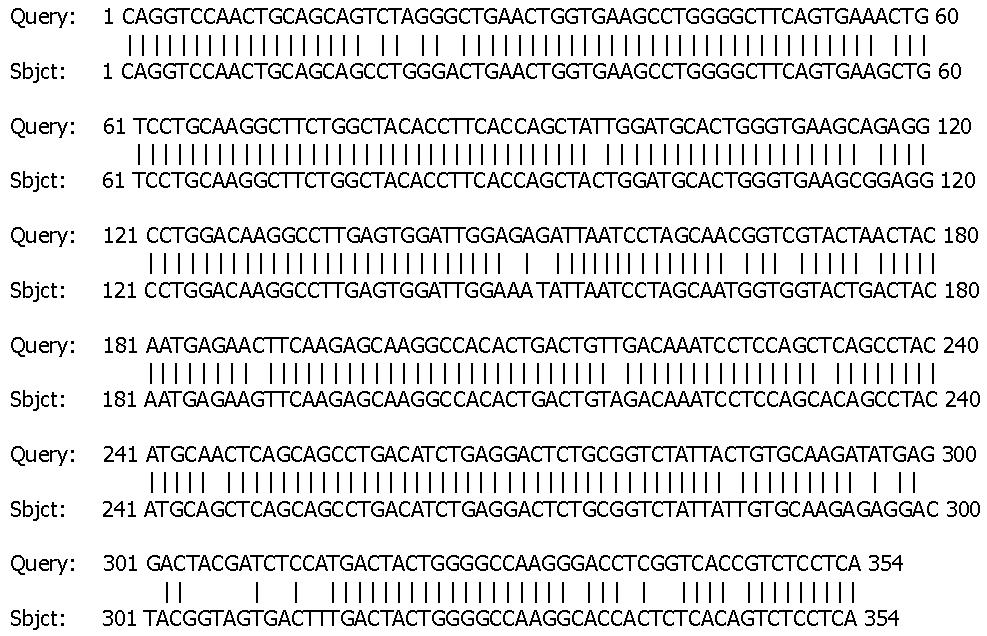
用S3和S6引物从基因两端测序, 测出相应的核苷酸序列并进行同源性分析(图1). 将VH基因序列输入计算机与Internet网中的所有基因数据库中的已知基因进行同源性比较, 其同源序列均为鼠Ig重链可变区基因, 与之同源性最高的为MM2F2A(Score 1464).
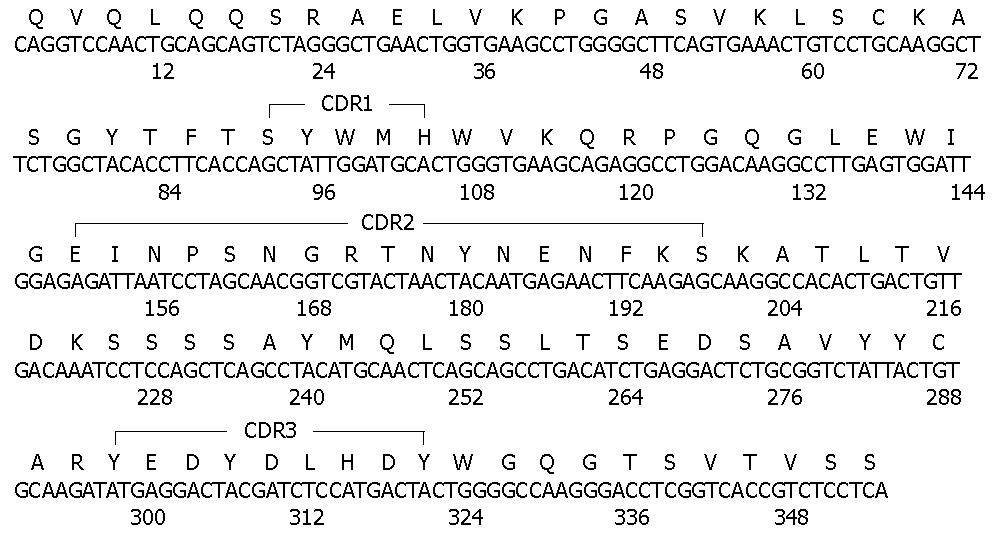
VH基因长度为354 bp, 系一开放读框, 利用http://www.expasy.ch对其进行序列分析, 对应的翻译模式见图2. 根据推导的氨基酸序列进行Kabat分类分析, 确定4个FR区和3个CDR区(图2). 通过确定的FR、CDR区以及抗体结构所必须的两个恒定半胱氨酸残基, 表明其结构符合鼠Ig重链可变区的氨基酸序列特征.
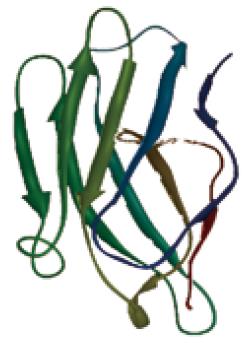
以VH序列为探针, 用BLASTp程序在PDB库中搜索VH的同源蛋白. 选择模板蛋白免疫球蛋白重链(PDB号: 1E4X)和目标蛋白VH两者具有85%的序列相似性. 将1E4X和VH进行序列比对, 考虑残基的理化性质, 确定6个SCR区, 5个Loop区. 利用模板蛋白1E4X重链可变区晶体结构的Cα原子坐标确定了VH主链构象并完成了侧链安装, 组装出完整的VH三维结构. 对模建的VH三维结构在CVFF力场下, 经过最陡下降, 牛顿力学等方法进行能量的分子力学优化, 获得能量最低构象. 该稳定构象为全-β模式, N端5个反平行β片层, C端6个反平行β片层, N端2个高度保守的半胱氨酸残基形成1对二硫键结构(图3). 模建获得的VH空间构象与1FVB晶体结构进行主链C原子的Cα迭合, 迭合后的均方根位移RMS = 0.15 nm, 表明理论模建的VH空间结构是可靠的. 应用Profile-3D对优化后的VH三维结构进行理论评估. 结果显示, 模建结构中无不兼容的残基, 说明结构较合理, 而且优化后结构更合理.
在肿瘤的诊断和治疗方面, 小分子抗体具有极高的理论和应用价值. 因为其具有分子小, 免疫原性弱, 穿透力强易于进入实体瘤周围的微循环, 血清和全身廓清快, 无肾脏蓄积作用, 肿瘤显像时清晰度高, 可作载体与药物、同位素、毒素等结合之优点[6]. 本研究从已构建的抗人胃癌单链抗体基因文库及噬菌体表面呈现文库中筛选出了抗人胃癌VH基因, 经序列测定及同源性分析表明其结构符合鼠Ig重链可变区的氨基酸序列特征, 为今后的基因工程改造提供了条件.
为了能更好地应用于临床, 必须将鼠源性的抗体先进行人源化改造. 而进行人源化设计必须了解其三维结构. 人们通过对类似蛋白质空间结构的对比发现, 蛋白质的三维结构较其一级序列更为保守. 氨基酸残基序列有50%相同的蛋白质, 约有90%的Cα原子偏差不超过0.3 nm, 均方根偏差约0.1 nm, 氨基酸的残基替换通常发生在蛋白质表面回折区域. 蛋白质主链结构, 特别是疏水中心的结构受序列变异的影响很小[7]. 因此, 用类似蛋白来预测目标蛋白的空间构象是比较可靠的. 根据此原理, 人们已成功预测了许多目标蛋白, 在抗体的结构预测中尤为突出[8-14]. 本研究利用Biosym公司的Homology软件包, 同源模建了用噬菌体抗体库技术获得的抗人胃癌抗体VH结构域的三维空间结构. 经过分子力学和分子动力学优化, Profile-3D的合理性评价, 提示所获得的VH结构域的三维结构有较高的可靠性, 为今后制备人源化单链抗体奠定了基础.
在肿瘤的诊断治疗中, 抗体占有很重要的作用. 本研究利用噬菌体抗体库技术, 绕过杂交瘤技术, 获得了鼠抗人胃癌重链可变区片段, 为今后胃癌的显像和导向治疗奠定了一定的基础.
李奇林, 教授, 南方医科大学珠江医院消化内科
目前所获得的抗体大部分为鼠源性, 在应用过程中会引发HAMA反应而限制了其应用, 必须先进行人源化改造, 才能更好地应用于临床, 如何进行人源化改造是亟待研究的问题.
人们通过对类似蛋白质空间结构的对比发现, 蛋白质的三维结构较其一级序列更为保守. 氨基酸残基序列有50%相同的蛋白质, 约有90%的Cα原子偏差不超过0.3 nm, 均方根偏差约0.1 nm, 氨基酸的残基替换通常发生在蛋白质表面回折区域. 蛋白质主链结构, 特别是疏水中心的结构受序列变异的影响很小. 因此, 用类似蛋白来预测目标蛋白的空间构象是比较可靠的.
本文先从噬菌体抗体库着手, 得到抗体后, 利用三维重建得到空间结构, 为人源化改造奠定基础.
本研究具有新颖性, 有一定的意义.
编辑: 程剑侠 电编: 何基才
| 1. | Bradbury AR, Marks JD. Antibodies from phage antibody libraries. J Immunol Methods. 2004;290:29-49. [PubMed] [DOI] |
| 3. | Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 1997;25:3389-3402. [PubMed] [DOI] |
| 4. | Molecular Simulation Inc. Homology 2000.0 User Guide, Version 2000[EB] San Diego. 2000;. |
| 5. | Molecular Simulation Inc. Discover-3 User Guide, Version 2000[EB] San Diego. 2000;. |
| 6. | Colcher D, Bird R, Roselli M, Hardman KD, Johnson S, Pope S, Dodd SW, Pantoliano MW, Milenic DE, Schlom J. In vivo tumor targeting of a recombinant single-chain antigen-binding protein. J Natl Cancer Inst. 1990;82:1191-1197. [PubMed] [DOI] |
| 7. | Blundell TL, Sibanda BL, Sternberg MJ, Thornton JM. Knowledge-based prediction of protein structures and the design of novel molecules. Nature. 1987;326:347-352. [PubMed] [DOI] |
| 8. | Fontayne A, Vanhoorelbeke K, Pareyn I, Van Rompaey I, Meiring M, Lamprecht S, Roodt J, Desmet J, Deckmyn H. Rational humanization of the powerful antithrombotic anti-GPIbalpha antibody: 6B4. Thromb Haemost. 2006;96:671-684. [PubMed] |
| 9. | Paula S, Monson N, Ball WJ. Molecular modeling of cardiac glycoside binding by the human sequence monoclonal antibody 1B3. Proteins. 2005;60:382-391. [PubMed] [DOI] |
| 10. | Thompson AJ, Price KL, Reeves DC, Chan SL, Chau PL, Lummis SC. Locating an antagonist in the 5-HT3 receptor binding site using modeling and radioligand binding. J Biol Chem. 2005;280:20476-20482. [PubMed] [DOI] |
| 11. | Méndez R, Leplae R, Lensink MF, Wodak SJ. Assessment of CAPRI predictions in rounds 3-5 shows progress in docking procedures. Proteins. 2005;60:150-169. [PubMed] [DOI] |
| 12. | Gee GV, Tsomaia N, Mierke DF, Atwood WJ. Modeling a sialic acid binding pocket in the external loops of JC virus VP1. J Biol Chem. 2004;279:49172-49176. [PubMed] [DOI] |
| 13. | Arndt MA, Krauss J, Schwarzenbacher R, Vu BK, Greene S, Rybak SM. Generation of a highly stable, internalizing anti-CD22 single-chain Fv fragment for targeting non-Hodgkin's lymphoma. Int J Cancer. 2003;107:822-829. [PubMed] [DOI] |