修回日期: 2008-10-28
接受日期: 2008-11-03
在线出版日期: 2008-12-08
本文主要阐明了代谢组学的概念, 代谢与肿瘤的关系, 介绍代谢组学的研究状况及研究技术, 着重讲述了其在肿瘤生物学领域研究中的应用, 归纳了代谢组学在肿瘤的早期诊断、治疗和预后评估中的最新应用进展.
引文著录: 马延磊, 秦环龙. 代谢组学及其在肿瘤生物学研究中的应用进展. 世界华人消化杂志 2008; 16(34): 3877-3883
Revised: October 28, 2008
Accepted: November 3, 2008
Published online: December 8, 2008
The primary aim of this review is to introduce the research status of metabonomics/metabolomics and the application of metabonomics in oncobiology research. At first, the concept of metabonomics and the relationship between metabonomics and tumors are elucidated in detail. Then the research technologies of metabonomics in oncobiology are introduced. Finally, the latest advances in the application of metabonomics in early diagnosis, treatment and prognosis of tumors are summarized.
- Citation: Ma YL, Qin HL. Metabonomics and its application in oncobiology research. Shijie Huaren Xiaohua Zazhi 2008; 16(34): 3877-3883
- URL: https://www.wjgnet.com/1009-3079/full/v16/i34/3877.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i34.3877
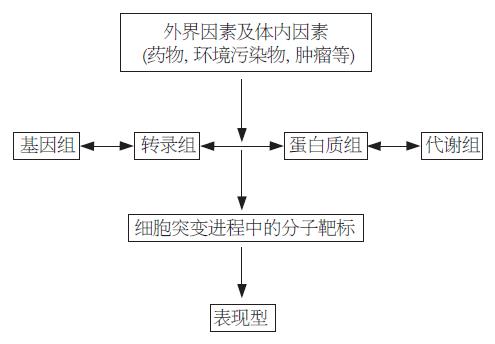
随着人类基因组测序的完成, 生命科学的主要任务已经转为功能基因组分析, 到目前为止, 功能基因组的主要目标是基因产物以及可能的治疗靶标的确定, 研究的策略主要集中在转录组学和蛋白质组学. 这些"组学"(-omics)方法在肿瘤生物学领域中的应用也取得了一些成功, 例如: 新的肿瘤亚型的鉴定, 相应基因水平和蛋白水平特定类型肿瘤标志物的鉴定等. 但是他们并不能提供对于疾病生化改变所反映的终点诊断标志物及最终的诊断信息. 于是出现了代谢组学(metabolomics/metabonomics), 他处于基因组和蛋白质组的下游, 是基因组和蛋白质组的补充, 能够更为灵敏地鉴定出基因改变、疾病和环境因素作用所产生的特定代谢型(metabotype)[1-2](图1). 因此与其他"组学"相比, 代谢组学在环境变化对机体产生作用的研究中更具优势. 目前, 代谢组学被广泛应用于各种研究领域, 尤其是在肿瘤生物学领域逐渐成为近年来研究的热点.
代谢组学是采用高通量化学分析技术, 如质谱(mass spectrometry, MS)技术和核磁共振技术(nuclear magnetic resonance, NMR)并结合化学计量学方法对生物体系(细胞、组织或生物体)在给定时间和条件下所有小分子代谢物质的定量分析, 关注的对象是分子质量1000以下的小分子化合物, 从而定量描述生物体内源性代谢物整体代谢变化情况及其对内因和外因应答变化规律的一门新兴科学[1,3]. 其中"metabonomics"主要是对处于不同发育阶段或不同生长环境的生物系统进行整体及其动态变化规律的研究[4]; 而"metabolomics"主要是研究细胞层次上的所有小分子代谢物, 强调静态代谢物的分析[5]. Fiehn et al[6]认为metabolomics是metabonomics的一部分.
代谢组学的分析技术强调整体分析过程, 要求测定生物体系中所有代谢产物的信息, 然而要实现这一目的, 目前的技术仍然面临着极大的挑战[7]. 如: 利用NMR为基础的方法, 在组织的提取液中可以检测到20-40个代谢物, 尿样中可检测到100-200个代谢物, 而应用灵敏度更高的气质联用(gas chromatography-mass spectrometry, GC-MS), 在这些样品中大约1000个代谢物可被检测到. 目前各种不同的分离及分析手段已应用于代谢组学的研究中, 这些代谢组学分析技术的比较见表1.
| 分析技术 | 概述 | 优点 | 缺点 |
| 气质联用质谱(GC-MS[8]) | 用GC分离代谢物, 后MS鉴定, | 高灵敏度、高重复性, | 样品的准备较为耗时, |
| 可查库搜索, 植物代谢组研究 | 费用较低 | 并不适合所有化合物分离 | |
| 的优选方法 | |||
| 液质联用质谱[LC-MS | 除了在分离上用LC分离, | 越来越多地用于替代 | 费用较高, 较难操作, 且非 |
| (Lipid chromatography- | 其他与GC-MS相似 | GC-MS作为样品分离 | 极性物质难以分离 |
| mass spectrometry)[9]] | 的手段, 耗时很少, | ||
| 高灵敏度 | |||
| 核磁共振(NMR[10]) | 广泛应用于药学和疾病中血 | 非破坏性的技术, 完全 | 与MS相比灵敏度较低, |
| 和尿样分析 | 自动化, 可重复性好, | 共振波谱的代谢物难以 | |
| 从简单的一维图谱中 | 鉴定 | ||
| 较易鉴定代谢物 | |||
| 傅里叶变换红外光谱 | 用代谢物的震动频率产生 | 高通量的首选筛选方法, | 难以鉴定引起变化的代谢 |
| 测定法[FT-IR (Fourier- | 代谢指纹图谱 | 费用较低, 较为准确 | 物, 对同类化合物的代谢 |
| Transform infrared | 产物分辨率低 | ||
| spectrometry)[11]] | |||
| 拉曼光谱测定法[Raman | FT-IR和红外光谱技术的拓展, | 由于水有微弱的拉曼 | 分辨率低, 较难分辨同类 |
| spectroscopy[12]] | 依赖于激光照射后分散的 | 光谱, 因此可以观测到 | 化合物的代谢产物 |
| 光点筛选 | 许多的官能团 | ||
| 代谢物微阵列 | 用96-孔板监测系统进行 | 在既定的条件下是很好 | 难以对未知的和异源性代 |
| (Metabolite arrays[13]) | 表性分析和筛选 | 的筛选工具 | 谢产物进行筛选 |
| 薄层色谱测定法(TLC | 用来追踪在不同培养条件下 | 便宜, 便捷 | 存在内在测定的变异, |
| Thin-layer | 大肠杆菌中14C的糖代谢产物 | 限于特定条件下代谢物 | |
| chromatography[14]) | 的定量测定 |
Fiehn et al[15]将代谢组学的研究目的和策略分为4个层次, 即: (1)代谢物靶标分析(metabolite target analysis)[8]: 针对生物样品中的一个或数个特定代谢物进行有选择性的定性和定量分析. (2)代谢谱分析(metabolite profiling analysis)[16-17]: 针对预设代谢物, 采用特定的分析技术对其代谢过程中的变化进行定量和定性分析. (3)代谢物组(matabonom)[1-2,10,18]: 生物系统对体系内外因素做出动态应答的所有代谢物小分子. (4)代谢指纹分析(metabolic fingerprinting)[19]: 对样品进行整体定性分析, 比较谱图差异, 并对样品中的差异代谢物进行快速鉴别和分类, 但并不分析或测量具体组分.
70年前经典的生物化学研究已表明: 相对于正常细胞, 肿瘤细胞出现了代谢谱的改变以及葡萄糖吸收率增高和糖酵解过程的增强, 尽管这些代谢方面的改变并非是引起肿瘤发生的本源, 但是他们却赋予不同类型肿瘤细胞一个共同的特点-能够使肿瘤细胞逃避死亡和免疫侵袭.
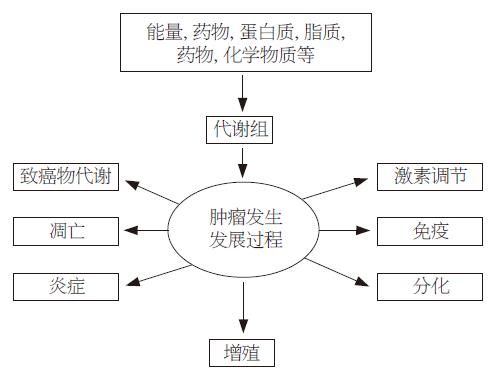
目前认为肿瘤是环境与宿主内外因素交互作用的结果, 据估计80%以上的恶性肿瘤与环境因素相关. 而这些环境因素所导致的肿瘤相关基因和蛋白的变化, 最终会反映在肿瘤的代谢组变化上. 因为代谢组反映了基因组和蛋白质组变化所引起的共同"终点"代谢型小分子的变化[5]. 近来研究表明: 大量遗传改变引起的肿瘤也直接影响了肿瘤细胞的糖酵解过程[20]. 肿瘤发生和进展过程中各种因素间平衡的变化与其代谢组的变化密切相关(图2).
与正常组织相比, 恶性肿瘤组织的代谢状态较旺盛. 由于体内外各种因素的影响, 其合成DNA和RNA酶的活性增高, 蛋白质的合成代谢及分解代谢均增强, 且合成代谢超过了分解代谢, 甚至利用正常组织蛋白质的分解产物用于合成肿瘤本身所需的营养物质, 从而导致机体呈现出严重的恶病质状态. 肿瘤细胞不仅仅在能量的代谢率上大幅度提高, 而且当他们受损时所导致的"自杀"(commit suicide)行为也会大大减少. 其中肿瘤细胞的死亡和代谢相关的分子机制研究近来成为肿瘤生物学领域研究的热点[21]. Danial et al[22]指出在肿瘤细胞死亡相关的代谢调节过程中, 主要是通过一杀伤性蛋白(Bcl-xl/Bcl-2-associated death, BAD)来调节. BAD蛋白对肿瘤细胞的代谢存在重要的影响, 最主要的是其刺激了细胞内葡萄糖的转换, 己糖激酶和线粒体的重组以及糖酵解过程的加速.
肿瘤细胞不仅仅表现出旺盛的代谢状态, 而且肿瘤的发生和发展也离不开其周围的微环境. 肿瘤生长赖以生存的周围微环境主要是一些关键代谢物(critical metabolites)[23-26], 如氧分、葡萄糖和其他的一些营养性生长因子, 其周围的微环境呈现出明显的酸性环境, 构成这种酸性环境的物质主要是乳酸盐(lactate)等一些小分子. 大量的肿瘤细胞有很强的增殖性, 实体瘤细胞也呈现出明显的缺氧现象, 然而肿瘤细胞在增殖过程中却能适应这种状态, 这主要是由于肿瘤细胞在缺氧或低氧状态能够通过糖酵解代谢的增加把葡萄糖作为提供滋生能量的源泉[27-28]. 小分子代谢组分在肿瘤形成过程中发挥了重要的作用, 大量研究已经发现了这些变化的小分子代谢物, 例如: 大量研究发现肿瘤细胞周围的低氧或缺氧以及低血糖环境激发了肿瘤血管的发生, 诱导了血管源性生长因子(vascular endothelial growth factor, VEGF)的大量表达, VEGF诱发了新的微血管形成, 从而为肿瘤带来了更多的营养物, 促进了肿瘤体积的增长[21,29-30].
在肿瘤发生的早期阶段利用代谢组学技术将可能监测特定代谢过程中代谢物的变化或波动情况, 预测肿瘤的进展, 或是监测肿瘤对于干预的反应[31-33]. 代谢组学技术的定量分析联合先进的统计分析方法, 能够得出特定器官早期变化的特征性代谢谱[34-35]. 通过特定代谢物的变化来评估既定肿瘤的分子病因, 鉴定在生物学效应下的代谢物变化, 并且辅助鉴定肿瘤易感性的特征. 生物标志物的发现将可能找出肿瘤细胞特异性损伤的特征性变化情况, 从而将可能用于制定对易感人群的特征性干预策略. 理解代谢物变化情况与肿瘤的生长、停滞、凋亡和分化间的关系将有助于发现肿瘤相关的代谢标志物, 从而提高肿瘤的早期诊断. 例如脂质和胆固醇变化谱是细胞膜破坏的标志, 这些变化往往表明细胞正在经受各种因素刺激而导致凋亡的过程. 同样, 抗癌剂对肿瘤细胞生长的抑制作用也在糖类和氨基酸类代谢途径的变化中得以体现. 目前随着代谢组学方法的不断进展, 越来越多的肿瘤相关代谢标志物在肿瘤疾病中被鉴定(表2), 以此用于肿瘤的分型、诊断、治疗和预后评估.
| 代谢标志物 | 相关代谢功能 | 相关肿瘤特征 |
| 缬氨酸(Alanine) | 与乳酸盐相结合, 在缺氧状态下组织中显著增高; | 肝癌和脑肿瘤, 包括星形胶质瘤, 神经胶质瘤, |
| 通过丙酮酸盐的转氨基反应产生以致于阻止 | 脑膜瘤及神经上皮的肿瘤 | |
| 乳酸盐的显著升高[36] | ||
| 饱和脂质 | 细胞膜的重要组成成分, 由NMR的脂质峰可以 | 其含量的变化水平与增殖、炎症恶性肿瘤、 |
| (Saturated lipids) | 确定其是在细胞膜的功能区还是在胞质囊泡内 | 坏死和凋亡紧密相关[37-41] |
| CCMs | 包括胆碱、磷酸胆碱、磷酸卵磷脂等, | 在凋亡和坏死期间其水平发生变化, 目前在各 |
| 均是细胞膜的关键构成组分 | 种类型肿瘤中已发现其水平的改变. 如: 脑肿 | |
| 瘤、肉瘤、前列腺癌和肝癌[42-45] | ||
| 甘氨酸(Glycine) | 形成嘌呤所必须的前体 | 随着HIF-1信号通路的破坏其水平显著下降[46] |
| 乳酸盐(Lactate) | 糖酵解的终产物 | 在缺血和低氧期间其水平显著增高; 随其增高 |
| 在弱血管化的肿瘤出现细胞内pH值显著降低; | ||
| 其增加率与肿瘤的范围即特定肿瘤的类型有关[47] | ||
| 肌醇(Myo-inositol) | 包含在渗透调节和容积调控的过程中 | 其水平在结肠腺癌、神经胶质瘤、神经鞘瘤、 |
| 卵巢癌、星形胶质瘤和子宫内膜癌增高; 而在 | ||
| 乳腺癌中降低[48-50] | ||
| 核苷酸 | 产生DNA和RNA; 在脂肪酸和糖代谢 | 在神经胶质瘤细胞凋亡过程中其水平增高[51]; |
| (Nucleotides) | 中是一个关键的中间代谢物; 在ATP | CDP-胆碱在凋亡期间水平也增高[52-53] |
| 浓聚物中的改变是肿瘤活力状态的 | ||
| 重要指示物 | ||
| 多不饱和脂肪酸 | 细胞膜尤其是线粒体的组分 | 凋亡期间, 在胶质瘤、去分化的多形态脂肪瘤 |
| (PUFAs) | 中增加[38,54-55] | |
| 牛磺酸(Taurine) | 与渗透和容量调节相关; 是抗氧化剂, | 在鳞状细胞瘤、前列腺癌和肝脏次生肿瘤中 |
| 阻止细胞受自由基损伤 | 增加[56-57] |
由于肿瘤会导致机体病理生理的变化, 同时机体的代谢产物也会相应地变化, 对这些变化的代谢产物进行相应的代谢组学分析, 将能够更好地理解病变过程及机体内物质的代谢途径, 从而发现肿瘤相关的生物标志物, 并辅助临床诊断. 代谢组学也将从机体的动态代谢途径中寻找出具有特异性诊断价值的肿瘤生物标志物. 其在肿瘤诊断方面具有独特优势: 其一, 只需采集机体体液, 易于临床取材和获得动态观察结果; 其二, 癌细胞的产生与肿瘤宿主微环境密切相关, 调控癌细胞生长所必须的各种氨基酸、肽类、脂肪酸、小分子代谢物及细胞分裂产物在组织液中的浓度远大于细胞内.
Odunsi et al[58]应用H-NMR和PCA及SIMCA技术对38例卵巢上皮性癌, 12例卵巢良性囊肿, 53例正常妇女的血清的代谢物分析, 并且用受试者工作特征(ROC)曲线对灵敏度和特异度权衡, 此方法对正常绝经前妇女、卵巢良性肿瘤和卵巢上皮性癌的识别率达100%, 对绝经后妇女和卵巢癌的识别率分别为97.4%和97%, 并且成功诊断出2例Ⅰ期卵巢癌患者. Yang et al[59]采用最新技术魔角自转核磁共振分光检定法(magic-angle spinning 1H nuclear magnetic resonance spectroscopy, HPMAS)并结合主成份分析(PCA)来研究人肝癌的特征性代谢模式, 结果发现了在肝癌中乳酸盐、谷氨酸、谷氨酰胺、甘氨酸、亮氨酸、丙氨酸和磷酸乙醇胺(PE)等代谢产物相对于邻近正常组织明显升高, 而甘油三酯、葡萄糖和糖原呈现出明显的下降, 得出的结果再利用PCA进行分析, 从而可以成功区分肝癌和正常组织, 表明这些代谢小分子簇可作为肝癌早期诊断和预后的生物标志物. 最近Fang et al[60]应用1H-NMR(核磁共振技术)研究SD大鼠慢性胰腺炎和胰腺癌完整胰腺组织的代谢谱差异, 结果显示: 与胰腺炎相比, 在胰腺癌中磷酸胆碱和甘油磷酸胆碱显著降低, 而亮氨酸、异亮氨酸、缬氨酸、乳酸盐和丙氨酸呈现出明显升高, 同时结合主成份分析(PCA)方法成功区分出了胰腺癌和慢性胰腺炎SD大鼠.
代谢组学分析亦广泛应用到鉴别诊断中. Xu et al[61]采用毛细管电泳方法(CE), 通过代谢靶标分析, 以尿中13-15种核苷浓度为数据矢量, 用PCA法处理数据, 对分别患有10多种癌症的68位癌症患者和54位正常人进行分类研究, 识别率达72%. 对用HPLC法测定206位正常人和296位肿瘤患者尿中15种核苷排放水平进行研究, 也得到类似的结果. 采用人工神经元网络软件对数据进行处理, 对肿瘤患者的识别率可达83%. 最近国外学者Kim et al[62]在综述了大量代谢组学应用于肿瘤的研究后, 指出利用代谢组学技术方法可以预测早期肿瘤细胞表型的改变, 从而使得在肿瘤细胞表型改变前, 即在肿瘤的代谢组发生微小的改变时做出了诊断, 从而在很大程度上可发现新的早期诊断特征性代谢小分子, 作为肿瘤早期诊断的生物标志物.
代谢组学的出现打破了传统的以手术、放疗和化疗为主的肿瘤治疗方法, 通过对药物代谢动力学及对药物所引起的内源性代谢物变化方面的研究, 使得人们系统的、整体的来认识机体代谢谱的变化以为他能够更直接地以此来针对肿瘤的异质性和患者的个体差异实现个体化治疗, 同时指导抗肿瘤药物的用量以及评价治疗效果和安全性.
Sasano et al[63]在乳腺癌内分泌治疗的相关研究中, 指出瘤内代谢与雌激素合成在雌激素依赖性乳腺癌的发生、发展中发挥着关键的作用, 尤其是绝经后乳腺癌的发生. 瘤内芳香化酶作为雌激素来源的重要转化动力, 而其他酶类如: 17β-羟基类固醇脱氢酶同工酶(17beta-HSD isozymes)、雌激素硫酸酯酶(STS)和雌激素磺基转移酶(EST)也在雌激素生成过程中起到了重要作用. 最后得出结论, 通过使用芳香化酶(尤其是17beta-HSD isozymes)抑制剂并监测瘤内其他酶系(尤其是STS)和相关微环境的变化情况, 可以指导个体化的治疗. Ippolito et al[64]在利用MRS研究前列腺神经内分泌癌所衍生的前列腺神经内分泌癌细胞(prostate neuroendocrine cell, PNEC)中, 发现了与能量代谢密切相关的小分子代谢标志物丙二醇(propylene glycol, PG)和γ-氨基丁酸(gamma-aminobutyric acid, GABA), 两者在癌细胞中的含量均明显增高, 与细胞间通讯、能量利用和细胞增殖密切相关, 当GABA、谷氨酰胺(glutamine, Gln)/甘氨酸(glycine, Gly)的受体受刺激被激活时, PNEC的增殖速度将显著加快, 试验中将三种抑制GABA通路的抑制药物(amiloride、carbidopa和flumazenil)注射入PNEC肿瘤嫁接的裸鼠, 结果出现了肿瘤生长的显著抑制, 利用471例神经内分泌肿瘤的瘤组织进行基因芯片分析, 结果也显示了GABA代谢通路重要组分包括ABP-1在神经内分泌瘤中显著升高, 该项代谢组学研究对于神经内分泌型肿瘤的治疗有着重要的临床指导意义.
Ichikawa[65]在讨论了遗传因素对氟尿嘧啶治疗效果的影响后, 指出5-FU相关代谢途径中某些酶活性的差异能显著影响其代谢范围和化疗效果, 这些酶主要包括: 胸核苷酸合酶、胸苷磷酸化酶、二氢嘧啶脱氢酶和乳酸磷酸核糖苷转移酶, 他们的基因表达阳性与5-FU在转移性胃癌的化疗效果密切相关, 因此利用代谢组学技术监测这些酶的变化水平将有可能准确判断和预测其化疗患者的预后, 在胃癌化疗患者的预后评估中具有重要的临床指导意义.
代谢组学是基因组学、转录组学和蛋白质组学有力的补充, 是系统生物学不可缺少的一部分, 虽是一门新兴科学, 但其发展迅速, 研究技术方法也越来越得以完善, 应用领域也越来越广, 尤其在肿瘤生物学领域中的研究日益升温. 基于肿瘤中血、尿相关特征性小分子代谢标志物的异常来诊断特定肿瘤的方法也正向着临床实用性方向发展. 随着研究的逐步深入, 代谢组学将在功能基因组学中发挥更大的作用, 同时也提供了一个了解肿瘤相关基因表型的独特途径, 在肿瘤代谢相关的生物学领域中必将发挥更大的作用.
随着人类基因组测序的完成, 生命科学的主要任务已经转变为功能基因组分析, 研究的策略主要集中在转录组学和蛋白质组学, 然而他们并不能提供对于疾病生化改变所反映的终点诊断标志物和最终的诊断信息. 因此出现了代谢组学, 他处于基因组和蛋白质组的下游, 是二者的补充, 能够更为敏感地鉴定出基因改变、疾病和环境因素作用所产生的特定代谢型. 目前代谢组学被应用于各种研究领域, 尤其是在肿瘤生物学研究领域中的应用成为近年来研究的热点.
刘海林, 主任医师, 上海交通大学医学院附属第九人民医院消化科
通过代谢组学的各种分析技术, 检测肿瘤患者体液中的各种代谢小分子, 确定肿瘤患者的特征性代谢模式, 将有助于实现肿瘤的早期诊断, 并能够鉴定出肿瘤的类型及其分化程度, 从而对肿瘤的诊断、治疗和预后有着重大的影响, 对传统肿瘤标志物提出了挑战.
目前代谢组学在各种肿瘤方面的研究越来越多, 同时也越来越深入. 国内外的研究主要集中在代谢组学在肿瘤诊断、治疗和预后中的应用方面. 同时代谢组学在生命科学的其他领域(如: 药物毒理学、环境科学、检验医学、食品学等)方面也有较多的研究.
本文主要阐明了代谢组学的概念, 代谢与肿瘤的关系, 介绍代谢组学的研究状况, 同时对目前代谢组学的各种研究技术进行了对比, 着重讲述了代谢组学在肿瘤生物学领域研究中的应用, 归纳了代谢组学在肿瘤的早期诊断、治疗和预后评估中的最新应用进展, 内容较新颖.
本文介绍代谢组学在肿瘤生物学研究中的进展, 内容较新, 具有一定的新颖性.
编辑: 史景红 电编: 何基才
| 1. | Nicholson JK, Lindon JC, Holmes E. 'Metabonomics': understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica. 1999;29:1181-1189. [PubMed] [DOI] |
| 2. | Nicholson JK, Connelly J, Lindon JC, Holmes E. Metabonomics: a platform for studying drug toxicity and gene function. Nat Rev Drug Discov. 2002;1:153-161. [PubMed] [DOI] |
| 3. | Raamsdonk LM, Teusink B, Broadhurst D, Zhang N, Hayes A, Walsh MC, Berden JA, Brindle KM, Kell DB, Rowland JJ. A functional genomics strategy that uses metabolome data to reveal the phenotype of silent mutations. Nat Biotechnol. 2001;19:45-50. [PubMed] [DOI] |
| 4. | Sumner LW, Mendes P, Dixon RA. Plant metabolomics: large-scale phytochemistry in the functional genomics era. Phytochemistry. 2003;62:817-836. [PubMed] [DOI] |
| 5. | Fiehn O. Metabolomics--the link between genotypes and phenotypes. Plant Mol Biol. 2002;48:155-171. [PubMed] [DOI] |
| 6. | Fiehn O, Kopka J, Dörmann P, Altmann T, Trethewey RN, Willmitzer L. Trethewey RN, Willmitzer L. Metabolite profiling for plant functional genomics. Nat Biotechnol. 2000;18:1157-1161. [PubMed] [DOI] |
| 7. | Griffin JL, Shockcor JP. Metabolic profiles of cancer cells. Nat Rev Cancer. 2004;4:551-561. [PubMed] [DOI] |
| 8. | Seger C, Sturm S. Analytical aspects of plant metabolite profiling platforms: current standings and future aims. J Proteome Res. 2007;6:480-497. [PubMed] [DOI] |
| 9. | Plumb RS, Stumpf CL, Granger JH, Castro-Perez J, Haselden JN, Dear GJ. Use of liquid chromatography/time-of-flight mass spectrometry and multivariate statistical analysis shows promise for the detection of drug metabolites in biological fluids. Rapid Commun Mass Spectrom. 2003;17:2632-2638. [PubMed] [DOI] |
| 10. | Lindon JC, Holmes E, Nicholson JK. So what's the deal with metabonomics? Anal Chem. 2003;75:384A-391A. [PubMed] [DOI] |
| 11. | Oliver SG, Winson MK, Kell DB, Baganz F. Systematic functional analysis of the yeast genome. Trends Biotechnol. 1998;16:373-378. [PubMed] [DOI] |
| 12. | Hanlon EB, Manoharan R, Koo TW, Shafer KE, Motz JT, Fitzmaurice M, Kramer JR, Itzkan I, Dasari RR, Feld MS. Prospects for in vivo Raman spectroscopy. Phys Med Biol. 2000;45:R1-R59. [PubMed] [DOI] |
| 13. | Bochner BR, Gadzinski P, Panomitros E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Res. 2001;11:1246-1255. [PubMed] [DOI] |
| 14. | Tweeddale H, Notley-McRobb L, Ferenci T. Effect of slow growth on metabolism of Escherichia coli, as revealed by global metabolite pool ("metabolome") analysis. J Bacteriol. 1998;180:5109-5116. [PubMed] |
| 15. | Fiehn O. Combining genomics, metabolome analysis, and biochemical modelling to understand metabolic networks. Comp Funct Genomics. 2001;2:155-168. [PubMed] [DOI] |
| 16. | Fernie AR, Trethewey RN, Krotzky AJ, Willmitzer L. Metabolite profiling: from diagnostics to systems biology. Nat Rev Mol Cell Biol. 2004;5:763-769. [PubMed] [DOI] |
| 17. | Niwa T. Metabolic profiling with gas chromatography-mass spectrometry and its application to clinical medicine. J Chromatogr. 1986;379:313-345. [PubMed] |
| 18. | Nicholson JK, Wilson ID. Opinion: understanding 'global' systems biology: metabonomics and the continuum of metabolism. Nat Rev Drug Discov. 2003;2:668-676. [PubMed] [DOI] |
| 19. | Ellis DI, Goodacre R. Metabolic fingerprinting in disease diagnosis: biomedical applications of infrared and Raman spectroscopy. Analyst. 2006;131:875-885. [PubMed] [DOI] |
| 20. | Costello LC, Franklin RB. 'Why do tumour cells glycolyse?': from glycolysis through citrate to lipogenesis. Mol Cell Biochem. 2005;280:1-8. [PubMed] [DOI] |
| 22. | Danial NN, Gramm CF, Scorrano L, Zhang CY, Krauss S, Ranger AM, Datta SR, Greenberg ME, Licklider LJ, Lowell BB. BAD and glucokinase reside in a mitochondrial complex that integrates glycolysis and apoptosis. Nature. 2003;424:952-956. [PubMed] [DOI] |
| 23. | Cairns R, Papandreou I, Denko N. Overcoming physiologic barriers to cancer treatment by molecularly targeting the tumor microenvironment. Mol Cancer Res. 2006;4:61-70. [PubMed] [DOI] |
| 24. | Sutherland RM. Cell and environment interactions in tumor microregions: the multicell spheroid model. Science. 1988;240:177-184. [PubMed] [DOI] |
| 25. | Liotta LA, Kohn EC. The microenvironment of the tumour-host interface. Nature. 2001;411:375-379. [PubMed] [DOI] |
| 26. | Helmlinger G, Yuan F, Dellian M, Jain RK. Interstitial pH and pO2 gradients in solid tumors in vivo: high-resolution measurements reveal a lack of correlation. Nat Med. 1997;3:177-182. [PubMed] [DOI] |
| 28. | Shaw RJ. Glucose metabolism and cancer. Curr Opin Cell Biol. 2006;18:598-608. [PubMed] [DOI] |
| 29. | Semenza GL. Targeting HIF-1 for cancer therapy. Nat Rev Cancer. 2003;3:721-732. [PubMed] [DOI] |
| 30. | Semenza GL, Roth PH, Fang HM, Wang GL. Transcriptional regulation of genes encoding glycolytic enzymes by hypoxia-inducible factor 1. J Biol Chem. 1994;269:23757-23763. [PubMed] |
| 31. | Lindon JC, Holmes E, Nicholson JK. Metabonomics and its role in drug development and disease diagnosis. Expert Rev Mol Diagn. 2004;4:189-199. [PubMed] [DOI] |
| 32. | Goodacre R, Vaidyanathan S, Dunn WB, Harrigan GG, Kell DB. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 2004;22:245-252. [PubMed] [DOI] |
| 33. | Brenton JD, Carey LA, Ahmed AA, Caldas C. Molecular classification and molecular forecasting of breast cancer: ready for clinical application? J Clin Oncol. 2005;23:7350-7360. [PubMed] [DOI] |
| 34. | Florian CL, Preece NE, Bhakoo KK, Williams SR, Noble M. Characteristic metabolic profiles revealed by 1H NMR spectroscopy for three types of human brain and nervous system tumours. NMR Biomed. 1995;8:253-264. [PubMed] [DOI] |
| 35. | Florian CL, Preece NE, Bhakoo KK, Williams SR, Noble MD. Cell type-specific fingerprinting of meningioma and meningeal cells by proton nuclear magnetic resonance spectroscopy. Cancer Res. 1995;55:420-427. [PubMed] |
| 36. | Ben-Yoseph O, Badar-Goffer RS, Morris PG, Bachelard HS. Glycerol 3-phosphate and lactate as indicators of the cerebral cytoplasmic redox state in severe and mild hypoxia respectively: a 13C- and 31P-n.m.r. study. Biochem J. 1993;291:915-919. [PubMed] |
| 37. | Hakumäki JM, Poptani H, Puumalainen AM, Loimas S, Paljärvi LA, Ylä-Herttuala S, Kauppinen RA. Quantitative 1H nuclear magnetic resonance diffusion spectroscopy of BT4C rat glioma during thymidine kinase-mediated gene therapy in vivo: identification of apoptotic response. Cancer Res. 1998;58:3791-3799. [PubMed] |
| 38. | Tate AR, Crabb S, Griffiths JR, Howells SL, Mazucco RA, Rodrigues LM, Watson D. Lipid metabolite peaks in pattern recognition analysis of tumour in vivo MR spectra. Anticancer Res. 1996;16:1575-1579. [PubMed] |
| 39. | Millis K, Weybright P, Campbell N, Fletcher JA, Fletcher CD, Cory DG, Singer S. Classification of human liposarcoma and lipoma using ex vivo proton NMR spectroscopy. Magn Reson Med. 1999;41:257-267. [PubMed] [DOI] |
| 40. | Mountford CE, Wright LC. Organization of lipids in the plasma membranes of malignant and stimulated cells: a new model. Trends Biochem Sci. 1988;13:172-177. [PubMed] [DOI] |
| 41. | Callies R, Sri-Pathmanathan RM, Ferguson DY, Brindle KM. The appearance of neutral lipid signals in the 1H NMR spectra of a myeloma cell line correlates with the induced formation of cytoplasmic lipid droplets. Magn Reson Med. 1993;29:546-550. [PubMed] [DOI] |
| 42. | Cheng LL, Lean CL, Bogdanova A, Wright SC Jr, Ackerman JL, Brady TJ, Garrido L. Enhanced resolution of proton NMR spectra of malignant lymph nodes using magic-angle spinning. Magn Reson Med. 1996;36:653-658. [PubMed] [DOI] |
| 43. | Chen JH, Enloe BM, Fletcher CD, Cory DG, Singer S. Biochemical analysis using high-resolution magic angle spinning NMR spectroscopy distinguishes lipoma-like well-differentiated liposarcoma from normal fat. J Am Chem Soc. 2001;123:9200-9201. [PubMed] [DOI] |
| 44. | Millis K, Weybright P, Campbell N, Fletcher JA, Fletcher CD, Cory DG, Singer S. Classification of human liposarcoma and lipoma using ex vivo proton NMR spectroscopy. Magn Reson Med. 1999;41:257-267. [PubMed] [DOI] |
| 45. | Tomlins AM, Foxall PJD, Lindon JC, Lynch MJ, Spraul M, Everett JR, Nicholson JK. High resolution magic angle spinning 1H nuclear magnetic resonance analysis of intact prostatic hyperplastic and tumour tissues. Anal Comm. 1998;35:113-115. [DOI] |
| 46. | Griffiths JR, Stubbs M. Opportunities for studying cancer by metabolomics: preliminary observations on tumors deficient in hypoxia-inducible factor 1. Adv Enzyme Regul. 2003;43:67-76. [PubMed] [DOI] |
| 47. | Preul MC, Caramanos Z, Collins DL, Villemure JG, Leblanc R, Olivier A, Pokrupa R, Arnold DL. Accurate, noninvasive diagnosis of human brain tumors by using proton magnetic resonance spectroscopy. Nat Med. 1996;2:323-325. [PubMed] [DOI] |
| 48. | Howells SL, Maxwell RJ, Peet AC, Griffiths JR. An investigation of tumor 1H nuclear magnetic resonance spectra by the application of chemometric techniques. Magn Reson Med. 1992;28:214-236. [PubMed] [DOI] |
| 49. | Carmichael PL. Mechanisms of action of antiestrogens: relevance to clinical benefits and risks. Cancer Invest. 1998;16:604-611. [PubMed] [DOI] |
| 50. | Beckonert O, Monnerjahn J, Bonk U, Leibfritz D. Visualizing metabolic changes in breast-cancer tissue using 1H-NMR spectroscopy and self-organizing maps. NMR Biomed. 2003;16:1-11. [PubMed] [DOI] |
| 51. | Lehtimäki KK, Valonen PK, Griffin JL, Väisänen TH, Gröhn OH, Kettunen MI, Vepsäläinen J, Ylä-Herttuala S, Nicholson J, Kauppinen RA. Metabolite changes in BT4C rat gliomas undergoing ganciclovir-thymidine kinase gene therapy-induced programmed cell death as studied by 1H NMR spectroscopy in vivo, ex vivo, and in vitro. J Biol Chem. 2003;278:45915-45923. [PubMed] [DOI] |
| 52. | Williams SN, Anthony ML, Brindle KM. Induction of apoptosis in two mammalian cell lines results in increased levels of fructose-1,6-bisphosphate and CDP-choline as determined by 31P MRS. Magn Reson Med. 1998;40:411-420. [PubMed] [DOI] |
| 53. | Anthony ML, Zhao M, Brindle KM. Inhibition of phosphatidylcholine biosynthesis following induction of apoptosis in HL-60 cells. J Biol Chem. 1999;274:19686-19692. [PubMed] [DOI] |
| 54. | Griffin JL, Lehtimäki KK, Valonen PK, Gröhn OH, Kettunen MI, Ylä-Herttuala S, Pitkänen A, Nicholson JK, Kauppinen RA. Assignment of 1H nuclear magnetic resonance visible polyunsaturated fatty acids in BT4C gliomas undergoing ganciclovir-thymidine kinase gene therapy-induced programmed cell death. Cancer Res. 2003;63:3195-3201. [PubMed] |
| 55. | Singer S, Millis K, Souza K, Fletcher C. Correlation of lipid content and composition with liposarcoma histology and grade. Ann Surg Oncol. 1997;4:557-563. [PubMed] [DOI] |
| 56. | El-Sayed S, Bezabeh T, Odlum O, Patel R, Ahing S, MacDonald K, Somorjai RL, Smith IC. An ex vivo study exploring the diagnostic potential of 1H magnetic resonance spectroscopy in squamous cell carcinoma of the head and neck region. Head Neck. 2002;24:766-772. [PubMed] [DOI] |
| 57. | Moreno A, López LA, Fabra A, Arús C. 1H MRS markers of tumour growth in intrasplenic tumours and liver metastasis induced by injection of HT-29 cells in nude mice spleen. NMR Biomed. 1998;11:93-106. [PubMed] [DOI] |
| 58. | Odunsi K, Wollman RM, Ambrosone CB, Hutson A, McCann SE, Tammela J, Geisler JP, Miller G, Sellers T, Cliby W. Detection of epithelial ovarian cancer using 1H-NMR-based metabonomics. Int J Cancer. 2005;113:782-788. [PubMed] [DOI] |
| 59. | Yang Y, Li C, Nie X, Feng X, Chen W, Yue Y, Tang H, Deng F. Metabonomic studies of human hepatocellular carcinoma using high-resolution magic-angle spinning 1H NMR spectroscopy in conjunction with multivariate data analysis. J Proteome Res. 2007;6:2605-2614. [PubMed] [DOI] |
| 60. | Fang F, He X, Deng H, Chen Q, Lu J, Spraul M, Yu Y. Discrimination of metabolic profiles of pancreatic cancer from chronic pancreatitis by high-resolution magic angle spinning 1H nuclear magnetic resonance and principal components analysis. Cancer Sci. 2007;98:1678-1682. [PubMed] [DOI] |
| 61. | Xu G, Liebich HM, Lehmann R, Müller-Hagedorn S. Capillary electrophoresis of urinary normal and modified nucleosides of cancer patients. Methods Mol Biol. 2001;162:459-474. [PubMed] |
| 62. | Kim YS, Maruvada P, Milner JA. Metabolomics in biomarker discovery: future uses for cancer prevention. Future Oncol. 2008;4:93-102. [PubMed] [DOI] |
| 63. | Sasano H, Suzuki T, Nakata T, Moriya T. New development in intracrinology of breast carcinoma. Breast Cancer. 2006;13:129-136. [PubMed] [DOI] |
| 64. | Ippolito JE, Merritt ME, Bäckhed F, Moulder KL, Mennerick S, Manchester JK, Gammon ST, Piwnica-Worms D, Gordon JI. Linkage between cellular communications, energy utilization, and proliferation in metastatic neuroendocrine cancers. Proc Natl Acad Sci USA. 2006;103:12505-12510. [PubMed] [DOI] |
| 65. | Ichikawa W. Prediction of clinical outcome of fluoropyrimidine-based chemotherapy for gastric cancer patients, in terms of the 5-fluorouracil metabolic pathway. Gastric Cancer. 2006;9:145-155. [PubMed] [DOI] |