修回日期: 2008-07-28
接受日期: 2008-08-04
在线出版日期: 2008-08-28
目的: 证实乙型肝炎病毒(hepatitis B virus, HBV)X基因一种新的变异方式.
方法: 从HBV慢性感染患者血清中提取HBV DNA, 扩增X基因序列, 克隆入pMD19 T载体, 选择阳性克隆进行DNA测序, 与已知HBV基因相应序列比较该患者体内HBV基因变异位点以及变异形式.
结果: 从21例患者中共挑选74个克隆测序, 测序结果提示54个克隆X基因下游大段缺失突变, 长度达234 nt, 位于1601-1834 nt处, 另有1个克隆发生245 nt缺失突变. 发生缺失变异的病毒株同时存在G/A1515C、G1518C和A1585T替换突变, 这两种突变具有联动特征. 缺失突变株HBx仅编码76 aa, 其第44和45位编码为LL, 具有特异性.
结论: 观察到一种X蛋白变异方式, 这种大段缺失突变导致X蛋白下游编码序列丢失, 其为X因子还是X蛋白以及这种变异是否为常态形式尚需进一步研究.
引文著录: 肖鸿敏, 任建林, 毛乾国, 许鸿志, 陈美娅, 张志平, 周飞, 潘金水, 蔡稼燕, 董菁. 一种新的乙型肝炎病毒X基因变异方式. 世界华人消化杂志 2008; 16(24): 2695-2701
Revised: July 28, 2008
Accepted: August 4, 2008
Published online: August 28, 2008
AIM: To identify a novel X gene mutation pattern of hepatitis B virus (HBV) in patients with chronic HBV infection.
METHODS: A pair of primers was designed on the basis of nucleotide sequences of X gene. Polymerase chain reaction (PCR) was used to amplify the target region from HBV DNA samples extracted from chronic hepatitis B patients in Xiamen city. After electrophoresis of the PCR products in 9 g/L agarose gel, the target regions were cut, re-purified and TA-cloned into pMD19 T vector. The inserted regions in positive clones were sequenced. Sequence comparison with HBV genome submitted in GenBank was made to find the mutation sites.
RESULTS: Totally 74 strains from 21 patients with chronic HBV infection were sequenced, and the results showed that there was a characteristic deletion region, with a length of 234 nt (nt 1601-1834) in 54 clones, and a length of 245 nt in 1 clone. There were 3 replacement mutations bounding to deletion mutation: G/A1515C, G1518C and A1585T, which caused substitutions in the 44th and 45th amino acid site to LL. These mutant strains only coded 76 aa of up-stream HBx.
CONCLUSION: A novel deletion mutation in HBV X gene is observed in patients with chronic HBV infection. The deletion mutants encode 76-aa X factor instead of X protein.
- Citation: Xiao HM, Ren JL, Mao QG, Xu HZ, Chen MY, Zhang ZP, Zhou F, Pan JS, Cai JY, Dong J. A new mutation pattern of hepatitis B virus X gene. Shijie Huaren Xiaohua Zazhi 2008; 16(24): 2695-2701
- URL: https://www.wjgnet.com/1009-3079/full/v16/i24/2695.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i24.2695
乙型肝炎病毒(HBV)基因组含有4个开放读码框架(ORF), 以前的研究表明前C/C区和P区为突变高发区, X区相对较为保守. X基因长度465 nt, 位于1372-1836 nt处[1](V01460), 编码154 aa. 1990年Loncarevic et al[2]克隆了一株adr血清型的HBV基因组, 发现了前X区, 长度168 nt, 编码56 aa. Takahashi et al[3]于1995年的初步研究认为前X区编码与肝细胞癌(HCC)的形成有关. 我们早期[4-6]的研究证实前X区的存在并非个体现象, 因此提出X基因可能具有2种形式: 编码154 aa的X基因和编码210 aa的全X基因.
最近我们[7]观察到一例患者HBeAg/抗-HBe血清学转换过程中, HBV X基因中下游发生缺失突变, 长度达234 nt, 缺失突变后的X基因无前X区, 仅编码76 aa, 提示X基因可能存在第三种形式. 为了解这种变异的普遍性, 我们进一步检测了21例HBV感染者X基因的变异情况.
患者诊断符合2000年西安会议《病毒性肝炎防治方案》(试行)标准. 采集患者静脉血, 蛋白酶K消化-饱和酚∶氯仿(1∶1)抽提法提取200 μL血清中的HBV DNA, -20℃保存备用.
多聚酶链反应(PCR)扩增目的片段: 参考我们[4]以往克隆的中国大陆地区HBV基因组相关区域序列设计引物, 上游引物为: 5'- CAAGTGTTTGCTGACGCAACC-3', 1176-1198 nt; 下游引物为: 5'-ACAGCTTGGCGGCTTG AACAG-3', 1859-1880 nt, 目的片段长度约704 bp, 靶区域为前X区至X基因下游. PCR参数如下: 94℃ 1 min预变性, 94℃ 40 s变性, 58℃ 40 s退火, 72℃ 40 s延长, 共35个循环, 72℃再延长10 min. 将PCR产物在9 g/L琼脂糖(Promega公司)凝胶中电泳, 切取目的片段, 玻璃奶法(博大泰克公司)回收PCR产物, 与pMD19 T载体(TaKaRa公司)连接过夜. 将重组质粒转入TOP 10菌株(本室保存), 氨苄青霉素和X-gal蓝白斑法筛选阳性菌落. 考虑到HBV的准种[8-9]特性, 自同一患者阳性克隆中选择至少2个以上进行DNA测序, 由上海英骏生物公司测序, 测序结果结果存入美国国立卫生院GenBank中. 应用Vector NTI 8.0软件将测序所得核苷酸序列结果进行比较, 选择的对照为Galibert et al[1]存于GenBank中的HBV全基因组序列: V01460(2003年修正版), 全文编码位置是以该序列为对照.
21例患者, 男性15例, 女性6例, 年龄跨度为16-46岁, 发现HBV感染时间跨度自1-15年不等. 19例临床诊断为慢性乙型肝炎(CHB), 2例诊断为乙肝肝硬化, 临床情况见表1.
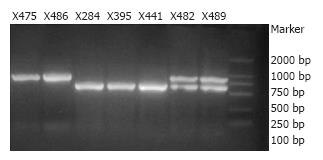
预计PCR产物长度为704 bp, 将产物电泳后发现PCR产物长度存在两种形式, 约700 bp和约500 bp两种. 不同患者来源病毒PCR产物电泳后展示为3种形式: 单独700 bp(3例)、单独500 bp条带(7例)和两条不同长度条带同时存在(11例, 图1). 长度约500 bp的PCR产物提示在靶基因内部具有缺失突变, 其范围大约200 bp左右.
经PCR法获得的靶基因经回收后连接入pMD 19 T载体, 21例患者共克隆出421个克隆. 考虑到HBV是以准种形式存在, 每例患者的克隆群中至少选择2个阳性克隆进行测序, 共选择74个克隆进行DNA测序. 测序结果存储于GenBank中: EU043336-EU043352.
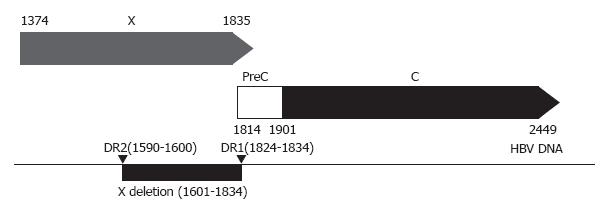
经过比较, 本研究观察到的缺失突变株中有54株表现为nt 1601-1834处发生大段缺失突变, 长度234 nt(图2), 涵盖了X基因1372-1836 nt处的中下游部分, 1株表现为245 nt长度的缺失突变, 查询GenBank及既往文献未见类似报道. 6例患者的19个克隆未检测出X基因大段缺失突变, 其中X475和X486的PCR产物表现为单一的700 bp条带(图1), 测序结果未发现有大段缺失突变, 其他4例患者的测序结果提示为两种方式共存. 将本研究发现的X基因缺失突变的涵盖区域简化示意图(图3).
检测出的X基因缺失株编码X蛋白发生截短型变异, 其编码长度仅为76 aa(图4). 由于绝大多数蛋白质其最低长度为100 aa, 因此我们将这种突变株编码产物暂且定义为X因子. 74株测序的病毒株中, 来自6例患者的19株序列编码至少154 aa的全长X蛋白, 有1株缺失突变长度达245 nt的病毒株无法编码X蛋白/X因子. 54株X基因下游缺失突变234 nt的病毒株编码形式有9种(图4), 其中XX395-3编码的X因子序列为主流编码形式, 代表了18例患者的41株克隆编码形式.
X因子与X蛋白之间重要的变异是第44和第45位aa, X蛋白在这两个位置编码VV/IV, 而X因子编码为LL(仅1株编码PL), 这种点变异与G/A1515C、G1518C点突变相对应.
1979年Galibert et al[1]首次解密HBV基因组全序列并定位了4个开放读码框(open reading frame, ORF), 分别为C、P、S、X区, 目前多数学者对4个ORF分区的界定并无异议. 以往研究多认为HBV X基因相对较为保守, 内部突变的发生率较低, 以往研究曾报道一些点替换突变, 如94[10], 31[11], 38[12] aa, 这些突变可能影响HBx功能. 1990年Loncarevic et al[2]首先报告了前X区的存在, Takahashi et al[3,13] 研究认为前X区存在与否与原发性肝细胞癌(hepatic cell carcinoma, HCC)关系密切, 我们早期的研究[4,6]认为前X区的存在可能存在一定的普遍性. 由此提出一个问题: HBx是否存在结构上的多态性?
最初我们[7]在1例HBeAg转换期间观察到其体内HBV的20株克隆(EF608587-EF608597)表现为nt 1601-1834处发生大段缺失突变, 长度达234 nt, 不仅导致核心蛋白启动子功能缺失, 包括Enh Ⅱ和DRⅠ缺失, 同时也导致HBX下游缺失突变, 变异株克隆的X截短型基因仅编码前76 aa. 为了验证该变异的普遍性, 本研究对21例HBV感染者血清中病毒株X基因进行DNA测序, 研究发现: 19例患者共55株克隆表现为X基因中下游缺失突变, 除1株外, 缺失突变位置与早期发现完全一致, 因此本文暂且把nt 1601-1834处发生的缺失突变定义为XD. 本研究还发现与XD同时发生的3个点替换突变, 即G/A1515C、G1518C和A1585T突变, 这3个位置的替换突变与缺失突变同时存在, 而这3个位置未发生替换突变的病毒株其下游均为发生缺失突变. 这种联动突变的内在分子机制并不明了, 其变异相关关系还需要进一步研究. 将我们测序结果在GenBank中搜索以及对相关文献查询, 未发现相关报道. 早年我们[14]的研究提示X基因下游有短的缺失突变, 长度为8或20 nt两种类型; Zhu et al[15]发表的文章也发现有短的缺失突变(19 nt)存在, 但跨度长达234 nt并造成HBX结构改变的缺失突变尚未见报道. 这个分子流行病学调查结果提示XD不是一个偶然的特有现象, 很可能是一种新的变异方式, 或者至少是具有地域特异性的一种变异方式.
本文观察厦门地区HBV感染者19例患者血清中HBV病毒株X基因中下游存在缺失突变, XD基因编码一个长度仅76 aa的多肽, 其中来自18例患者的41个克隆编码同一个氨基酸序列. 一般而言蛋白质最低长度不小于100 aa, 我们姑且将发现的长度为76 aa的多肽称之为X因子. 近期学者[16-17]研究HBx的结构, 把其结构分为2部分, 1-51 aa的负性调节功能域, 52-148 aa的反式激活功能域, X因子不含有反式激活作用, 可能的调节作用与HBx大相径庭. 除此之外, 我们发现的自然状态下存在的XD变异株编码的X因子还可能导致原HBx细胞内定位[18]、信号转导[19]或与宿主蛋白结合[20]等功能丢失或障碍.
在X因子与HBX之间存在一特征性替换突变位点, 即第44和第45位aa, HBx在这两个位置编码VV/IV, 除1株编码PL, 其他53株X因子编码序列为LL, 这2个氨基酸替换突变是由G/A1515C和G1518C核苷酸替换突变所造成的. 点替换突变与缺失突变具有联动性特征, 即便来自不同的患者, X因子编码区这3个位置的变异是相同的, 提示变异之间具有内部分子机制. 新发现的XD变异株提示HBV X基因可能有3种形式: 经典(typical)X基因, 前X/经典X基因, XD编码序列. 本研究观察到的病毒株均不编码前X区. XD特有的替换突变联合缺失突变产生一种调节因子, 这种因子是否与人类免疫缺陷病毒(HIV)的小分子调节因子(Vpr、Vpu)[21-22]类似? 抑或为一种病毒返祖现象? 禽类乙型肝炎病毒, 如鸭乙型肝炎病毒[23]、雪鹅乙型肝炎病毒[24]并不编码X基因, 而旱獭乙型肝炎病毒[25]编码X基因, 随着病毒适应哺乳动物, X基因成为一种必需, 而我们观察到的现象也许是一种病毒返祖现象, 这种功能学推断还需要试验的证实.
总之, 为了解X基因的多态性, 我们通过技术可靠的PCR-TA克隆-DNA测序方法探寻厦门本地HBV感染者体内病毒株X基因的差异方式, 结果发现19例患者体内病毒株X基因下游存在缺失突变, 长度为234 nt, 该突变与G/A1515C、G1518C和A1585T替换突变同时存在, 突变株编码一种长度为76 aa的多肽, 我们初步命名其为X因子. 本研究PCR靶产物的长度为704 bp, 目前应用的PCR Taq酶的精度为1/104-5 bp, 已被广泛用于单核苷酸多态性[26]研究, 方法可靠, 结果可信. 因此本研究报告的可能是一种尚未被广泛研究的突变形式, 但仍需要更大样本的调查验证.
HBV X蛋白的编码区相对保守, 以往主要报道是一长度465 bp的固定区域, 有学者提出存在前-X区, 本研究组早期研究曾发现一患者自发出现HBeAg血清学转换时, X基因内部出现长度234 nt的缺失突变.
樊红, 副教授, 东南大学医学院发育与疾病相关基因教育部重点实验室.
既往研究提示X基因下游可有8、19、20 nt的缺失突变, 未曾报道过长度为234 nt的缺失突变, 这种突变将完全改变HBx的功能.
本文以技术成熟可靠的PCR-TA克隆-DNA测序的方法探讨了厦门地区部分患者体内HBV X基因的变异情况, 发现无论HBeAg阳性或者阴性的患者都存在XD变异株, 该变异株编码X因子, 长度仅为76 aa.
通过本研究组的分子生物学调查, 厦门地区CHB患者血清中X基因存在长度达234 nt的缺失突变, 且位置相同, 初步提示这种变异形式并非个体孤立现象. 此外, 有3个位置的点替换突变与XD同时发生, 提示内部可能存在一定的变异机制, 值得进一步研究.
XD: X基因缺失突变, 特指本研究展示的位置于nt 1601-1834的缺失突变. X因子, XD编码的长度为76 aa的截短型HBx.
本研究方法得当, 材料充分, 结果较可靠, 具有较好的研究意义.
编辑: 李军亮 电编: 吴鹏朕
| 1. | Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature. 1979;281:646-650. [PubMed] [DOI] |
| 2. | Loncarevic IF, Zentgraf H, Schröder CH. Sequence of a replication competent hepatitis B virus genome with a preX open reading frame. Nucleic Acids Res. 1990;18:4940. [PubMed] [DOI] |
| 3. | Takahashi K, Kishimoto S, Ohori K, Yoshizawa H, Akahane Y, Okamoto H, Mishiro S. A unique set of mutations in the "preX" region of hepatitis B virus DNA frequently found in patients but not in asymptomatic carriers: implication for a novel variant. Int Hepatol Commun. 1995;3:131-138. [DOI] |
| 8. | Ngui SL, Teo CG. Hepatitis B virus genomic heterogeneity: variation between quasispecies may confound molecular epidemiological analyses of transmission incidents. J Viral Hepat. 1997;4:309-315. [PubMed] [DOI] |
| 9. | Cane PA, Mutimer D, Ratcliffe D, Cook P, Beards G, Elias E, Pillay D. Analysis of hepatitis B virus quasispecies changes during emergence and reversion of lamivudine resistance in liver transplantation. Antivir Ther. 1999;4:7-14. [PubMed] |
| 10. | Stemler M, Weimer T, Tu ZX, Wan DF, Levrero M, Jung C, Pape GR, Will H. Mapping of B-cell epitopes of the human hepatitis B virus X protein. J Virol. 1990;64:2802-2809. [PubMed] |
| 11. | Yeh CT, Shen CH, Tai DI, Chu CM, Liaw YF. Identification and characterization of a prevalent hepatitis B virus X protein mutant in Taiwanese patients with hepatocellular carcinoma. Oncogene. 2000;19:5213-5220. [PubMed] [DOI] |
| 12. | Muroyama R, Kato N, Yoshida H, Otsuka M, Moriyama M, Wang Y, Shao RX, Dharel N, Tanaka Y, Ohta M. Nucleotide change of codon 38 in the X gene of hepatitis B virus genotype C is associated with an increased risk of hepatocellular carcinoma. J Hepatol. 2006;45:805-812. [PubMed] [DOI] |
| 13. | Takahashi K, Akahane Y, Hino K, Ohta Y, Mishiro S. Hepatitis B virus genomic sequence in the circulation of hepatocellular carcinoma patients: comparative analysis of 40 full-length isolates. Arch Virol. 1998;143:2313-2326. [PubMed] [DOI] |
| 15. | Zhu P, Tan D, Peng Z, Liu F, Song L. Polymorphism analyses of hepatitis B virus X gene in hepatocellular carcinoma patients from southern China. Acta Biochim Biophys Sin (Shanghai). 2007;39:265-272. [PubMed] [DOI] |
| 16. | Tang H, Delgermaa L, Huang F, Oishi N, Liu L, He F, Zhao L, Murakami S. The transcriptional transactivation function of HBx protein is important for its augmentation role in hepatitis B virus replication. J Virol. 2005;79:5548-5556. [PubMed] [DOI] |
| 17. | Tang H, Oishi N, Kaneko S, Murakami S. Molecular functions and biological roles of hepatitis B virus x protein. Cancer Sci. 2006;97:977-983. [PubMed] [DOI] |
| 18. | Henkler F, Hoare J, Waseem N, Goldin RD, McGarvey MJ, Koshy R, King IA. Intracellular localization of the hepatitis B virus HBx protein. J Gen Virol. 2001;82:871-882. |
| 19. | Bouchard MJ, Wang L, Schneider RJ. Activation of focal adhesion kinase by hepatitis B virus HBx protein: multiple functions in viral replication. J Virol. 2006;80:4406-4414. [PubMed] [DOI] |
| 20. | Marusawa H, Matsuzawa S, Welsh K, Zou H, Armstrong R, Tamm I, Reed JC. HBXIP functions as a cofactor of survivin in apoptosis suppression. EMBO J. 2003;22:2729-2740. [PubMed] [DOI] |
| 21. | Huard S, Elder RT, Liang D, Li G, Zhao RY. Human immunodeficiency virus type 1 Vpr induces cell cycle G2 arrest through Srk1/MK2-mediated phosphorylation of Cdc25. J Virol. 2008;82:2904-2917. [PubMed] [DOI] |
| 22. | Hussain A, Wesley C, Khalid M, Chaudhry A, Jameel S. Human immunodeficiency virus type 1 Vpu protein interacts with CD74 and modulates major histocompatibility complex class II presentation. J Virol. 2008;82:893-902. [PubMed] [DOI] |
| 23. | Guo H, Mason WS, Aldrich CE, Saputelli JR, Miller DS, Jilbert AR, Newbold JE. Identification and characterization of avihepadnaviruses isolated from exotic anseriformes maintained in captivity. J Virol. 2005;79:2729-2742. [PubMed] [DOI] |
| 24. | Chang SF, Netter HJ, Bruns M, Schneider R, Frölich K, Will H. A new avian hepadnavirus infecting snow geese (Anser caerulescens) produces a significant fraction of virions containing single-stranded DNA. Virology. 1999;262:39-54. [PubMed] [DOI] |
| 25. | Girones R, Cote PJ, Hornbuckle WE, Tennant BC, Gerin JL, Purcell RH, Miller RH. Complete nucleotide sequence of a molecular clone of woodchuck hepatitis virus that is infectious in the natural host. Proc Natl Acad Sci USA. 1989;86:1846-1849. [PubMed] [DOI] |
| 26. | Huard RC, Fabre M, de Haas P, Lazzarini LC, van Soolingen D, Cousins D, Ho JL. Novel genetic polymorphisms that further delineate the phylogeny of the Mycobacterium tuberculosis complex. J Bacteriol. 2006;188:4271-4287. [PubMed] [DOI] |