修回日期: 2008-06-16
接受日期: 2008-06-17
在线出版日期: 2008-07-28
目的: 分离并鉴定整合表达HBV的人肝癌细胞系HepG2.2.15及其母细胞系HepG2的差异HLAⅠ类分子结合肽, 寻找与HBV感染相关的HLA结合肽.
方法: 洗涤法得到HepG2和HepG2.2.15细胞表面的HLAⅠ类分子及与其结合的肽段. 效液相色谱(HPLC)对两种细胞的洗脱肽段进行分离和图谱比较, 挑选出HepG2.2.15特有的峰段进行纳升电喷雾串联质谱(nanoESI-MS/MS)分析, 结合MASCOT数据库检索和从头测序, 分析多肽的序列和来源. 后采用RT-PCR法进行mRNA表达检测.
结果: HPLC对比显示HepG2和HepG2.2.15的HLA结合肽存在显著差异. 利用nanoESI-MS/MS技术从HPLC差异峰段中分离鉴定出一条来源于已知蛋白人烯醇酶-1(enolase1, ENO1)的多肽SPDDPSRYISPDQ. RT-PCR结果显示, ENO1在HepG2和HepG2.2.15细胞中均有表达, 且在HepG2.2.15细胞中表达明显高于HepG2细胞.
结论: 来自ENO1的多肽SPDDPSRYISPDQ能够被HLAⅠ类分子提呈到HepG2.2.15细胞表面, 且ENO1 mRNA在HepG2.2.15细胞中的表达显著高于HepG2细胞. 提示HBV感染可能引起ENO1表达上调.
引文著录: 陈黎, 谢兴旺, 张恒辉, 费然, 丛旭, 魏来, 陈红松. 人肝癌细胞系HepG2与HepG2.2.15差异HLA结合肽的分离与鉴定. 世界华人消化杂志 2008; 16(21): 2343-2348
Revised: June 16, 2008
Accepted: June 17, 2008
Published online: July 28, 2008
AIM: To screen the differential HLA-binding peptides between HepG2 and HepG2.2.15 cell lines, and to find some HLA-binding peptides correlated with hepatitis B virus (HBV) infection.
METHODS: HepG2 and HepG2.2.15 cells were harvested (108 cells), and the peptides were isolated from the cell membrane by mild acid elution, respectively. Then the mixture of peptides was fractionated by high performance liquid chromatography (HPLC) and the differential fractions only expressed in HepG2.2.15 cell line were identified by nanoESI-MS/MS analysis. Bioinformatic analysis and MASCOT index were used to investigate the sequence and source of the peptides. Finally the expression of mRNA was detected by reverse transcription-polymerase chain reaction (RT-PCR).
RESULTS: HPLC fractions were markedly different between HepG2 and HepG2.2.15 cells. A peptide, SPDDPSRYISPDQ, from enolase 1 (ENO1) was obtained, which was only expressed in HepG2.2.15 cells, by nanoESI-MS/MS analysis. The result of RT-PCR confirmed that ENO1 expression was significantly higher in HepG2.2.15 cells than that in HepG2 cells.
CONCLUSION: A human peptide SPDDPSRYISPDQ from ENO1 can be presented on the surface of HepG2.2.15 cells by HLA, and ENO1 mRNA expression is significantly higher in HepG2.2.15 than that in HepG2, suggesting that HBV infection may cause the up-regualtion of ENO1 expression.
- Citation: Chen L, Xie XW, Zhang HH, Fei R, Cong X, Wei L, Chen HS. Identification of differential HLA-binding peptide between two hepatoma cell lines HepG2 and HepG2.2.15. Shijie Huaren Xiaohua Zazhi 2008; 16(21): 2343-2348
- URL: https://www.wjgnet.com/1009-3079/full/v16/i21/2343.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i21.2343
乙型肝炎病毒(hepatitis B virus, HBV)的感染是一项严重的全球性公共卫生课题. 据WHO估计[1], 全球有20多亿人感染HBV, 而HBsAg携带者为3.5亿. 我国属HBV感染高流行区, 一般人群的HBsAg阳性率为9.09%[2]. HepG2.2.15细胞是由HepG2细胞衍生而来的稳定支持HBV复制、组装和分泌的细胞系, 是一个研究HBV生命活动的良好细胞系[3]. HepG2.2.15不仅表现HepG2的生物学特性, 同时又因为其携带有HBV基因组而表达不同于HepG2的特性, 比如他可以分泌HBeAg及HBsAg[4]. 所以我们可以通过对比这两种细胞表面HLA分子结合的抗原肽寻找差异所在, 从而筛选出HepG2.2.15细胞所递呈的HBV特异性天然表位或者HBV感染相关蛋白来源的HLA结合肽, 为乙肝的免疫发病机制研究及预防治疗提供新的思路.
细胞培养所用DMEM, 胎牛血清均购自Gibco公司; 色谱所用三氟乙酸、乙腈购自Sigma公司, C-18 Spe-Pak柱(经典型)购自Waters公司; 流式细胞所用抗体FITC标记的抗人的HLA-A2, 阴性对照为抗鼠的IgG, 均购自BD公司; 免疫组化所用抗体为ABCam公司的抗鼠的HLA-A2.
1.2.1 细胞培养: HepG2购自ATCC, HepG2.2.15为本所保存. HepG2和HepG2.2.15用含100 mL/L胎牛血清, 100 mg/L青霉素和100 mg/L链霉素的DMEM培养基, 并于HepG2.2.15培养液中加入400 mg/L的G418; 细胞均于37℃, 50 mL/L CO2孵箱培养.
1.2.2 弱酸法提取细胞表面结合肽: 每108细胞与1 mL的柠檬酸缓冲液(0.131 mol/L枸橼酸, 0.066 mol/L磷酸氢二钠, 调节pH至3.3), 在室温下作用5 min, 离心, 4℃, 2000 r/min, 5 min[5-6]. 收集上清, 即为多肽混合液. 为了验证后期实验的稳定性和可靠性, 我们将HepG2样品分成两份, 其中一份掺入一段来源于HCV的多肽(RLIVFPDLGV)做为参照.
1.2.3 弱酸洗脱细胞表面HLA结合肽效率的检测: 应用流式细胞术, 免疫荧光检测细胞表面HLA-A2的表达.
1.2.4 HPLC纯化: 从HepG2和HepG2.2.15细胞上提取的肽分别用反相HPLC系统(安捷伦, HP1100型)进行分馏. 样品注入C18柱(4.6 mm×150 mm, Waters), 进行梯度洗脱, B液为0%-100%乙腈, A液为1 g/L TFA, 梯度设置如下: 0%-0% B 10 min, 0%-60% B 60 min, 60%-100% B 10 min, 100%-100% B 10 min, 流速1 mL/min. 紫外检测214 nm, 280 nm. 收集两种细胞差异的峰段, 冻干, -20℃保存.
1.2.5 NanoESI-MS/MS分析: 挑选出HepG2.2.15样品中特有峰段的馏分和两组HepG2样品的差异峰, 将他们分别溶于10 μL 1 g/L的TFA中, 注入带有电喷雾离子源的纳升电喷雾串联质谱仪(nanoESI-MS/MS)中. 质谱仪的扫描范围500-15 000, 用于测定每个样品中肽段的分子量和质荷比. 为了对天然肽进行MS测序, 每个肽段的撞击诱导解离(CID)的质谱结果都按单电荷的离子形式记录.
1.2.6 HLA结合肽的鉴定: NanoESI-MS/MS的结果采用MASCOT数据库检索和从头测序两种方法进行. 我们主要通过MASCOT数据库检索的方法来确定多肽的来源和序列: 由Q-TOF质谱图得到的原始数据文件经Masslynix软件(Micromass公司)处理转换成.pkl文件, 然后提交到MASCOT(http://www.matrixscience.com)上进行检索, 检索数据库为NCBInr的人和病毒数据库. 搜库参数设置如下: 酶切选用无酶切; 可变修饰选择M-氧化和C-烷基化; 母离子误差值设为100×10-6, 碎片离子为0.1 Da, P<0.05; 肽段匹配的打分值超过显著性检验水准时初步判定该肽段的来源被鉴定. 另一方面, 质谱图经Micromass的专用软件MaxEnt3处理后, 借助Micromass的专用软件MasSeq直接推导出肽段序列, 同时对该质谱图进行手工读图确认, 判定原则为二级质谱中所有主要肽谱峰都与理论计算的片段离子相匹配. 从头测序的结果可以辅助已知肽段的鉴定, 还能够提供新的不能查找到来源的肽段的序列.
1.2.7 肽段HLA结合力的分析: 利用SYFPEITHI数据库中的算法对上述两个肽段的HLA结合力进行评分(http//http://www.syfpeithi.de/scripts/mhcserver.dll/home.htm). 在线预测的HLA位点限定在HepG2和HepG2.2.15已知的HLA分型: HLA-A2, HLA-A24, HLA-B51及HLA-B53[7].
1.2.8 RT-PCR验证: 用RT-PCR法验证我们鉴定的多肽来源蛋白ENO1在HepG2.2.15和HepG2中的表达. ENO1 RT-PCR的引物及退火温度见表1, 循环条件为94℃预变性5 min, 94℃ 15 s, 60℃ 30 s, 72℃ 45 s, 35个循环后接72℃延伸10 min[8].
| 基因 | 引物 | 退火温度(℃) | 产物片段大小(bp) |
| ENO1 | 5'-GCGTTCAATGTCATCAATGG-3' | 60 | 591 |
| 5'-GTTGACTTTGAGCAGGAGGC-3' | |||
| GAPDH | 5'-CCACATCGCTCAGACACCAT-3' | 60 | 114 |
| 5'-GGCAACAATATCCACTTTACCAGA-3 |
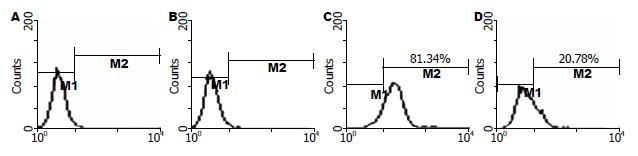
利用流式细胞技术比较弱酸洗脱前后细胞表面的HLA-A2表达量发现, 经过弱酸处理后细胞表面的HLA-A2标记显著减少, 由洗脱前的81.34%减少到洗脱后的20.78%(图1). 利用免疫荧光技术比较洗脱前后细胞表面HLA-A2的量也得到类似结果. 提示弱酸处理能有效洗脱细胞表面的HLAⅠ类分子及其结合的多肽.
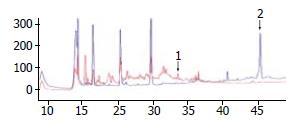
以反向HPLC纯化分离肽段, 比较掺入HCV肽段的HepG2和未掺入HCV肽段的HepG2 HPLC图, 以及HepG2.2.15和HepG2细胞的HPLC图, 挑选出HepG2样品差异的峰段和HepG2.2.15细胞特有的峰段(图2), 进行质谱分析.
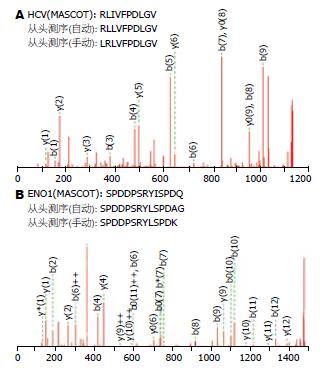
从掺入HCV肽段和未掺入HCV肽段的HepG2弱酸洗脱液差异色谱峰中, 鉴定出了实验掺入的HCV肽段, 其序列为RLIVFPDLGV. 同时从HepG2.2.15和HepG2的一个差异峰中, 鉴定出了1条人源的肽段, 即ENO1来源多肽: SPDDPSRYISPDQ(图3).
利用SYFPEITHI数据库, 按照既定的HLA分型对ENO1的多肽HLA结合力进行了在线预测. 发现该肽段与HLA-B51的结合力为22分. 肽段结合力的预测结果见表2.
| 多肽来源蛋白 | HCV(参照多肽) | 人烯醇酶-1 | |
| 实际序列 | RLIVFPDLGV | SPDDPSRYISPDQ | |
| 测序序列 | LRLVFPDLGV | SPDDPSRYLSPDK | |
| M/Z | 564.87 | 738.8 | |
| 理论分子量 | 1127.7254 | 1475.5854 | |
| 实测分子量 | 1127.6703 | 1475.6528 | |
| Accession number | gi/13936941 | gi/39644850 | |
| SYFPEITHI评分 | 未分析 | 22 | |
| HLA 分子 | HLA-A*02 | HLA-B*51 | |
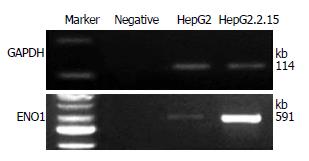
RT-PCR结果显示, ENO1在HepG2和HepG2.2.15细胞中均有表达, 且在HepG2.2.15细胞中表达明显高于HepG2细胞(图4).
内源性抗原是指细胞内产生的非己成分, 以一种不同于外源性抗原加工和递呈的方式进行加工和递呈, 而被加工的内源性抗原肽与HLAⅠ类分子结合形成复合体[9]. 淋巴细胞只能识别与HLA分子结合的限制性多肽(8-12个氨基酸), 即细胞表位(epitope)[10]. 弱酸洗脱法已经被证实可以有效的洗脱细胞表面HLAⅠ类分子及其结合的多肽[11-14]. 我们以HepG2和HepG2.2.15细胞为研究对象, 大量培养两种细胞至108, 用弱酸洗脱法获得浓度较高的多肽洗液, 其中主要包含HLAⅠ类分子及其结合多肽. 然后利用HPLC和nanoESI-MS/MS分析, 寻找在HepG2和HepG2.2.15细胞有差异表达的HLAⅠ类分子结合肽.
为了检测高效液相色谱联用串联质谱技术从多肽混合物中分离和鉴定多肽技术的稳定性和可靠性, 事先在HepG2的弱酸洗脱液中掺入了一段来自HCV聚合酶的参照肽段(RLIVFPDLGV). HPLC分离出了在掺入HepG2细胞弱酸洗脱液中的多肽并进行二级质谱分析. 用质谱分析的原始.pkl文件在MASCOT数据库中进行检索分析, 我们成功地找到事先掺入的参照肽段, 同时质谱程序自动分析得到的序列为LRLVFPDLGV, 与肽段实际序列基本匹配. 由此可见, HPLC联用nanoESI-MS/MS技术是一种有效的分离和鉴定多肽的方法. 但这种方法也有一定的局限性, 从上述的分析结果可以看出, nanoESI-MS/MS不能精确区分谷氨酰胺/赖氨酸(Gln/Lys)和异亮氨酸/亮氨酸(Ile/Leu), 而且在读序列过程中偶尔会有前后氨基酸置换的现象. 如果分离鉴定的是新的肽段, 从头测序的结果可能与实际的肽段序列有一定的差异.
应用同样的方法, 我们从HepG2和HepG2.2.15的一个较为明显的色谱差异峰中鉴定出了1条来源于人ENO1的多肽: SPDDPSRYIS PDQ. 既往的研究发现, ENO1的表达与包括HBV在内的多种病毒感染相关[15-17]. 最有意义的是, Yoon et al[18]利用表达谱芯片发现ENO1在HBV相关性肝癌中的表达显著上调, 并进一步证实了ENO1在4个HBV相关性细胞系中的表达显著高于正常组织(HBV相关性细胞系: SNU354, SNU368, SNU387和SNU475). 我们的实验结果证实ENO1在HepG2.2.15中表达明显高于HepG2细胞, 这同上述的相关报道均提示HBV感染可能会引起ENO1的表达上调, 并参与HBV导致的相关疾病的发生发展. 更为重要的是我们发现ENO1的SPDDPSRYISPDQ肽段能被HLA分子呈递于HepG2.2.15表面, 且肽段HLA结合力的分析表明该肽段与HLA-B51分子有较强的结合力, 提示他可能是被HLA-B51分子呈递的, 这与目前报道的HepG2.2.15细胞的HLA分型是吻合的. 现有的研究和进展均证实, HBV感染确实能够导致某些自身抗原出现, 而且这些自身抗原在乙肝的免疫发病过程中具有十分重要的作用[19]. 精氨酸酶[20]和230 kDa高尔基体相关蛋白(GCP230)[21]就被证实为HBV感染相关的自身抗原. 另外, 也已有研究发现ENO1可以在一些自身免疫性疾病中作为自身抗原引起免疫反应[22]. 所以, 我们推测来源于ENO1的多肽SPDDPSRYISPDQ很可能是与HBV感染引起的自身抗原表位, 需要进行更深入的研究来明确其在HBV免疫发病中的作用.
质谱技术已在蛋白组学研究中广泛使用[23-28], 但在乙肝表位鉴定方面尚处于探索阶段. 通常情况下, 一个细胞表面有105-106个MHCⅠ类分子, 呈递大约104个不同种类的肽[29], 不同种类肽的丰度相差极大, 而病理衍生肽往往是低丰度的, 在检测时会被高丰度的自身肽掩盖[30]. 因此对MHCⅠ类分子结合肽的研究, 需要先进的分离和检测技术. 利用nanoESI-MS/MS技术可以对微量而又复杂的多肽混合物进行较准确的分析. 本实验利用HPLC和nanoESI-MS/MS分离鉴定出1条HepG2.2.15表面的HLAⅠ类分子结合肽, 初步证实了HPLC联合nanoESI-MS/MS的方法在细胞HLA结合肽筛选过程中的可行性, 并展示了其快捷的优点.
本研究通过对HepG2和HepG2.2.15细胞株分析, 分离并鉴定出1条来自ENO1, 并能够被HLAⅠ类分子提呈到HepG2.2.15细胞表面的肽段SPDDPSRYISPDQ. 且ENO1 mRNA在HepG2.2.15细胞中的表达明显高于HepG2细胞. 提示HBV感染可能引起ENO1表达的上调, 并被呈递到细胞表面. 这为进一步研究乙肝病毒感染相关的蛋白功能和免疫学致病机制的研究提供了重要线索.
慢性乙型肝炎(CHB)是我国最常见的慢性传染性疾病之一, 易于慢性化、重症化, 已经成为严重威胁人类健康的公共卫生问题. 目前, 除干扰素和核苷类似物可使部分乙肝患者血清HBV DNA转阴外, 尚没有其他特效的治疗药物. 因此, 开发同时有治疗与预防作用的HBV疫苗具有重要的理论意义及潜在巨大的经济及社会价值.
林菊生, 教授, 华中科技大学同济医学院附属同济医院肝病研究所; 张小晋, 主任医师, 北京积水潭医院消化内科.
乙肝疫苗经历了传统的血源疫苗到重组蛋白疫苗, 到1990年代出现了DNA疫苗, 伴随着DNA疫苗的发展出现了多表位DNA疫苗. 蛋白质抗原不是通过完整的分子发挥作用的, 抗原分子表位的研究成了近年来新的发展趋势; 随着分子生物学及免疫学技术的日益成熟, 大量表位得以鉴定, 为特异性细胞免疫治疗奠定了基础. 目前正在研制更好的HBV疫苗, 如: DNA疫苗、含其他蛋白序列的HB疫苗、联合疫苗、单剂疫苗、口服疫苗等, 以增强疫苗的免疫原性, 简化乙肝疫苗接种.
Liu对105位中国乙肝患者核心抗原的研究发现, 93.3%缬氨酸突变位异亮氨酸(V27I). 突变后的多肽与HLA-A2分子有很高的结合力, 并且能在急性乙肝患者中引发特异性CTL反应, V27表位可以与I27表位特异性的CTL发生交叉反应, 反之则不然.
本研究通过HPLC和nanoESI-MS/MS分析, 直接在Hep G2.2.15细胞表面分离鉴定出与HLA结合的多肽, 并确定其来源分别是ENO1.
目前, 治疗性乙肝疫苗的研究集中在针对乙肝优势表位的多表位乙肝疫苗上, 本实验提供了快速有效的筛选乙肝HLA结合肽的方法, 为后期乙肝优势表位的筛选奠定了理论基础.
本文题目较明确, 研究方法较新颖, 书写规范, 可读性强, 对临床医生开拓眼界有一定的帮助.
编辑: 李军亮 电编:何基才
| 1. | World Health Organization. Hepatitis B. World Health Organization Fact Sheet 204 dex, 2000-10, cited: 2008-05; 1(1): 24 screens. Available from: http://www.who.int/mediacentre/factsheets/fs204/en/in html. |
| 2. | 梁 晓峰, 陈 园生, 王 晓军, 贺 雄, 陈 丽娟, 王 骏, 林 长缨, 白 呼群, 严 俊, 崔 钢. 中国3岁以上人群乙型肝炎 血清流行病学研究. 中华流行病学杂志. 2005;26:655-658. |
| 3. | Sells MA, Chen ML, Acs G. Production of hepatitis B virus particles in Hep G2 cells transfected with cloned hepatitis B virus DNA. Proc Natl Acad Sci U S A. 1987;84:1005-1009. [PubMed] [DOI] |
| 4. | Acs G, Sells MA, Purcell RH, Price P, Engle R, Shapiro M, Popper H. Hepatitis B virus produced by transfected Hep G2 cells causes hepatitis in chimpanzees. Proc Natl Acad Sci U S A. 1987;84:4641-4644. [PubMed] [DOI] |
| 5. | Sugawara S, Abo T, Kumagai K. A simple method to eliminate the antigenicity of surface class I MHC molecules from the membrane of viable cells by acid treatment at pH 3. J Immunol Methods. 1987;100:83-90. [PubMed] [DOI] |
| 6. | Storkus WJ, Zeh HJ 3rd, Salter RD, Lotze MT. Identification of T-cell epitopes: rapid isolation of class I-presented peptides from viable cells by mild acid elution. J Immunother Emphasis Tumor Immunol. 1993;14:94-103. [PubMed] |
| 7. | Datema G, Mulder A, Claas FH, van den Elsen PJ. Detection and characterization of HLA class I molecules in the supernatant of an hepatocarcinoma cell line and of EBV-transformed B cell lines. Hum Immunol. 1999;60:435-441. [PubMed] [DOI] |
| 8. | Ito S, Honma T, Ishida K, Wada N, Sasaoka S, Hosoda M, Nohno T. Differential expression of the human alpha-enolase gene in oral epithelium and squamous cell carcinoma. Cancer Sci. 2007;98:499-505. [PubMed] [DOI] |
| 9. | Lau JY, Bird GL, Naoumov NV, Williams R. Hepatic HLA antigen display in chronic hepatitis B virus infection. Relation to hepatic expression of HBV genome/gene products and liver histology. Dig Dis Sci. 1993;38:888-895. [PubMed] [DOI] |
| 10. | Mitchison NA. The carrier effect in the secondary response to hapten-protein conjugates. II. Cellular cooperation. Eur J Immunol. 1971;1:18-27. [PubMed] [DOI] |
| 11. | 周 迈, 彭 吉润, 王 红霞, 钟 朝辉, 郭 晏同, 潘 秀英, 冷 希圣. 利用质谱技术对肝癌组织中自然呈递的MAGE表位进行鉴定. 世界华人消化杂志. 2005;13:1395-1399. [DOI] |
| 13. | 朱 学军, 曹 雪涛, 雷 虹, 于 益芝, 王 建莉, 章 卫平, 马 施华. 弱酸洗脱提取的肿瘤抗原肽致敏的树突状细胞对CTL的体内外激活效应. 中华微生物学和免疫学杂志. 2000;20:98-101. |
| 15. | Niikura M, Liu HC, Dodgson JB, Cheng HH. A comprehensive screen for chicken proteins that interact with proteins unique to virulent strains of Marek's disease virus. Poult Sci. 2004;83:1117-1123. [PubMed] |
| 16. | Ghosh AK, Steele R, Ray RB. Functional domains of c-myc promoter binding protein 1 involved in transcriptional repression and cell growth regulation. Mol Cell Biol. 1999;19:2880-2886. [PubMed] |
| 17. | Leong WF, Chow VT. Transcriptomic and proteomic analyses of rhabdomyosarcoma cells reveal differential cellular gene expression in response to enterovirus 71 infection. Cell Microbiol. 2006;8:565-580. [PubMed] [DOI] |
| 18. | Yoon SY, Kim JM, Oh JH, Jeon YJ, Lee DS, Kim JH, Choi JY, Ahn BM, Kim S, Yoo HS. Gene expression profiling of human HBV- and/or HCV-associated hepatocellular carcinoma cells using expressed sequence tags. Int J Oncol. 2006;29:315-327. [PubMed] |
| 19. | Pelli N, Fensom AH, Slade C, Boa F, Mieli-Vergani G, Vergani D. Argininosuccinate lyase: a new autoantigen in liver disease. Clin Exp Immunol. 1998;114:455-461. [PubMed] [DOI] |
| 20. | Funaki T, Fujiwara T, Hong HS, Misumi Y, Nishioka M, Ikehara Y. Identification and characterization of a 230-kDa Golgi- associated protein recognized by autoantibodies from a patient with HBV hepatitis. Cell Struct Funct. 1996;21:63-72. [PubMed] |
| 21. | Chang GC, Liu KJ, Hsieh CL, Hu TS, Charoenfuprasert S, Liu HK, Luh KT, Hsu LH, Wu CW, Ting CC. Identification of alpha-enolase as an autoantigen in lung cancer: its overexpression is associated with clinical outcomes. Clin Cancer Res. 2006;12:5746-5754. [PubMed] [DOI] |
| 22. | Ditzel HJ, Masaki Y, Nielsen H, Farnaes L, Burton DR. Cloning and expression of a novel human antibody-antigen pair associated with Felty's syndrome. Proc Natl Acad Sci U S A. 2000;97:9234-9239. [PubMed] [DOI] |
| 23. | Chen P, Zhang L, Li X, Wang X, Cao R, Liu Z, Xiong J, Peng X, Wei Y, Ying X. Evaluation of strategy for analyzing mouse liver plasma membrane proteome. Sci China C Life Sci. 2007;50:731-738. [PubMed] [DOI] |
| 24. | Huang CM. Comparative proteomic analysis of human whole saliva. Arch Oral Biol. 2004;49:951-962. [PubMed] [DOI] |
| 25. | Liu J, Zhu P, Peng J, Li K, Du J, Gu J, Ou Y. Identification of disease-associated proteins by proteomic approach in ankylosing spondylitis. Biochem Biophys Res Commun. 2007;357:531-536. [PubMed] |
| 26. | Feng JT, Liu YK, Song HY, Dai Z, Qin LX, Almofti MR, Fang CY, Lu HJ, Yang PY, Tang ZY. Heat-shock protein 27: a potential biomarker for hepatocellular carcinoma identified by serum proteome analysis. Proteomics. 2005;5:4581-4588. [PubMed] [DOI] |
| 27. | Juan HF, Chen JH, Hsu WT, Huang SC, Chen ST, Yi-Chung Lin J, Chang YW, Chiang CY, Wen LL, Chan DC. Identification of tumor-associated plasma biomarkers using proteomic techniques: from mouse to human. Proteomics. 2004;4:2766-2775. [PubMed] [DOI] |
| 28. | Hansson L, Rabbani H, Fagerberg J, Osterborg A, Mellstedt H. T-cell epitopes within the complementarity-determining and framework regions of the tumor-derived immunoglobulin heavy chain in multiple myeloma. Blood. 2003;101:4930-4936. [PubMed] [DOI] |