修回日期: 2008-03-19
接受日期: 2008-05-06
在线出版日期: 2008-06-18
目的: 研究胰腺癌细胞基因组范围内的纯合性缺失和杂合性缺失(loss of heterozygosity, LOH).
方法: 应用高密度单核苷酸多态性芯片和专用分析软件, 检测17种胰腺癌细胞株基因组范围内的纯合性缺失和LOH, 并筛选可能与胰腺癌发生、发展有关的基因区域, 用PCR验证纯合性缺失.
结果: 经过PCR验证, 26个区域确实为纯合性缺失, 芯片的准确度为83.9%(26/31). 这些缺失区域中, 平均每个区域只涉及1.29个基因. 每一种细胞都有不同程度的LOH; 不同染色体臂出现LOH频率不同, 出现频率最高的为染色体臂9p和18q, 均为94.1%.
结论: 胰腺癌全基因组范围内出现多处LOH和纯合性缺失, 这些区域可能含有新抑癌基因.
引文著录: 林连捷, 王玉峰, 郑长青, 金玉, 胡刚正, 刘香, 林艳. 胰腺癌细胞基因组范围内的基因缺失. 世界华人消化杂志 2008; 16(17): 1849-1854
Revised: March 19, 2008
Accepted: May 6, 2008
Published online: June 18, 2008
AIM: To investigate genome-wide loss of heterozygosity (LOH) and homozygous deletion in human pancreatic cancer cell lines.
METHODS: Genome-wide LOH and homozygous deletion in 17 pancreatic cancer cell lines were studied using high-density single nucleotide polymorphism array and the data were analyzed using a special analytical software. PCR was performed to verify homozygous deletion following screening for potential genetic domains associated with development of pancreatic cancer.
RESULTS: A total of 26 homozygous deletions were verified by PCR and the accuracy of the chip was 83.9% (26/31). On average, 1.29 genes were involved in each region. Each pancreatic cancer cell line had different LOH. Different chromosome arms presented with various LOH frequency with the most common abnormalities in 9p and 18q, occurring in 16 cell lines (94.1%).
CONCLUSION: Genome-wide LOH and homozygous deletions are common in pancreatic cancer cell lines, indicating existence of novel tumor suppressor genes.
- Citation: Lin LJ, Wang YF, Zheng CQ, Jin Y, Hu GZ, Liu X, Lin Y. Genome-wide gene loss in human pancreatic cancer cells. Shijie Huaren Xiaohua Zazhi 2008; 16(17): 1849-1854
- URL: https://www.wjgnet.com/1009-3079/full/v16/i17/1849.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i17.1849
自20世纪50年代以来, 世界各国就展开了对肿瘤发生机制的探讨, 70年代末, 分子生物学尤其是分子遗传学的兴起为肿瘤的研究带来曙光, 自80年代末期开始, 人类基因组计划的实施和遗传学的进步更是大大推动了肿瘤生物学的研究, 数个抑癌基因被发现和克隆, 如p53, Rb, p16等. 染色体异常是肿瘤的重要特点, 包括原癌基因的扩增和抑癌基因的缺失等, 染色体异常分析在肿瘤研究中有重要意义. 理想的分析方法是对每个肿瘤标本进行全基因组的测序, 与其正常对照组织进行对比. 但目前的技术和资金所限, 尚缺乏可行性. 近年来, 比较基因组杂交(comparative genomic hybridization, CGH)成为检测基因组范围内基因拷贝数异常的主要方法, 而杂合性缺失(loss of heterozygosity, LOH)的研究也成为定位和发现抑癌基因的有效手段. 两种方法各有其优缺点, 如果两者结合能够得到更多的信息. 新近出现的高密度单核苷酸多态性(single nucleotide polymorphism, SNP)芯片, 可以同时检测基因拷贝数异常和LOH, 提供了基因异常的高解析度的图谱[1]. 为了揭示胰腺癌发生的分子机制, 发现新的抑癌基因候选基因, 我们应用高密度的SNP分析芯片, 研究17种胰腺癌细胞株基因组范围内的纯合性缺失及LOH.
人类胰腺癌细胞株17种, 其中AsPC-1, BxPC-3, Capan-1, Capan-2, CFPAC-1, HPAF-II, Hs 700T, Hs 766T, PANC-1, Panc 02.03, Panc 03.27, Panc 05.04, Panc 08.13, Panc 10.05, SU.86.86和SW 1990来自美国细胞库American Type Culture Collection(ATCC, Manassas, VA), MIA PaCa-2来自日本细胞库Japanese Collection of Research Bioresources(JCRB, Osaka, Japan), 细胞株培养液购自Sigma公司.
1.2.1 基因组DNA提取: 应用试剂盒PUREGENE DNA Isolation Kit(Gentra Systems, Minneapolis, MN)根据说明书提取细胞基因组DNA: 收集2×106细胞装入1.5 mL Eppendoff管, 加入300 μL细胞溶解液, 混匀, 加入1.5 μL RNase A液, 37 ℃温育30 min, 加入100 μL蛋白沉淀液, 反复离心取上清, 直至没有沉淀为止. 加入600 μL的1000 mL/L乙醇, 轻柔手摇50次, 混合. 离心后去上清, 试管中加入300 μL的700 mL/L乙醇. 多次颠倒试管, 洗DNA沉淀. 离心, 倒除乙醇, DNA于空气中干燥10-15 min. 加入50 μL的DNA水化液. 分光光度计测其浓度和纯度.
1.2.2 芯片制作和分析: Affymetrix基因芯片系统包括全自动芯片洗涤工作站, 高分辨率芯片扫描仪和杂交箱. 根据GeneChip Human Mapping 100 K Array操作手册, 从细胞株中提取DNA后, 250 ng DNA用2种限制性内切酶XbaⅠ或HindⅢ消化, 末端连接adapter后进行PCR. PCR产物片段化后, 末端进行标志(labeling), 然后与Affymetrix公司的GeneChip®Human Mapping 100 K Mapping microarray上的探针杂交. 检测基因芯片上近100 000个SNP探针的荧光信号, 应用分析软件Copy Number Analyzer for the Affymetrix GeneChip Mapping 100 K array (CNAG), 自动通过分析信号的强弱判定拷贝数目, 通过杂合性信号的有无分析胰腺癌基因组的LOH的存在与否[2].
1.2.3 纯合性缺失结果验证: 分析17种胰腺癌细胞的数据, 筛选可能与胰腺癌发生、发展有关的基因, 用PCR验证纯合性缺失. 设计基因特异的引物扩增细胞株的DNA, 同时提取健康人的DNA作为正常对照, 蒸馏水作为阴性对照. 引物设计是应用软件GENETYX-WIN Ver 7.0, 并由Invitrogen(Carlsbad, CA)公司合成. PCR仪是 Applied Biosystems(Foster City, CA)GeneAmp PCR System 9600.
应用SNP芯片和CNAG软件详细分析胰腺癌细胞株的基因组的异常变化, 绘制了基因组图谱. Hs 766T细胞株的全基因组图谱, 红色的点是每个SNP的原始荧光信号, 蓝色曲线是5个连续的SNP点信号综合分析后得到的平均值(箭头所指处拷贝数为0, 即纯合性缺失), 染色体下方的绿色条表示杂合性信号, 无杂合性信号的区域表示LOH阳性(图1). 胰腺癌基因组出现多处DNA拷贝数异常, Hs 766T细胞株在8p, 9p, 10q及20p等多处出现纯合性缺失(经位点分析, 8p, 9p, 10q处无已知基因, 20p处有一个基因).
筛选可能与胰腺癌发生和发展有关的含基因区域31个, 应用特异性引物进行PCR扩增验证纯合性缺失, 经过PCR验证, 26个区域确实为纯合性缺失, 芯片的准确度为83.9%. 经过PCR证实的26个有基因的纯合性缺失区域位置及所涉及基因和细胞. 这些缺失区域中, 14个区域只涉及1个基因, 11区域涉及2个基因, 1区域涉及4个基因, 平均每个区域1.29个基因(表1).
| 染色体号 | 染色体区带 | 起始位置(kb) | 终止位置(kb) | 涉及基因 | 细胞株 |
| 3 | 3p14.2 | 60 152 | 60 449 | FHIT | HPAF-II |
| 3 | 3p14.2 | 60 215 | 60 273 | FHIT | AsPC-1 |
| 6 | 6q22.33 | 129 215 | 129 220 | LAMA2 | MIA PaCa-2 |
| 6 | 6q25.3 | 156 991 | 157 399 | ARID1B | MIA PaCa-2 |
| 8 | 8p22 | 15 119 | 15 775 | TUSC3 | MIA PaCa-2 |
| 8 | 8q23.3 | 114 310 | 114 656 | CSMD3 | MIA PaCa-2 |
| 9 | 9p22.1-p21.2 | 19 726 | 26 039 | MTAP, CDKN2A | Capan-1 |
| 9 | 9p21.3 | 20 487 | 23 511 | MTAP, CDKN2A | MIA PaCa-2 |
| 9 | 9p21.3 | 20 866 | 22 041 | MTAP, CDKN2A | SU.86.86 |
| 9 | 9p21.3 | 21 204 | 22 895 | MTAP, CDKN2A | BxPC-3 |
| 9 | 9p21.3 | 21 948 | 22 090 | CDKN2A, CDKN2B | Panc 03.27 |
| 9 | 9p21.3 | 21 948 | 22 185 | CDKN2A, CDKN2B | Panc 08.13 |
| 9 | 9p21.3 | 21 948 | 22 185 | CDKN2A, CDKN2B | PANC-1 |
| 9 | 9p21.3 | 21 971 | 22 185 | CDKN2A, CDKN2B | Panc 05.04 |
| 9 | 9q32 | 112 811 | 113 577 | DEC1, TNC, TNFSF15, TNFSF8 | BxPC-3 |
| 10 | 10p12.33-p12.31 | 18 233 | 18 738 | SLC39A12, CACNB2 | BxPC-3 |
| 10 | 10q22.1 | 72 560 | 73 052 | CDH23, PSAP | BxPC-3 |
| 18 | 18q21.1 | 46 937 | 46 997 | RKHD2 | Panc 05.04 |
| 18 | 18q21.1 | 46 937 | 47 606 | RKHD2 | BxPC-3 |
| 18 | 18q21.2 | 49 275 | 49 712 | DCC | MIA PaCa-2 |
| 18 | 18q21.2 | 49 977 | 49 987 | MBD2 | MIA PaCa-2 |
| 20 | 20p12.1 | 15 048 | 15 087 | C20orf133 | Hs 766T |
| 21 | 21q22.3 | 41 752 | 41 802 | TMPRSS2 | BxPC-3 |
| 21 | 21q22.3 | 42 289 | 42 472 | ZNF295, UMODL1 | BxPC-3 |
| 23 | xp11.3 | 42 956 | 43 424 | EFHC2 | MIA PaCa-2 |
| 23 | Xq26.3 | 136 515 | 136 773 | FGF13 | Capan-1 |
应用SNP芯片, 在检测基因拷贝数的同时, 根据杂合性信号的有无, 可以判定LOH的存在与否, 可以显示每种胰腺癌细胞株的每一条染色体臂LOH情况. 17种胰腺癌细胞基因组中, 每一种细胞都有不同程度的LOH, 其中Panc 08.13细胞的22条染色体臂发生LOH, 频率最高(50.0%), 其次是Hs 700T和MIA PaCa-2细胞21条染色体臂有LOH(47.7%). Panc 05.04出现频率最低, 只有5条染色体臂发生LOH(11.4%).
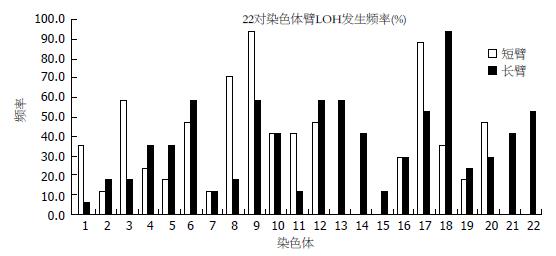
胰腺癌细胞株基因组范围内不同染色体臂出现LOH频率不同, 图2显示22对染色体臂LOH出现频率. 高频率的(50%以上)LOH出现在染色体臂9p(16/17, 94.1%), 18q(16/17, 94.1%), 17p(15/17, 88.2%), 8p(12/17, 70.6%), 3p(10/17, 58.8%), 6q(10/17, 58.8%), 9q(10/17, 58.8%), 12q(10/17, 58.8%), 13q(10/17, 58.8%), 17q(9/17, 52.9%)和22q(9/17, 52.9%).
胰腺癌由于解剖位置特殊和生物学侵袭性高, 症状缺乏特异性, 易发生早期转移, 因而确诊时多属晚期, 除了手术之外, 尚无其他的有效治疗方法. 术后5年生存率不到5%[3-4], 是预后最差的消化道恶性肿瘤之一. 同其他肿瘤一样, 胰腺癌的发生、发展是一个多阶段, 受多基因调控的过程, 由多种肿瘤相关基因表达失常或肿瘤抑制基因失活所致.
基因组DNA的片段扩增或缺失在许多疾病的发生、发展中起重要作用. 最初检测基因组范围内基因拷贝数异常的方法是CGH[5], 原理是用两份标记不同荧光素的样品(检测样品和对照样品)同时与中期染色体进行杂交, 通过比较荧光信号强度, 快速而又直观地检测实验样品和对照样品之间基因组DNA的拷贝数量的差异. 在此基础上发展起来的比较基因组杂交芯片(array-CGH), 将DNA克隆或cDNAs做成微阵列, 代替中期染色体作为杂交靶, 使分辨率提高从20 Mb提高到<1 Mb[6]. Array-CGH中微阵列探针通常是PCR扩增的细菌人工染色体(bacterial artificial chromosome, BAC)克隆或cDNA分子. 近期出现的一种含有32 433 BAC芯片解析度提高到80 kb[7]. 多位学者研究胰腺癌的基因组异常. 有学者应用2464个BAC clones芯片, 解析度为1.4 Mb[8]; 有人结合BAC和cDNA芯片, 解析度大约1 Mb[9]; 有研究利用5400个BAC克隆芯片, 解析度大约420 kb[10]; 还有利用12 232个克隆cDNA芯片, 解析度大约300 kb[11]; 14 160 cDNA芯片, 解析度大约100.1 kb[12]; 26 000 cDNA芯片, 解析度大约60 kb[13].
除了解析度的限制, 这种检测方法的主要缺陷是CGH只能检测到扩增或缺失这种拷贝数的异常, 但不能发现Allelic homozygosity(AH), 即一个等位基因缺失, 而余下的一个等位基因扩增, 基因数目没有变化, 这需要研究LOH. 所谓LOH即一个位点上两个多态性的等位基因中的一个出现突变或缺失, 在肿瘤细胞中是一种常见的DNA变异. 抑癌基因的杂合性缺失会导致肿瘤的发生, 因此分析肿瘤组织LOH为定位和发现抑癌基因的有效手段. 全基因组扫描LOH传统方法包括微卫星标记法(polymorphic microsatellite markers)和限制性片段长度多态性标记(restriction fragment length polymorphisms, RFLPs). 但是这两种检测LOH方法的缺点是操作费时费力, 需要大量的样本DNA, 检测能力受制于标志物的量.
应用SNP芯片, 可以克服上述方法的缺点. SNP芯片的优点是需要的样本DNA量少, 解析度高, 可以在检测基因拷贝数的变化的同时, 判定LOH的存在与否. SNP主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性, 是人类可遗传的变异中最常见的一种, 占所有已知多态性的90%以上. 2004年美国Matthew Meyerson研究小组首次应用SNP芯片研究人类肿瘤全基因组拷贝数目异常[14], 此研究应用约10 000个SNP的芯片研究了肺癌和乳腺癌的基因组异常, 同时与传统CGH芯片进行了对比, SNP芯片解析度最高, 提供了更为精确的包含抑癌基因和癌基因的候选位点. Calhoun et al发现与微卫星标记法相比较, 应用SNP芯片可以提高120倍杂合性信号[15], 有利于LOH的研究. SNP芯片引起了学者关注, 应用于膀胱癌[16]、卵巢癌[17]、肺癌[18]及前列腺癌[19-20]等肿瘤的研究.
我们所用的GeneChip®Human Mapping 100 K Mapping microarray芯片上近100 000个SNP探针的荧光信号, 这些SNP位点间的中位物理距离接近8.5 kb,平均间距是23.6 kb, 这些高密度的探针, 使解析度明显提高, 有利于抑癌基因的筛选. 我们发现高频率的(50%以上)LOH出现在染色体臂9p, 18q, 17p, 8p, 3p, 6q, 9q, 12q, 13q, 17q和22q. 出现频率最高的9p和18q(16/17, 94.1%)上分别存在抑癌基因CDKN2A和SMAD4, 他们与胰腺癌的关系已经被广泛关注[21-25]. 也有研究发现12q, 17p和18q的LOH与胰腺癌预后不良有关[26]. 虽然一些高频率LOH的染色体臂上已经确定了抑癌基因的存在, 例如CDKN2A(9p), SMAD4(18q)及TP53(17p)等, 但是, 多数染色体臂上的杂合性缺失区域中需要进一步的研究. LOH研究提供了范围较广的抑癌基因的候选区域, 研究纯合性缺失可以提供更加小范围的候选区域. 经过验证的26个有基因的纯合性缺失区域, 平均每个区域1.29个基因, 这种高解析度为今后的研究提供了更加明确的方向. 这些纯合性缺失区域涉及的基因中, 9号染色体短臂上编码CDKN2A基因缺失出现频率最高, 此基因蛋白产物为P16蛋白, 是一种抑癌基因, 在胰腺癌及其他肿瘤中广泛研究[21-22,27-29]. FHIT基因是属于普遍脆性位点(common fragile sites, CFSs), 他是一种抑癌基因, 在胰腺癌等多种肿瘤癌变过程中发挥作用[30-31]. 有些基因形成基底膜的成分, 或者在细胞连接, 血管形成中有一定作用, 例如CDH23, LAMA2. 近来的研究也发现CDH23在胰腺癌中纯合性缺失[13], 但具体功能尚有待研究.
应用SNP芯片可以同时研究杂合性缺失和纯合性缺失, 提供一个更为精细的胰腺癌全基因组的"扫描图". 本研究为今后胰腺癌的研究提供了新的线索和方向, 筛选出的基因在胰腺癌临床标本中的变化以及基因功能分析有待进一步研究.
高密度单核苷酸多态性芯片可以同时检测基因组范围内的基因数目异常和杂合性缺失, 提供基因异常的高解析度的图谱.
谷俊朝, 主任医师, 首都医科大学附属北京友谊医院普外科; 刘海林, 主任医师, 上海交通大学医学院附属第九人民医院消化科
胰腺癌细胞株的全基因组范围内, 同时研究纯合性缺失和杂合性缺失可以提供更准确的抑癌基因的候选区域, 甚至精细到具体基因.
有学者应用芯片研究胰腺癌基因组异常, 提供了大量信息, 但是信息量过于繁多而粗糙, 难以确定具体基因, 有待提高解析度. 例如应用2464个BAC clones芯片, 解析度为1.4 Mb; 结合BAC和cDNA芯片, 解析度大约1 Mb; 利用5400个BAC克隆芯片, 解析度大约420 kb; 26 000 cDNA芯片, 解析度大约60 kb.
较前研究, 本研究应用的芯片有2个主要优势: 第一、解析度高, 这样使候选基因范围缩小, 更易于确定抑癌基因, 即如表1中所示, 每个区域候选基因为1-4个; 第二、此芯片较传统芯片相比, 可以同时研究基因组范围内的基因数目异常和杂合性缺失.
本研究为今后胰腺癌的研究提供了新的线索和方向, 可以研究筛选出的基因在胰腺癌临床标本中的变化以及基因功能.
单核苷酸多态性(SNP): 主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性, 是人类可遗传的变异中最常见的一种, 他在人类基因组中广泛存在, 平均每500-1000个碱基对中就有1个, 是继限制性片段长度多态性和微卫星标记之后的第三代遗传标记.
本文选题新颖, 结构合理, 参考文献较新, 具有一定的临床价值.
编辑: 李军亮 电编:郭海丽
| 1. | Matsuzaki H, Dong S, Loi H, Di X, Liu G, Hubbell E, Law J, Berntsen T, Chadha M, Hui H. Genotyping over 100,000 SNPs on a pair of oligonucleotide arrays. Nat Methods. 2004;1:109-111. [PubMed] [DOI] |
| 2. | Nannya Y, Sanada M, Nakazaki K, Hosoya N, Wang L, Hangaishi A, Kurokawa M, Chiba S, Bailey DK, Kennedy GC. A robust algorithm for copy number detection using high-density oligonucleotide single nucleotide polymorphism genotyping arrays. Cancer Res. 2005;65:6071-6079. [PubMed] [DOI] |
| 3. | Greenlee RT, Hill-Harmon MB, Murray T, Thun M. Cancer statistics, 2001. CA Cancer J Clin. 2001;51:15-36. [PubMed] |
| 4. | Yoshizawa K, Nagai H, Kurihara K, Sata N, Kawai T, Saito K. Long-term survival after surgical resection for pancreatic cancer. Hepatogastroenterology. 2001;48:1153-1156. [PubMed] |
| 5. | Kallioniemi A, Kallioniemi OP, Sudar D, Rutovitz D, Gray JW, Waldman F, Pinkel D. Comparative genomic hybridization for molecular cytogenetic analysis of solid tumors. Science. 1992;258:818-821. [PubMed] [DOI] |
| 6. | Pinkel D, Segraves R, Sudar D, Clark S, Poole I, Kowbel D, Collins C, Kuo WL, Chen C, Zhai Y. High resolution analysis of DNA copy number variation using comparative genomic hybridization to microarrays. Nat Genet. 1998;20:207-211. [PubMed] [DOI] |
| 7. | Ishkanian AS, Malloff CA, Watson SK, DeLeeuw RJ, Chi B, Coe BP, Snijders A, Albertson DG, Pinkel D, Marra MA. A tiling resolution DNA microarray with complete coverage of the human genome. Nat Genet. 2004;36:299-303. [PubMed] [DOI] |
| 8. | Gysin S, Rickert P, Kastury K, McMahon M. Analysis of genomic DNA alterations and mRNA expression patterns in a panel of human pancreatic cancer cell lines. Genes Chromosomes Cancer. 2005;44:37-51. [PubMed] [DOI] |
| 9. | Heidenblad M, Schoenmakers EF, Jonson T, Gorunova L, Veltman JA, van Kessel AG, Hoglund M. Genome-wide array-based comparative genomic hybridization reveals multiple amplification targets and novel homozygous deletions in pancreatic carcinoma cell lines. Cancer Res. 2004;64:3052-3059. [PubMed] [DOI] |
| 10. | Nowak NJ, Gaile D, Conroy JM, McQuaid D, Cowell J, Carter R, Goggins MG, Hruban RH, Maitra A. Genome-wide aberrations in pancreatic adenocarcinoma. Cancer Genet Cytogenet. 2005;161:36-50. [PubMed] [DOI] |
| 11. | Mahlamaki EH, Kauraniemi P, Monni O, Wolf M, Hautaniemi S, Kallioniemi A. High-resolution genomic and expression profiling reveals 105 putative amplification target genes in pancreatic cancer. Neoplasia. 2004;6:432-439. [PubMed] [DOI] |
| 12. | Aguirre AJ, Brennan C, Bailey G, Sinha R, Feng B, Leo C, Zhang Y, Zhang J, Gans JD, Bardeesy N. High-resolution characterization of the pancreatic adenocarcinoma genome. Proc Natl Acad Sci U S A. 2004;101:9067-9072. [PubMed] [DOI] |
| 13. | Bashyam MD, Bair R, Kim YH, Wang P, Hernandez-Boussard T, Karikari CA, Tibshirani R, Maitra A, Pollack JR. Array-based comparative genomic hybridization identifies localized DNA amplifications and homozygous deletions in pancreatic cancer. Neoplasia. 2005;7:556-562. [PubMed] [DOI] |
| 14. | Zhao X, Li C, Paez JG, Chin K, Janne PA, Chen TH, Girard L, Minna J, Christiani D, Leo C. An integrated view of copy number and allelic alterations in the cancer genome using single nucleotide polymorphism arrays. Cancer Res. 2004;64:3060-3071. [PubMed] [DOI] |
| 15. | Calhoun ES, Hucl T, Gallmeier E, West KM, Arking DE, Maitra A, Iacobuzio-Donahue CA, Chakravarti A, Hruban RH, Kern SE. Identifying allelic loss and homozygous deletions in pancreatic cancer without matched normals using high-density single-nucleotide polymorphism arrays. Cancer Res. 2006;66:7920-7928. [PubMed] [DOI] |
| 16. | Koed K, Wiuf C, Christensen LL, Wikman FP, Zieger K, Moller K, von der Maase H, Orntoft TF. High-density single nucleotide polymorphism array defines novel stage and location-dependent allelic imbalances in human bladder tumors. Cancer Res. 2005;65:34-45. [PubMed] |
| 17. | Thompson ER, Herbert SC, Forrest SM, Campbell IG. Whole genome SNP arrays using DNA derived from formalin-fixed, paraffin-embedded ovarian tumor tissue. Hum Mutat. 2005;26:384-389. [PubMed] [DOI] |
| 18. | Zhao X, Weir BA, LaFramboise T, Lin M, Beroukhim R, Garraway L, Beheshti J, Lee JC, Naoki K, Richards WG. Homozygous deletions and chromosome amplifications in human lung carcinomas revealed by single nucleotide polymorphism array analysis. Cancer Res. 2005;65:5561-5570. [PubMed] [DOI] |
| 19. | Liu W, Chang B, Sauvageot J, Dimitrov L, Gielzak M, Li T, Yan G, Sun J, Sun J, Adams TS. Comprehensive assessment of DNA copy number alterations in human prostate cancers using Affymetrix 100K SNP mapping array. Genes Chromosomes Cancer. 2006;45:1018-1032. [PubMed] [DOI] |
| 20. | Torring N, Borre M, Sorensen KD, Andersen CL, Wiuf C, Orntoft TF. Genome-wide analysis of allelic imbalance in prostate cancer using the Affymetrix 50K SNP mapping array. Br J Cancer. 2007;96:499-506. [PubMed] [DOI] |
| 21. | Li J, Weghorst CM, Tsutsumi M, Poi MJ, Knobloch TJ, Casto BC, Melvin WS, Tsai MD, Muscarella P. Frequent p16INK4A/CDKN2A alterations in chemically induced Syrian golden hamster pancreatic tumors. Carcinogenesis. 2004;25:263-268. [PubMed] [DOI] |
| 22. | Wada K. p16 and p53 gene alterations and accumulations in the malignant evolution of intraductal papillary-mucinous tumors of the pancreas. J Hepatobiliary Pancreat Surg. 2002;9:76-85. [PubMed] [DOI] |
| 23. | Sunamura M, Lefter LP, Duda DG, Morita R, Inoue H, Yokoyama T, Yatsuoka T, Abe T, Egawa S, Furukawa T. The role of chromosome 18 abnormalities in the progression of pancreatic adenocarcinoma. Pancreas. 2004;28:311-316. [PubMed] [DOI] |
| 24. | Jazag A, Ijichi H, Kanai F, Imamura T, Guleng B, Ohta M, Imamura J, Tanaka Y, Tateishi K, Ikenoue T. Smad4 silencing in pancreatic cancer cell lines using stable RNA interference and gene expression profiles induced by transforming growth factor-beta. Oncogene. 2005;24:662-671. [PubMed] [DOI] |
| 25. | Ijichi H, Otsuka M, Tateishi K, Ikenoue T, Kawakami T, Kanai F, Arakawa Y, Seki N, Shimizu K, Miyazono K. Smad4-independent regulation of p21/WAF1 by transforming growth factor-beta. Oncogene. 2004;23:1043-1051. [PubMed] [DOI] |
| 26. | Yatsuoka T, Sunamura M, Furukawa T, Fukushige S, Yokoyama T, Inoue H, Shibuya K, Takeda K, Matsuno S, Horii A. Association of poor prognosis with loss of 12q, 17p, and 18q, and concordant loss of 6q/17p and 12q/18q in human pancreatic ductal adenocarcinoma. Am J Gastroenterol. 2000;95:2080-2085. [PubMed] [DOI] |
| 27. | van Zutven LJ, van Drunen E, de Bont JM, Wattel MM, Den Boer ML, Pieters R, Hagemeijer A, Slater RM, Beverloo HB. CDKN2 deletions have no prognostic value in childhood precursor-B acute lymphoblastic leukaemia. Leukemia. 2005;19:1281-1284. [PubMed] [DOI] |
| 28. | Akervall J, Bockmuhl U, Petersen I, Yang K, Carey TE, Kurnit DM. The gene ratios c-MYC:cyclin-dependent kinase (CDK)N2A and CCND1:CDKN2A correlate with poor prognosis in squamous cell carcinoma of the head and neck. Clin Cancer Res. 2003;9:1750-1755. [PubMed] |
| 29. | Anzola M, Cuevas N, Lopez-Martinez M, Martinez de Pancorbo M, Burgos JJ. p16INK4A gene alterations are not a prognostic indicator for survival in patients with hepatocellular carcinoma undergoing curative hepatectomy. J Gastroenterol Hepatol. 2004;19:397-405. [PubMed] [DOI] |
| 30. | Smith DI, Zhu Y, McAvoy S, Kuhn R. Common fragile sites, extremely large genes, neural development and cancer. Cancer Lett. 2006;232:48-57. [PubMed] [DOI] |
| 31. | Iliopoulos D, Guler G, Han SY, Druck T, Ottey M, McCorkell KA, Huebner K. Roles of FHIT and WWOX fragile genes in cancer. Cancer Lett. 2006;232:27-36. [PubMed] [DOI] |