修回日期: 2008-03-14
接受日期: 2008-05-20
在线出版日期: 2008-05-28
目的: 在传统基因芯片技术基础上, 应用生物传感器技术, 研制一种能将基因芯片信号原位放大后达肉眼判读灵敏度, 无需专用基因芯片检测仪器就可使用, 易于在基层医疗单位推广的薄膜生物传感器基因芯片诊断系统.
方法: 在薄膜生物传感器基片基础上, 经过表面化学处理, 使特定的基因捕获探针在传感器表面固定, 形成特定检测目的生物传感器基因芯片. 并以乙肝病毒YMDD区的特异序列设计为例, 通过特定的YMDD区的捕获探针, 以矩阵的形式点样于传感器芯片表面来实现本系统. 同时, 纳米金标记的检测探针取代了传统芯片中的荧光标记探针. 扩增后的目的PCR片段与捕获探针、生物素标记探针、链亲蛋白纳米金探针进行反应, 最后得到探针-生物素-链亲蛋白-纳米金复合物, 并在芯片表面经生物传感器芯片将信号原位放大, 获得肉眼观的芯片信号并进行分析, 完成芯片诊断. 以乙型肝炎病毒YMDD突变为例, 观察该芯片系统对临床诊断标本诊断的可靠性.
结果: 基因芯片的检测信号经生物传感器原位放大后能肉眼判读或借助普通数码照像机或计算机扫描, 根据信号出现的特定位置即可确定突变的类型. 且该生物传感器基因芯片系统信噪比高, 在人工合成的寡核苷酸及临床血清的检测中, 均可实现生物芯片阴阳性信号完全的有或无的判读; 临床血清标本检测证实, 使用该传感器芯片系统对前期经过测序确定为YMDD突变的23份临床血清结果与测序结果完全一致.
结论: 薄膜生物传感器基因芯片集纳米材料、生物传感器技术及原位放大技术为一体, 实现了信号的肉眼判读, 具有通用性高, 准确可靠, 无需大型设备, 易于在基层医疗单位推广使用.
引文著录: 宋家武, 信芝, 姚蓝, 李啸峰, 唐健熹, 周小军, 吴波, 孙爱静, 吴洲清. 高灵敏度薄膜生物传感器基因芯片系统的研制. 世界华人消化杂志 2008; 16(15): 1628-1633
Revised: March 14, 2008
Accepted: May 20, 2008
Published online: May 28, 2008
AIM: To develop a sensitive and less instrument-dependent clinical microarray system, in which the microarray signal can be amplified in situ and identified by naked eyes.
METHODS: A group of capture probes for a specific target nucleic acid that was made according to a specific region such as hepatitis B virus (HBV) YMDD motif were arrayed on a thin-film biosensor in a well. A set of detected probes labeled with Au nanoparticle were used to take place of the fluorence labeled probes in classic microarray. The single-strand PCR product was reacted with the capture and detected probes and the deposition of capture-biotin-streptin-Au nanoparticle compound appeared on the surface of the microarray. After in situ amplification in this biosensor based system, we could read the signal on this chips by naked eyes or the digital camera. HBV YMDD mutation detection was applied to identify the sensitivity and specificity of the microarray system.
RESULTS: The signal of the biosensor microarray could be acquired by common camera or naked eyes without any instruments and we could determine the kind of mutation according to the place of the positive signal. The signal-noise ratio were high enough to make the signal absolutely yes and no both in synthesized target oligos and serum samples. The microarray could identify a single base change of selected lamivudine resistance-related mutation as well as multiple mutations at the same time with a high stability, sensitivity, and specificity. We used the biosensor system to test 23 serum samples with YMDD mutation, and the result was coincident with the sequencing result and the signal could be aquired by naked eyes.
CONCLUSION: The thin-film based microarray system which exploits nanoparticle material and biosensor technique can amplify the signal in situ that can be detected by simple instruments or even unaided eyes. Its attractive features are the nonintervention of instrumentation required to detect signal, as well as its high versatility and accuracy.
- Citation: Song JW, Xin Z, Yao L, Li XF, Tang JX, Zhou XJ, Wu B, Sun AJ, Wu ZQ. Development of clinical highly sensitive biosensor-based microarray system. Shijie Huaren Xiaohua Zazhi 2008; 16(15): 1628-1633
- URL: https://www.wjgnet.com/1009-3079/full/v16/i15/1628.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i15.1628
基因芯片技术自问世以来[1], 以其基于核酸杂交的检测高度特异性, 检测时的高通量以及平行分析的特点[2-5], 1998年被美国科学促进会评为当年十大科技进展之一[6], 并将为临床基因诊断提供高效、高度特异的基因诊断技术, 为医学诊断及治疗技术进步做出杰出的贡献, 解决传统基因诊断技术所不能解决的系列难题[7-9].
传统的基因芯片技术, 无论是微阵列, 还是原位合成的寡核苷酸基因芯片, 均使用荧光标记探针技术[10-11], 应用时需要昂贵的激光扫描设备[12], 此外, 传统技术不能完全消除阴性信号, 阴阳性信噪比对比低, 存在人为判断其阴阳性结果.这些因素严重制约了基因芯片技术在临床中的广泛应用. 本款基因芯片在前期系列研究基础之上[13-16], 应用金银纳米材料标记技术及生物传感器技术, 将检测信号特异性原位放大, 肉眼或只需简单光学设备和仪器就能实现信号的准确判读, 较好地解决了基因芯片在临床应用中的瓶颈问题.
在直径约10-20 mm硅片上按真空气相沉积法镀膜镀以一定厚度的氮化硅, 再用旋转真空镀膜机将TSPS(T结构聚氨二甲基硅烷烃, UCT, Bristol, PA)的薄膜[17-18], 制备出相应的生物传感器基片, 再镀以多聚苯丙氨酸-赖氨酸涂层, 最后经1-10 fmol/L盐酸琥珀酰亚胺基肼烟酸盐处理约20 min, 清水洗净供芯片制作.
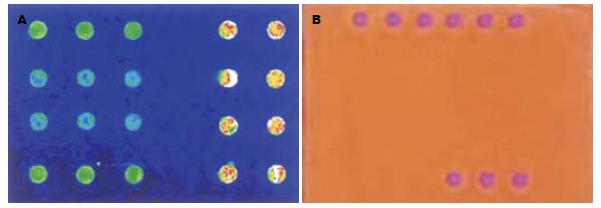
为实现对突变位点的特异性诊断, 我们以乙型肝炎病毒L528M突变位点设计了2个位点检测的的检测探针, 并在标记探针的3'端进行生物素标记, 由美国(Invitrogen Life Tech)合成, HPLC纯化, 以便于与金纳米材料标记链亲蛋白(streptavidin)商业化纳米金探针交联. 基因捕获探针C1的设计: 经检索分析现有基因库(ww3.ncbi.nih.nlm.gov)中所有乙型肝炎病毒(Hepatitis B virus, HBV)基因序列, 选择HBV高度保守的P基因YMDD区附近区域片段, 设计相应的探针并由美国合成(Invitrogen Life Tech)相应的探针[13]. 上述高效液相色谱纯化的捕获探针, 由珠海赛乐奇生物技术有限公司按以下矩阵(图1A)用德国Gisem Nanoplotter芯片点样仪进行点样, 室温下与生物传感器芯片表面的化学涂层进行化学反应, 形成共价结合的化合物, 经清水漂洗, 干燥, 备用. 传统基因芯片的制备方法同我们研究相同. 采用化学合成的含有L528M突变的DNA寡核苷酸(表1)、我室保存的YMDD突变质粒及长期使用拉米夫定的患者血清作为阳性标准品[13-14]. 按以往文献报道方法[13-15], 制备相应的传统基因芯片, 并将上述阳性标准品及扩增的PCR产物进行检测. 目的基因片段的PCR扩增按以下引物: L: 5'-GAT GTG TCT GCG GCG T-3', R: 5'-GTA AACTGA GCC AGG AGA AA(GenBank 4490408)为引物, 在50 μL的PCR扩增体系中加入20 mmol/L Tris-HCl, 50 mmol/L KCl, 2 mmol/L MgCl2及0.2 mmol/L dATP, dGCT, dTTP和dCTP(Amersham, UK)及100 nmol/L引物1 μL, 再加入Taq酶33.34 nkat, 经95℃ 5 min预变性后, 再以 94℃ 30 s, 50℃ 30 s和72℃ 30 s为一周期, 30个循环扩增. 扩增后的 PCR扩增产物95℃加热3 min, 迅速置于冰上; 取10 μL PCR扩增产物加入到25 μL 2×连接酶混合液, 加水至50 μL(以上试剂由珠海赛乐奇生物技术有限公司提供), 并据公司操作说明按以下步骤进行: 将60℃预热连接酶混合液芯片上, 在60℃反应10 min; 0.01 mol/L NaOH洗涤3×5-10 s; 再将芯片置0.1×SSC中洗涤3×1-2 min; 空气干燥, 将制备的streptoavidin金纳米探针每片3滴滴于芯片表面, 常温反应5 min; 用0.1×SSC洗涤芯片3次×1-2 min; 并在最后用干净空气吹干芯片表面, 凉干后扫描或数码相机照相, 计算机判读结果.
| L528M位点突变检测捕获探针 | AAAAAAAAAACGCAAAATACCTATGGGAGTGGGCCTCAGTCCGTTTCTCT |
| AAAAAAAAAACGCAAAATACCTATGGGAGTGGGCCTCAGTCCGTTTCTCA | |
| L528M检测标记探针 | TGGCTCAGTTTACTAGTGCC-biotin |
| 乙型肝炎YMDD区PCR扩增引物 | TGCACCTGTATTCCCATCC |
| TTTGTATTAGGAGGCTGTAG | |
| L528M阳性单链寡核苷酸 | L528M A GGCACTAGTAAACTGAGCCAAGAGAAACGGACTGAGGCCC |
| L528M T GGCACTAGTAAACTGAGCCATGAGAAACGGACTGAGGCCC |
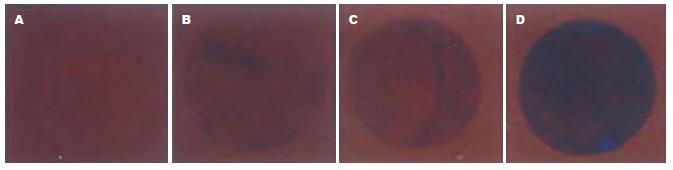
我们通过改变传统的探针, 将特异性杂交探针3'端标以生物素分子, 该特异性杂交探针再与可见包被有纳米金颗粒的链亲蛋白结合, 形成纳米金探针[19], 该探针在可见光条件下, 即可实现信号的显示(图2), 成功实现了传统荧光探针至可见光探针的转化, 为实现信号的肉眼判读奠定了基础.基于纳米金探针有效地替代了传统芯片基于荧光标记的探针, 但其灵敏度, 肉眼条件下的观察, 与荧光探针标记的激光扫描仪下获得的检测信号比较, 灵敏度较低, 不易形成稳定的阳性信号(宋家武, 未发表资料). 而本芯片研究结果显示: 生物传感器基因芯片能有效地在原位放大基因芯片上杂交后的生物信号, 在肉眼条件下, 或借助简单的数码相机, 获得清析的阳性和阴性信号(图1B-C).
为研究在肉眼可观察的条件下, 本芯片可检测的DNA水平, 我们合成了L528M单链DNA, 分别稀释成1、0.1、0.01、0.001 μmol/L, 取1 μL进行检测, 其最低检测浓度为0.01 μmol/L, 即最低检测病毒载量浓度为1×10-14 mol/L, 图2). 专用的生物传感器基因芯片信号处理技术, 有效增强了信号特异性; 显著增加阴阳性信号对比度, 并有效地消除了背景信号. 我们在芯片系统中引入了珠海赛乐奇生物技术有限公司的专利信号处理技术[16], 在生物传感器芯片有效放大检测信号的同时, 有效地降低了背景信号, 与传统基因芯片比较, 真正实现了检测阳性信号与阴性信号有和无的分辨(图3A-B).
为了验证YMDD突变检测基因芯片对YMDD突变检测的特异性, 我们应用化学合成的含有YMDD突变位点的寡核苷酸链作为阳性标准品及YMDD突变质粒对芯片以往经测序证实的23份血清, 对其中的共46个次突变位点标本进行了检测, 结果如前面图显示: 所有样本的突变均能实现完全有或无显示, 阴阳性结果对比显著, 阴性结果完全无信号.
基因芯片技术是1990年代发展起来的一项高新生物技术[1,6,20-21], 他可以实现一次检测全基因组在特定条件下的基因表达或检测全基因组或特定的核苷酸或序列的改变. 在各种基因相关性疾病, 如传染病和遗传性疾病的诊断、基因多态性分析等具有独特的应用前景[22-26].
基于荧光标记探针的基因芯片检测技术是其使用时需要大型设备的根本原因[27]. 由于其信号弱, 必须经过激发, 才能发光, 需要精密而昂贵的激光扫描设备. 在临床检测应用过程中, 由于检测单位需要投资购置大的激光扫描设备, 一方面, 显著增加了进行相关检测的投资成本, 另一方面, 亦直接增加了应用的成本, 显著地限制了该技术的应用范围, 并成为该技术在临床推广中的瓶颈问题. 因此, 从技术上解决其需要使用基因芯片扫描仪的必备条件是, 改变其基于荧光分子的探针标记方法和技术. 早期Taton et al研究报告提示[28], 基因芯片的检测信号可望通过纳米金材料技术的应用, 实现基因芯片检测信号由传统的荧光信号转化为可见光判读, 从而有望解决技术推广中需要荧光扫描仪的难题. 本研究结果表明, 纳米金标记的探针技术, 完全可以实现基因芯片信号的可见光判读. 然而, 在现有临床检测条件下, 现有纳米材料可见光探针技术在临床应用过程中, 存在着灵敏度偏低, 普通光学设备判读时, 灵敏度不够和获得信号欠稳定的问题(宋家武, 未发表资料). 因此, 其检测信号有待更进一步的放大, 其技术有待更进一步的完善.
生物传感器技术, 品种繁多, 对待检信号的实现方法很多, 如光电传感, 压电传感, 化学传感等传感器技术[29-31]. 本芯片平台引入了生物传感器原位放大技术, 使临床检测用的基因芯片信号, 转化为可见光信号, 并经原位放大, 达到肉眼观水平, 从而有效地解决了这一棘手的推广问题, 为临床上广泛使用基因芯片诊断多基因, 多位点突变疾病, 提供了一种全新的诊断技术. 我们开发研制的该款基因芯片有效地整合当前的多项高新技术, 特别是创造性地将生物传感器技术与基因芯片技术、纳米材料技术及通用纳米材料探针技术进行整合, 并充分利用了多项技术特点, 实现了基因芯片生物检测信号的原位放大, 显著地提高了检测的灵敏度, 实现了基因信号的肉眼判读或应用简单数字成像设备如数码相机、CCD成像即可获取信号, 从而从技术上解决了不需要荧光扫描仪等大型设备的难题. 与传统的基因芯片相比, 本技术和产品在灵敏度上, 较之传统的基因芯片技术, 将信号放大了数个数量级, 灵敏度显著增加. 另一方面, 由于杂交洗涤条件的革新, 在确保了特异性阳性信号实现的条件下, 由于杂交洗脱条件严谨, 非特异性信号降低至最低程度. 与传统的基因芯片技术相比, 真正实现了阴阳性信号的特异性检测. 同时, 该基因芯片开发出能基于高温连接酶技术的特异性信号实现方式, 有效地降低了背景, 使信噪比显著提高, 在信号上完全实现了有和无的分辨.
总之, 本研究结果证明, 本款基因芯片集生物传感器基因芯片技术与纳米材料技术为一体, 实现了基因芯片信号原位特异性放大, 达到肉眼判读的水平, 具有高度的灵敏度、特异性及可重复性, 背景清晰, 能特异性鉴别单个或多个基因位点的突变, 操作简单, 成本低廉, 有效地解决了传统基因芯片在应用过程中, 需要大型检测设备、不便于推广的瓶颈问题, 是一种既经济又可靠的新型基因芯片技术.
基因芯片技术是1990年代发展起来的一项高新生物技术, 以其检测时的高通量以及平行分析的特点, 一直为专家学者所瞩目, 被越来越多地应用于临床诊断. 但传统的芯片平台因基于荧光标记技术, 应用时需要昂贵的扫描设备以及操作复杂, 技术要求高, 在临床应用中一直难以推广普及.
肖恩华, 教授, 中南大学湘雅二医院放射教研室.
近年来, 芯片技术的发展较快, 特别是临床应用类芯片, 成为各国竞相发展的领域. 其突破点主要是从改变其基于荧光分子的探针标记技术及降低检测设备成本入手.
本研究实现了基因芯片生物检测信号的原位放大, 达到肉眼观水平.
本款基因芯片实现了基因芯片信号的肉眼判读, 集高度的灵敏度、特异性及可重复性于一体, 背景清晰, 能特异性鉴别单个或多个基因位点的突变, 操作简单, 成本低廉, 是一种既经济又可靠的新型基因芯片技术, 具有广阔的临床应用前景.
本研究内容新颖, 论据充分, 结论明确, 讨论条理分明, 但实验对照的设计欠合理可靠.
编辑: 师忠芳 电编:郭海丽
| 1. | Fodor SP, Read JL, Pirrung MC, Stryer L, Lu AT, Solas D. Light-directed, spatially addressable parallel chemical synthesis. Science. 1991;251:767-773. [PubMed] [DOI] |
| 2. | Pellois JP, Zhou X, Srivannavit O, Zhou T, Gulari E, Gao X. Individually addressable parallel peptide synthesis on microchips. Nat Biotechnol. 2002;20:922-9226. [PubMed] [DOI] |
| 3. | Schena M, Shalon D, Heller R, Chai A, Brown PO, Davis RW. Parallel human genome analysis: microarray-based expression monitoring of 1000 genes. Proc Natl Acad Sci U S A. 1996;93:10614-10619. [PubMed] [DOI] |
| 4. | Lockhart DJ, Winzeler EA. Genomics, gene expression and DNA arrays. Nature. 2000;405:827-836. [PubMed] [DOI] |
| 5. | Xiao Y, Segal MR, Yang YH, Yeh RF. A multi-array multi-SNP genotyping algorithm for Affymetrix SNP microarrays. Bioinformatics. 2007;23:1459-1467. [PubMed] [DOI] |
| 7. | Gerhold DL, Jensen RV, Gullans SR. Better therapeutics through microarrays. Nat Genet. 2002;32 Suppl:547-551. [PubMed] [DOI] |
| 8. | Ragoussis J, Elvidge G. Affymetrix GeneChip system: moving from research to the clinic. Expert Rev Mol Diagn. 2006;6:145-152. [PubMed] [DOI] |
| 9. | Shomaker TS, Ward K. Microarray technology in biomedical research. Hawaii Med J. 2006;65:253-256. [PubMed] |
| 11. | Schena M, Shalon D, Davis RW, Brown PO. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science. 1995;270:467-470. [PubMed] [DOI] |
| 13. | Song J, Lin J, Ni X, Kong X, Wu H, Xie Y, Mao Y. Development of lamivudine-resistance microarray and its reliability in clinical examination. Zhonghua Ganzangbing Zazhi. 2002;10:468. [PubMed] |
| 14. | Song JW, Lin JS, Kong XJ, Liang KH. Clinical study of oligonucleotide microarray on monitoring the lamivudine-resistance mutations in hepatitis B virus. Zhonghua Ganzangbing Zazhi. 2003;11:361-363. [PubMed] |
| 16. | 宋 家武, 杨 又春. 一种高灵敏度生物传感器基因芯片及临床诊断技术. 中华人民共和国知识产权局 CN200610024197.8. |
| 17. | Jenison R, La H, Haeberli A, Ostroff R, Polisky B. Silicon-based biosensors for rapid detection of protein or nucleic acid targets. Clin Chem. 2001;47:1894-1900. [PubMed] |
| 18. | Jenison R, Yang S, Haeberli A, Polisky B. Interference-based detection of nucleic acid targets on optically coated silicon. Nat Biotechnol. 2001;19:62-65. [PubMed] [DOI] |
| 19. | Huhtinen P, Soukka T, Lovgren T, Harma H. Immunoassay of total prostate-specific antigen using europium(III) nanoparticle labels and streptavidin-biotin technology. J Immunol Methods. 2004;294:111-122. [PubMed] [DOI] |
| 20. | Marshall A, Hodgson J. DNA chips: an array of possibilities. Nat Biotechnol. 1998;16:27-31. [PubMed] [DOI] |
| 22. | Belgrader P, Benett W, Hadley D, Long G, Mariella R Jr, Milanovich F, Nasarabadi S, Nelson W, Richards J, Stratton P. Rapid pathogen detection using a microchip PCR array instrument. Clin Chem. 1998;44:2191-2194. [PubMed] |
| 23. | Debouck C, Goodfellow PN. DNA microarrays in drug discovery and development. Nat Genet. 1999;21:48-50. [PubMed] [DOI] |
| 24. | Lipshutz RJ, Fodor SP, Gingeras TR, Lockhart DJ. High density synthetic oligonucleotide arrays. Nat Genet. 1999;21:20-24. [PubMed] [DOI] |
| 25. | Sapolsky RJ, Hsie L, Berno A, Ghandour G, Mittmann M, Fan JB. High-throughput polymorphism screening and genotyping with high-density oligonucleotide arrays. Genet Anal. 1999;14:187-192. [PubMed] |
| 26. | Jain KK. Applications of biochips: from diagnostics to personalized medicine. Curr Opin Drug Discov Devel. 2004;7:285-289. [PubMed] |
| 27. | Kricka LJ. Nucleic acid detection technologies -- labels, strategies, and formats. Clin Chem. 1999;45:453-458. [PubMed] |
| 28. | Taton TA, Mirkin CA, Letsinger RL. Scanometric DNA array detection with nanoparticle probes. Science. 2000;289:1757-1760. [PubMed] [DOI] |
| 29. | Ferrari M, Cremonesi L, Bonini P, Stenirri S, Foglieni B. Molecular diagnostics by microelectronic microchips. Expert Rev Mol Diagn. 2005;5:183-192. [PubMed] [DOI] |
| 30. | Ferrari M, Stenirri S, Bonini P, Cremonesi L. Molecular diagnostics by microelectronic microchips. Clin Chem Lab Med. 2003;41:462-467. [PubMed] [DOI] |
| 31. | Kumar Khanna V. Existing and emerging detection technologies for DNA (Deoxyribonucleic Acid) finger printing, sequencing, bio- and analytical chips: a multidisciplinary development unifying molecular biology, chemical and electronics engineering. Biotechnol Adv. 2007;25:85-98. [PubMed] [DOI] |