修回日期: 2008-02-29
接受日期: 2008-05-02
在线出版日期: 2008-05-08
目的: 分析来自黄疸和先天性巨结肠(hirsch-sprung's disease, HD)患儿的人巨细胞病毒临床分离株UL141片段的序列变化, 以及这种变化与人巨细胞病毒感染导致不同消化系疾病之间的关系.
方法: 对荧光定量PCR方法检测HCMV-DNA阳性的临床低传代分离株进行UL141基因全序列PCR扩增, 对扩增阳性的标本进行测序及分析.
结果: 15株临床低传代分离株在Toledo株UL141基因227位均缺失一个碱基T, 因此产生两个新的ORF(UL141A和UL141B). 与Toledo株相应片段比较, UL141A预测蛋白质第75位氨基酸后序列和翻译后修饰位点产生大量变异. UL141B的核苷酸和氨基酸序列均高度保守.
结论: 来自黄疸和先天性巨结肠患儿的人巨细胞病毒临床分离株的UL141片段产生两个新的ORF, 这两个ORF在黄疸和先天性巨结肠分离株之间均未发现特异性变异
引文著录: 马艳萍, 阮强, 何蓉, 齐莹, 孙峥嵘, 吉耀华. 人巨细胞病毒UL141A和B片段在黄疸和先天性巨结肠分离株中的序列分析. 世界华人消化杂志 2008; 16(13): 1471-1475
Revised: February 29, 2008
Accepted: May 2, 2008
Published online: May 8, 2008
AIM: To study the sequence variance of human cytomeglovirus (HCMV) UL141 in clinical isolates from infants with jaundice or Hirschsprung's disease (HD), and to find the relationship between the variance and the above two diseases.
METHODS: Polymerase chain reaction (PCR) was performed to amplify the entire UL141 region of clinical isolates, which were proven positive for HCMV-DNA by real time PCR. The PCR products were sequenced and analyzed
RESULTS: There was a nucleotide deletion at the site 227 in clinical isolates and two new UL141 open reading frames, named UL141A and UL141B, were found. In clinical isolates, the predicted proteins of UL141A were hypervariable after the position 75 of amino acid sequence. The predicted protein of UL141B was highly conserved.
CONCLUSION: There are two new UL141 open reading frames in clinical isolates from infants with jaundice or HD. The two open reading frames have no specific variation between the two groups of isolates.
- Citation: Ma YP, Ruan Q, He R, Qi Y, Sun ZR, Ji YH. Sequence analysis of human cytomeglovirus UL141 A and B genes in strains isolated from infants with jaundice or Hirschsprung's disease. Shijie Huaren Xiaohua Zazhi 2008; 16(13): 1471-1475
- URL: https://www.wjgnet.com/1009-3079/full/v16/i13/1471.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i13.1471
人巨细胞病毒(human cytomegalovirus, HCMV)属于β-疱疹病毒亚科, 人群中大多数呈现不显性感染或潜伏感染. 孕妇原发或复发感染均可引起新生儿宫内感染或围生期感染, 可导致小儿多个器官和系统受累, 消化系统是常见HCMV侵袭部位[1-2]. HCMV感染的不同临床表现一方面可能与宿主免疫状态有关, 亦可能与不同临床分离株基因及其编码产物的多态性有关[3-5]. 实验室株AD169的全部基因组测序工作已经完成. 1996年Cha et al[6]在Toledo的低传代分离株基因组UL/b'区中发现在AD169株中不存在的19个开放阅读框架(open reading frame, ORF), 依次命名为UL133-UL151. 推测, 实验室株AD169在细胞培养反复传代中出现了UL/b'区遗传信息的丢失, 引起了该区基因的缺失, 从而导致HCMV实验室株的复制水平、毒力和致病力明显减弱. 因此, 这19个基因可能在病毒潜伏、复制、逃避机体免疫和不同组织细胞嗜性等方面起重要作用. 我们应用PCR和DNA测序的方法, 分析来自黄疸和先天性巨结肠患儿的HCMV临床分离株UL141片段及其编码蛋白氨基酸序列的变化.
23株低传代临床分离株均来自1988-1993年住院患儿, 其中黄疸患儿(J)分离株17株, 先天巨结肠患儿(C)分离株6株. 年龄<14 mo. 标本取自HCMV分离实验, -70℃保存. 2000年应用荧光定量PCR方法[7]检测HCMV DNA, 结果均为阳性.
取HCMV低传代分离株接种细胞的培养上清液与等量裂解液(华美生物工程公司)混合, 煮沸15 min, 作为扩增模板. 按照Toledo株序列(参考株, GenBank accession numberU33331), 应用引物设计软件Primer premier5.0设计用于扩增HCMV UL141片段的全序列引物、鉴定引物及分段PCR扩增分段引物(表1). 引物合成由北京奥科公司完成. 引物也用于UL141基因测序. 应用全序列引物及搭配引物(全序列及鉴定引物之一)和分段PCR引物扩增UL141基因全序列. rTaq酶、10×Buffer、dNTP mixture为TaKaRa公司产品. 制备反应体系如下: 1×Buffer, MgCl2 1.5 mmol/L, dNTP mixture 0.2 mmol/L, 上下游引物各150 ng, rTaq酶0.5 U, 模板3.5 µL, 用双蒸水补至总反应体积50 µL. PCR循环条件见表1. PCR产物经15 g/L琼脂糖凝胶电泳后, 紫外检测仪下观察扩增结果. 测序前所有扩增产物应用TaKaRa公司的PCR Fragment Recovery kit切胶回收目的DNA片段, 以去除非特异产物带的干扰. PCR扩增阳性标本测序由上海联合基因公司完成. 通过正反两个方向分别对目的基因的正链及负链进行测序,以增加实验结果的可信性. 应用生物信息学软件DNAClub、BioEdit、Genedoc、DNASis、DNAStar等完成序列分析. 用Sequin软件向GenBank提交整理后的HCMV UL141 ORF序列.
| 引物 | 序列 | 位置(nt) | 产物长度(bp) | 扩增条件 | |
| Ul141 | 上游 | 5'CGTTCCTTCTTAAACATCCGA3' | 5070 | 1418 | 96℃ 4 min, 95℃ 45 s, 53℃ 1 min |
| 下游 | 5'ACAAAGTATCCGAATAACCACC3' | 6488 | 72℃ 2 min, 35 cycles, 72℃ 10 min | ||
| Ul141a1 | 上游 | 5'CGTTCCTTCTTAAACATCCGA3' | 5070 | 617 | 95℃ 4 min, 94℃ 45 s, 53℃ 1 min |
| 下游 | 5'CGGTACTGGAGTCCGTTCAT3' | 5687 | 72℃ 1 min, 35 cycles, 72℃, 10 min | ||
| Ul141b1 | 上游 | 5'CTGTTCTGGGTGCTGTTGAG3' | 5395 | 1055 | 95℃ 4 min, 94℃ 45 s, 52℃ 1 min |
| 下游 | 5'CAACGGATGACACAAAGTATC3' | 6450 | 72℃ 1 min, 35 cycles, 72℃ 10 min | ||
| UL141a2 | 上游 | 5'CGTTCCTTCTTAAACATCC3' | 5070 | 742 | 95℃ 4 min, 94℃ 45 s, 49℃ 1 min |
| 下游 | 5'GCGTGAGAATTACGAAGC3' | 5842 | 72℃ 1 min, 35 cycles, 72℃ 10 min | ||
| Ul141b2 | 上游 | 5'TCGGCTGATGAACGGACT3' | 5661 | 874 | 95℃ 4 min, 94℃ 45 s, 50℃ 1 min |
| 下游 | 5'CCAAGTGGTAACGATAGGAT3' | 6535 | 72℃ 1 min, 35 cycles, 72℃ 10 min |
对23株来自黄疸、先天性巨结肠患儿的低传代临床分离株进行HCMV UL141全序列PCR扩增, 15株阳性(图1), 其中黄疸11株, 巨结肠4株. 总阳性率65.21%. 15株阳性临床分离株均完成HCMV UL141基因全序列测定, HCMV UL141 ORF序列均已被GenBank收录, 序列号为AY496548, AY496550-AY496555, AY600459-AY600461, AY600464-AY600468.
应用BioEdi软件对Toledo株及15株临床分离株进行UL141基因核苷酸序列线形化比较, 结果表明所有低传代临床分离株UL141基因在Toledo株UL141序列的核苷酸位点227均有核苷酸T的缺失, 产生两个新的ORF, 分别命名为UL141A、UL141B. Toledo株UL141 ORF全长1278 bp, 所有临床分离株UL141A位于Toledo株UL141核甘酸序列第1-316位, 全长309 bp或315 bp, UL141B位于Toledo株UL141核甘酸序列第262至1278位, 全长1017 bp.
因所有临床分离株UL141A在Toledo株227核苷酸位点均缺失碱基T, 造成移码突变, 使预测编码蛋白质第76位以后的氨基酸产生大量变异(图2). 其中, 来自黄疸分离株7个和先天性巨结肠分离株2个, 在Toledo株第78-83核苷酸位点有6个碱基的缺失, 使预测编码蛋白质第26位氨基酸产生突变, 由天冬氨酸突变为谷氨酸, 而27、28位的甘氨酸和谷氨酸缺失. 其他变异均为核苷酸的替换. 临床分离株UL141A核苷酸和氨基酸与Toledo株相应区域比较, 同源性分别为95.9%-98.7%和70.2%-74.0%. 临床分离株UL141B核苷酸和氨基酸与Toledo株相应序列比较, 均高度保守, 同源性分别为97.6%-98.5%, 98.5%-99.4%. 核苷酸变异均为碱基替换, 多分布在ORF的中部和3'端.
应用Genedoc软件及Prosite数据库预测编码产物的翻译后修饰位点, UL141A包括硫酸化位点(SUL)、蛋白激酶C磷酸化位点(PKC)、酪蛋白激酶Ⅱ磷酸化位点(CKP)、N相连豆寇酰化位点(MYR)和酰胺化位点(AMID). 与Toledo株比较, 临床分离株UL141A在第75位氨基酸后的修饰位点产生大量变异(图2). 与Toledo株相应区域比较, 除分离株10j第138-144位MYR位点变为CK2位点外, 其余分离株UL141B预测蛋白质的翻译后修饰位点均高度保守.
Toledo株UL141基因预测编码蛋白质含有425个氨基酸, 临床分离株UL141A预测编码蛋白质含有102或104个氨基酸, UL141B预测编码蛋白质含有338个氨基酸. 编码蛋白质等电点预测值分别为, Toledo株8.95, 临床分离株UL141A 10.36-11.54, UL141B 7.99-8.18, 均为碱性蛋白质.
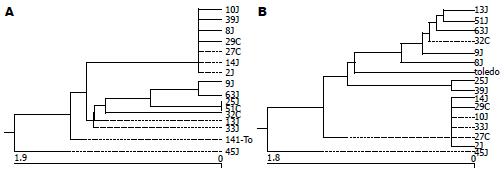
应用DNAStar软件包中的MegAlign软件进行UL141A和UL141B的进化树分析(图3). 未发现来自黄疸和先天性巨结肠患儿的HCMV临床分离株在UL141A和UL141B基因进化树中的分布具有明显规律.
HCMV在体内具有多种组织趋向性, 可以感染多种器官和组织, 产生不同的临床表现. 先天和围生期感染会造成较严重后果. HCMV侵犯肝细胞或肝内外胆管系统, 可破坏肝细胞或胆管上皮细胞, 造成直接和间接胆红素增高[2-8]. 另有研究表明, HCMV先天感染侵犯肠壁组织, 使其发育不良, 造成先天性巨结肠.
HCMV感染的不同临床表现, 一方面可能与宿主的免疫状态有关, 另一方面可能与不同临床分离株基因及其编码产物多态性有关. Cha et al[6]发现Toledo株大约有19个ORF在实验室株AD169中不存在, 却广泛存在于低传代的临床分离株中. 这些基因的编码产物可能与HCMV的致病性有关. UL144基因编码一种Ⅰ型跨膜糖蛋白, 是TNFR超家族成员之一, 很可能具有免疫调节功能, 并可能与HCMV的免疫逃避机制有关[9]. 不同研究人员对UL144的基因分型基本一致. 并证明UL144不同基因型可能与HCMV先天感染的类型和预后有关[10-12].
研究表明UL141基因能够下调CD155分子的表达, 从而逃避NK细胞的杀伤作用[13-14]. 因此UL141基因可能在HCMV致病过程中发挥重要作用. 我们主要探讨UL141基因在来自黄疸和先天性巨结肠患儿的临床低传代分离株中序列变化, 并分析些变化与临床致病性的关系. 对Toledo株及15株来自黄疸和先天性巨结肠患儿的临床分离株进行核苷酸线形化比较分析, 结果表明, 所有临床分离株在Toledo株UL141的核苷酸位点227均有碱基T的缺失, 造成移码突变, 产生两个新的ORF(UL141A、UL141B), 预测编码两种蛋白质. 国外的相关研究表明, 在HCMV其他分离中也存在227位碱基T的缺失, 并且预测了与UL141B 一致的ORF[14]. 那么, 这两个新的ORF哪个具有真正编码蛋白质的能力, 尚需要实验证实.
在临床分离株的UL141A中, 由于移码突变, 使预测编码蛋白质第76位以后的氨基酸几乎全部发生突变. 临床分离株与Toledo株进行线性化比较, 核苷酸水平虽高度保守, 氨基酸水平却发生较大变异. 预测编码蛋白质第75位氨基酸以后的翻译后修饰位点也产生大量变异. 但这些变异在黄疸和先天性巨结肠分离株中均存在, 并且未发现与疾病相关的特异性变异, 表明这种变异不会影响HCMV在肝胆系统和肠壁组织之间的组织趋向性.
15株来自黄疸和先天性巨结肠患儿的临床分离株UL141B无论在核苷酸水平或氨基酸水平, 与Toledo株相应片断比较均高度保守. 这一保守性是在受到生物进化选择压力下被保留下来的, 说明UL141B可能在HCMV生存、复制过程中具有重要意义.
对UL141A和UL141B的核苷酸序列进行进化树分析, 发现两个基因均未能按照疾病类型分组, 也未发现黄疸和先天性巨结肠分离株在进化树中分布的明显规律, 表明UL141A和UL141B基因可能不是决定HCMV感染导致黄疸或先天性巨结肠的关键基因.
HCMV感染具有复杂的致病机制, 基因的多态性很可能是造成HCMV感染致病性差异的重要原因. Toledo株中19个ORF的发现为揭示HCMV的致病机制提供了一条重要线索. 在本研究中, 发现来自黄疸和先天性巨结肠患儿的HCMV低传代病毒分离株UL141片段与Toledo株比较发生变异, 产生了两个新的ORF. HCMV可能具有更广泛的基因多态性. 因此, 全面了解HCMV基因组中每一个基因的结构和功能, 以及他们之间的联系, 是探讨HCMV致病机制的重要途径.
人巨细胞病毒(HCMV)是导致小儿先天畸形和出生缺陷的主要病原体. HCMV感染的不同临床表现可能与宿主免疫状态有关, 也可能与不同临床分离株基因及其编码产物的多态性有关.
管冬元, 副教授, 上海中医药大学基础医学院实验中医学教研室
HCMV UL/b'区19个基因可能在病毒潜伏、复制、逃避机体免疫和不同组织细胞嗜性等方面起重要作用. 这一区域的基因结构特点和编码产物的功能是目前HCMV 致病机制研究的一个热点.
本文首次报道UL141基因的多态性及其与HCMV先天感染致病性的关系. 且研究中的标本均来自先天和围产期感染的分离株.
本研究获得了大量的HCMV Ul141基因序列和结构特点, 为HCMV的致病机制研究提供了重要的基础资料.
本研究深入、全面, 方法合理, 结果可信, 具有较强的创新性.
编辑: 李军亮 电编:何基才
| 1. | Rahbar A, Boström L, Söderberg-Naucler C. Detection of cytotoxic CD13-specific autoantibodies in sera from patients with ulcerative colitis and Crohn's disease. J Autoimmun. 2006;26:155-164. [PubMed] [DOI] |
| 3. | Chou SW, Dennison KM. Analysis of interstrain variation in cytomegalovirus glycoprotein B sequences encoding neutralization-related epitopes. J Infect Dis. 1991;163:1229-1234. [PubMed] |
| 4. | Fries BC, Chou S, Boeckh M, Torok-Storb B. Frequency distribution of cytomegalovirus envelope glycoprotein genotypes in bone marrow transplant recipients. J Infect Dis. 1994;169:769-774. [PubMed] |
| 5. | Torok-Storb B, Boeckh M, Hoy C, Leisenring W, Myerson D, Gooley T. Association of specific cytomegalovirus genotypes with death from myelosuppression after marrow transplantation. Blood. 1997;90:2097-2102. [PubMed] |
| 6. | Cha TA, Tom E, Kemble GW, Duke GM, Mocarski ES, Spaete RR. Human cytomegalovirus clinical isolates carry at least 19 genes not found in laboratory strains. J Virol. 1996;70:78-83. [PubMed] |
| 9. | Benedict CA, Butrovich KD, Lurain NS, Corbeil J, Rooney I, Schneider P, Tschopp J, Ware CF. Cutting edge: a novel viral TNF receptor superfamily member in virulent strains of human cytomegalovirus. J Immunol. 1999;162:6967-6970. [PubMed] |
| 10. | Lurain NS, Kapell KS, Huang DD, Short JA, Paintsil J, Winkfield E, Benedict CA, Ware CF, Bremer JW. Human cytomegalovirus UL144 open reading frame: sequence hypervariability in low-passage clinical isolates. J Virol. 1999;73:10040-10050. [PubMed] |
| 11. | He R, Ruan Q, Xia C, Liu LQ, Lu SM, Lu Y, Qi Y, Ma YP, Liu Q, Ji YH. Sequence variability of human cytomegalovirus UL144 open reading frame in low-passage clinical isolates. Chin Med Sci J. 2004;19:293-297. [PubMed] |
| 12. | Mao ZQ, Huang Y, Sun M, Ruan Q, Qi Y, He R, Huang YJ, Ma YP, Ji YH, Sun ZR. Genetic polymorphism of UL144 open reading frame of human cytomegalovirus DNA detected in colon samples from infants with Hirschsprung's disease. World J Gastroenterol. 2007;13:4350-4354. [PubMed] |
| 13. | Tomasec P, Wang EC, Davison AJ, Vojtesek B, Armstrong M, Griffin C, McSharry BP, Morris RJ, Llewellyn-Lacey S, Rickards C. Downregulation of natural killer cell-activating ligand CD155 by human cytomegalovirus UL141. Nat Immunol. 2005;6:181-188. [PubMed] [DOI] |
| 14. | Wilkinson GW, Tomasec P, Stanton RJ, Armstrong M, Prod'homme V, Aicheler R, McSharry BP, Rickards CR, Cochrane D, Llewellyn-Lacey S. Modulation of natural killer cells by human cytomegalovirus. J Clin Virol. 2008;41:206-212. [PubMed] [DOI] |
| 15. | Dolan A, Cunningham C, Hector RD, Hassan-Walker AF, Lee L, Addison C, Dargan DJ, McGeoch DJ, Gatherer D, Emery VC. Genetic content of wild-type human cytomegalovirus. J Gen Virol. 2004;85:1301-1312. [PubMed] [DOI] |