修回日期: 2008-03-03
接受日期: 2008-04-11
在线出版日期: 2008-04-18
目的: 研究下调HDAC1的表达对大肠癌细胞增殖、凋亡和细胞周期的影响.
方法: 设计合成HDAC1的特异性shRNA, 将其插入至pSilencer载体中, 并将重组后的pSilencer质粒载体经脂质体包裹转染SW480细胞株. 用RT-PCR和Western blot检测shRNA对细胞内HDAC1基因表达的影响, 同时采用Western blot对细胞周期相关基因进行检测. 采用MTT法检测生长抑制作用. 运用流式细胞术检测细胞的凋亡情况和细胞周期分布.
结果: 成功构建和筛选出HDAC1特异性的shRNA质粒载体, 与空白对照组相比, 转染HDAC1-shRNA的大肠癌细胞HDAC1 mRNA蛋白质表达水平明显下降(30.4%±4.5% vs 64.6%±4.4%, P<0.01; 27.4%±4.5% vs 58.1%±3.3%, P<0.01), p21/WAF-1/CIP-1表达增加(97.4%±2.6% vs 62.6%±3.4%, P<0.01), CdK2和Cyclin E蛋白下降(CdK2: 27.7%±6.0% vs 42.6%±4.1%, P<0.01; Cyclin E: 42.0%±8.5% vs 82.8%±3.7%, P<0.01), 细胞生长抑制率增加(24 h: 35.9%±4.9% vs 1.2%±0.6%, P<0.01; 48 h: 47.5%±7.0% vs 1.3%±0.6%, P<0.01; 72 h: 45.7%±6.2% vs 1.0%±0.5%, P<0.01; 96 h: 48.2%±4.7% vs 1.2%±0.7%, P<0.01), 凋亡率明显增加(31.3%±2.8% vs 3.9%±0.7%, P<0.01), G0/G1期和G2/M期细胞比例增加(G0/G1: 64.5%±0.9% vs 57.8%±1.8%, P<0.01; G2/M: 17.4%±1.3% vs 14.5%±0.6%, P<0.05), S期细胞比例相应下降(17.5%±1.0% vs 27.7%±1.5%, P<0.01).
结论: HDAC1特异的shRNA能够有效下调HDAC1基因, 诱导大肠癌细胞发生凋亡和细胞周期阻滞, 从而抑制细胞的增殖.
引文著录: 张孟贤, 韩娜, 于世英. RNA干扰沉默HDAC1基因对大肠癌细胞增殖和凋亡的影响. 世界华人消化杂志 2008; 16(11): 1173-1178
Revised: March 3, 2008
Accepted: April 11, 2008
Published online: April 18, 2008
AIM: To construct the HDAC1 specific recombinant plasmid vector and investigate its effects on the apoptosis, proliferation and cell cycle distribution of colorectal cancer cells.
METHODS: HDAC1 specific short hairpin RNA (shRNA) plasmid vector was constructed and then transfected into the cultured SW480 cell line with lipofectamine 2000. Reverse transcription-polymerase chain reaction (RT-PCR) and Western blot were used to detect the expression of HDAC1 mRNA and protein, respectively, and p21/WAF-1/CIP-1, CdK2, and Cyclin E proteins were detected by Western blot, too. The growth inhibition of SW480 cells was evaluated by MTT assay. Cell apoptosis and cell cycle distribution were determined by flow cytometry.
RESULTS: The levels of HDAC1 mRNA and protein in HDAC1-shRNA group were significantly lower than those in negative control group (30.4% ± 4.5% vs 64.6% ± 4.4%, P < 0.01; 27.4% ± 4.5% vs 58.1% ± 3.3%; both P < 0.01). In comparison with negative control group, HDAC1 silence led to a significant increase of p21/WAF-1/CIP-1 protein (97.4% ± 2.6% vs 62.6% ± 3.4%, P < 0.01) and decrease of CdK2 and Cyclin E protein (27.7% ± 6.0% vs 42.6% ± 4.1%, P < 0.01; 42.0% ± 8.5 % vs 82.8% ± 3.7%, P < 0.01). MTT assay revealed that transfection of HDAC1-shRNA inhibited the growth of SW480 cells, and the inhibitory rates were markedly higher at 24, 48, 72, and 96 h time points than those in negative control group (24 h: 35.9% ± 4.9% vs 1.2% ± 0.6%, P < 0.01; 48 h: 47.5% ± 7.0% vs 1.3% ± 0.6%, P < 0.01; 72 h: 45.7% ± 6.2% vs 1.0% ± 0.5%, P < 0.01; 96 h: 48.2% ± 4.7% vs 1.2% ± 0.7%, P < 0.01). The percentage of apoptosis cells in HDAC1-shRNA group was significantly higher than that in negative control group (31.3% ± 2.8% vs 3.9% ± 0.7%, P < 0.01) and the cells were increased at G0/G1 and G2/M phase (G0/G1: 64.5% ± 0.9% vs 57.8% ± 1.8%, P < 0.01; G2/M: 17.4% ± 1.3% vs 14.5% ± 0.6%, P < 0.05), but decreased at S phase (17.5% ± 1.0% vs 27.7% ± 1.5%, P < 0.01).
CONCLUSION: HDAC1 silence by shRNA can inhibit the growth of colorectal cancer cells through inducing apoptosis and cell cycle arrest.
- Citation: Zhang MX, Han N, Yu SY. Effects of HDAC1 silence by RNA interference on the apoptosis and proliferation of colorectal cancer cells. Shijie Huaren Xiaohua Zazhi 2008; 16(11): 1173-1178
- URL: https://www.wjgnet.com/1009-3079/full/v16/i11/1173.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i11.1173
在真核细胞中, 乙酰化和去乙酰化之间的平衡对基因转录和不同蛋白质的功能发挥重要的作用. 组蛋白乙酰化水平由组蛋白乙酰化酶(histone acetylase, HAT)和组蛋白去乙酰化酶(histone deacetylase, HDAC)共同调节. 根据他们与酵母蛋白的同源性, 将HDAC分为4类, 每类又包括若干HDAC分子[1-2]. 当某些HDAC分子过度表达时, 组蛋白乙酰化水平降低, 染色质结构发生改变, 抑制某些抑癌基因的转录, 导致癌症的发生. 组蛋白去乙酰酶抑制剂(histone deacetylase inhibitor, HDACI)作为新一代的抗肿瘤药物, 在国外已成为研究热点[3-4]. 研究表明, HDACI可通过细胞周期阻滞、诱导分化和凋亡等机制对血液系统肿瘤[5-6]和一些实体肿瘤[7-9]有明显抑制作用. 但目前常用的HDAC抑制剂选择性差, 效能较低, 将会被具有更高选择性的HDAC抑制剂所取代[10]. 许多研究表明, 不同的类型的肿瘤可能具有不同的HDAC靶点[11], 因此明确某一肿瘤的HDAC靶点是发展高选择性HDAC抑制剂的关键. RNA干扰技术为此提供了最好的研究工具. 我们运用RNA干扰技术使人大肠癌细胞SW480中HDAC1基因沉默, 来研究HDAC1对大肠癌细胞凋亡、增殖、细胞周期分布的影响及其可能机制.
pSilencerTM2.0-U6质粒(Ambion公司)含氨苄青霉素Ampr抗性基因和人U6启动子. 质粒在埃希菌DH5α菌株(Gibco)中扩增. T4 DNA连接酶购自Ambion公司; Lipofectamine 2000脂质体为Invitrogen公司产品. DNA片段由上海生工生物工程公司合成. 根据GenBank中报道的HDAC1 cDNA(accession no.NM_004964)完整序列, 参考siRNA的设计策略, 利用Ambion公司提供的在线工具, 设计一对shRNA序列的模板DNA和不针对任何mRNA的模板DNA作为对照, 序列如下: HDAC1-shRNA: 正义链: 5'-GATCCG CTAATGAGCTTCCATACAA TTCAAGAGA TTGTATGGAAGCTCATTAGTT TTTTGGAAA-3'; 反义链: 5'-AGCTTTTCCAAAA AACTAATGAGCTTCCATACAA TCTCTTGAA TTGTATGGAAGCTCATTAG CG-3'. control-shRNA: 正义链: 5'-GATCCG CCGGATGTCGAGAACTTCA TTCAAGAGA TGAAGTTCTCGACATCCGGTT TTTTGGAAA-3'; 反义链: 5'-AGCTTTTCCAAAA AACCGGATGTCGAGAACTTCA TCTCTTGAA TGAAGTTCTCGACATCCGG CG-3'. 以上模板序列包括BamHⅠ酶切位点、19 nt正义序列、9 nt loop接头序列、19 nt反义序列、RNA PolⅢ终止子和HindⅢ酶切位点.
取1 g/L正义和反义shRNA模板DNA各2 μL以及退火缓冲液(DNA annealing solution)46 μL充分混匀, 加热至90℃, 3 min后自然冷却至室温. 每条正义链分别与其反义链退火后, 可形成一个限制性核酸内切酶BamHⅠ及HindⅢ的黏性末端. pSilencer 2.0-U6表达质粒由供应商分别以BamHⅠ及HindⅢ酶切处理, 形成线性化片段, 并除去酶切下来的小片段而纯化, 不会发生自身连接. 按照说明, 应用T4 DNA连接酶将退火后的双链核苷酸片段插入线性化的载体: 退火DNA片段(8 mg/µL)1 µL+PsilencerTM2.0-U6载体1 µL+10×T4 DNA Ligase Buffer 1 µL+T4 DNA Ligase 1 µL+ Nuclease-free water 6 µL充分混匀, 室温16℃水浴反应过夜. 各取连接产物5 µL转化感受态细胞DH5α, 涂布于含Ampr抗性(终浓度100 mg/L)的LB平板上, 37℃恒温培养过夜. 从每个培养皿上各挑取4个单克隆菌落接种于含Ampr抗性(终浓度100 mg/L)的LB培养液3 mL中, 37℃恒温摇床培养过夜. 挑选阳性克隆送上海博亚生物技术有限公司进行DNA测序分析加以确认. 构建的重组质粒命名为pshRNA-HDAC1和pshRNA-control.
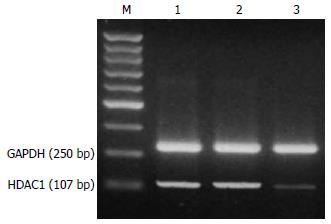
1.2.1 HDAC1 mRNA的表达: 大肠癌细胞SW480购自中国典型培养物保藏中心, 采用含100 mL/L小牛血清的RPMI 1640(Gibco)培养液, 在37℃, 50 mL/L CO2孵育箱中培养. 调整细胞浓度, 接种无菌6孔板, 每孔约1×106个细胞. 待其生长至80%-90%融合时, 按照Lipofectamine 2000脂质体转染试剂盒说明书进行转染操作, 同时设立空白对照孔. 细胞转染48 h后收集细胞, 按TRIzol试剂说明书操作提取细胞总RNA, 用于扩增人HDAC1 cDNA. PCR引物(上海生工生物工程公司合成)序列如下: HDAC1上游序列: 5'-GAATCCGCATGACTCATAAT-3', 下游序列: 5'-GCTGTGGTACTTGGTCATCT-3'. 同时以人GAPDH为内参照, 引物的上游序列: 5'-ACGGATTTGGTCGTATTGGG-3', 下游序列: 5'-TGATTTTGGAGGGATCTCGC-3'. PCR扩增条件如下: 94℃预变性5 min; 94℃变性30 s, 60℃退火30 s, 72℃延伸60 s, 35个循环; 72℃延伸10 min. HDAC1和GAPDH的PCR片段大小为107 bp和250 bp. PCR产物采用图象扫描仪拍照并进行条带灰度分析, 以HDAC1/GAPDH的比值代表其相对含量.
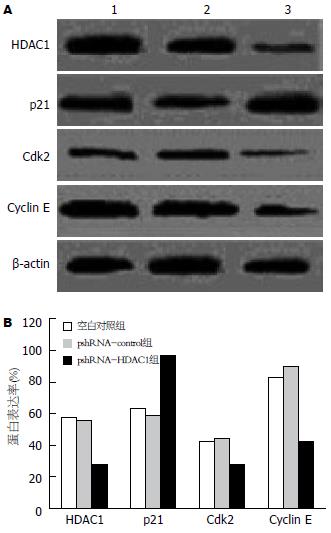
1.2.2 HDAC1、p21、Cdk2及Cyclin E蛋白检测: 细胞转染48 h后收集各孔细胞, 加入细胞裂解液, 提取细胞总蛋白, 用BCA试剂盒(美国Bio-Rad)检测蛋白质浓度, 灌制100 g/L SDS-PAGE 凝胶, 每孔上样50 µg, 经电泳, 转膜, 封闭, 加入HDAC1(Santa Cruz)、P21/WAF-1/CIP-1、Cdk2和Cyclin E(晶美公司)一抗, 工作液浓度为1:500, 加入辣根过氧化物酶标记的二抗(Santa Cruz), 工作液浓度1:1000, 化学发光法(ECL)曝光显影, 冲洗胶片, 用紫外分光光度计扫描条带灰度值反应蛋白质表达水平. β-actin为内参.
1.2.3 MTT法: 收集转染后不同时间段的各组细胞, 调整细胞密度至1×108/L, 每孔100 μL接种于96孔培养板内, 每组各设3个复孔. 每孔加入5 g/L的MTT溶液20 μL, 继续培养4 h, 离心后小心吸弃孔内培养液, 再加200 μL DMS0, 酶标仪测定490 nm波长处吸光度值(A). 以空白对照调零, 计算: 肿瘤细胞生长抑制率(%) = (1-实验组A/对照组A)×100%.
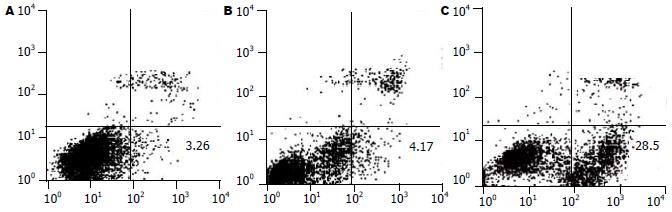
1.2.4 细胞凋亡及细胞周期的检测: 细胞转染48 h后收集各孔细胞, 按AnnexinV-FITC凋亡检测试剂盒(晶美公司)说明书操作, 简要步骤如下: 将细胞悬浮在结合缓冲液中, 调整细胞密度为1×109/L, 取细胞100 μL加入FITC标记Annexin V 5 μL和PI 10 μL混合, 室温避光孵育15 min后FACSsort流式细胞仪检测, CellQuest软件(BD公司)获取并分析数据. 另细胞转染48 h后收集各孔细胞, 经胰酶消化, 制成单细胞悬液, 冰乙醇固定, 加入终浓度为100 mg/L的Rnase A 10 μL, 37℃水浴30 min后移入冰浴中停止酶反应, 再加入终浓度为5 mg/L的PI染色剂10 μL, 4℃避光保存, 孵育过夜, 用流式细胞仪检测细胞周期分布.
统计学处理 采用SPSS10.0软件进行t检验, 数据以mean±SD表示, P<0.05为具有统计学意义.
在空白对照组和pshRNA-control组均可见明显的HDAC1目的DNA条带, 2组间HDAC1 mRNA的表达丰度差异无显著性(64.6%±4.4% vs 58.3%±4.6%, P>0.05). pshRNA-HDAC1转染SW480细胞后, HDAC1基因mRNA水平较空白对照组明显下调(图1), 两组间差异有统计学意义(30.4%±4.5% vs 64.6%±4.4%, P<0.01).
Western blot检测结果显示, HDAC1, p21/WAF-1/CIP-1, Cdk2和Cyclin E蛋白的表达在空白对照组和pshRNA-control组之间差异无统计学意义(58.1%±3.3% vs 55.2%±3.8%, 62.6%±3.4% vs 58.5%±1.7%, 42.6%±4.1% vs 44.2%±1.9%, 82.8%±3.7% vs 89.9%±5.95%, P>0.05, 图2). pshRNA-HDAC1转染SW480细胞后, HDAC1基因表达明显被抑制, p21/WAF-1/CIP-1明显上调, CDK2和Cyclin E蛋白则明显下调. 与空白对照相比差异有统计学意义(27.4%±4.5% vs 58.1%±3.3%, 97.4%±2.6% vs 62.6%±3.4%, 27.7%±6.0% vs 42.6%±4.1%, 42.0%±8.5% vs 82.8%±3.7%, P<0.01).
MTT检测结果显示, pshRNA-HDAC1转染SW480细胞24 h后及就已经出现明显的生长抑制, 48 h后达到抑制高峰, 维持至96 h细胞抑制作用未出现下降趋势(表1). 通过AnnexinV-FITC和PI双重标记后上流式细胞仪检测结果显示, pshRNA-HDAC1转染细胞48 h后出现大量的凋亡细胞, 较空白对照组明显增加, 差异有显著性(31.3%±2.8% vs 3.9%±0.7%, P<0.01), 而pshRNA-control组和空白对照组细胞凋亡率差异无统计学意义(图3).
流式细胞术检测检测结果显示, pshRNA-HDAC1转染SW480细胞48 h后, G0/G1期和G2/M期细胞比例增加, S期细胞比例相应下降, 差异有统计学意义, 而pshRNA-control组和空白对照组细胞的细胞周期分布差异无统计学意义(表2).
染色质核心组蛋白的乙酰化和去乙酰化与基因表达调控密切相关, 调节组蛋白乙酰化状态有两种酶, 即HAT和HDAC. HAT将乙酰辅酶A上的疏水乙酰基转移到组蛋白N端赖氨酸残基, 中和掉一个正电荷, 使DNA与组蛋白之间的静电引力和空间位阻增大, 二者之间的相互作用减弱, 染色质呈转录活性结构, DNA易于解聚、舒展, 有利于转录因子与DNA模板的结合, 激活转录. 相反, HDAC在组蛋白N端赖氨酸的εNH3+上引入乙酰基, 通过组蛋白N端的去乙酰化, 使组蛋白带正电荷, 从而与带负电荷的DNA紧密结合, 染色质呈致密卷曲的阻抑结构, 抑制转录[12]. 组蛋白去乙酰酶作为调控基因的关键蛋白酶, 其功能异常被证实与肿瘤的发生和发展密切相关[13]. 目前发现在哺乳动物中, HDAC家族至少有HDAC 7种, 他们之间具有高度的同源性, 其中HDAC1与癌症的关系最具代表性. HDAC抑制剂对多种肿瘤具有抗瘤活性, 其中包括淋巴瘤、肺癌、肝癌、前列腺癌及乳腺癌等[14-18]. 目前认为, 大肠癌的发生是多因素、多阶段和多种基因改变协同作用的过程. 在这个过程中, 涉及许多癌基因的激活和/或抑癌基因的失活. 而在基因的表达调控中, 表观遗传修饰有着重要的作用, 其中组蛋白乙酰化是修饰的主要方式之一. 研究表明, 多种HDAC抑制剂能够诱导大肠癌细胞凋亡和细胞周期阻滞, 抑制肿瘤细胞的生长[19-20]. 但选择性抑制某种特定的HDAC对大肠癌细胞的影响却少有报道. RNA干扰技术为此提供了最好的研究工具.
RNA干扰是最近发展起来的一种基因调控技术, 能够特异性地降解相应序列的mRNA[21-22], 为哺乳动物基因功能的研究提供了强大的工具, 在功能基因组学研究领域得到越来越多的重视. 许多研究表明shRNA在哺乳动物细胞中可以显著抑制内源性基因和外源性基因表达[23-24]. 本研究设计并构建HDAC1特异的shRNA真核表达质粒载体. 将重组载体转染大肠癌细胞系SW480, 在转染HDAC1 shRNA后分别检测HDAC1 mRNA和蛋白质水平, 发现HDAC1的表达明显受抑. SW480大肠癌细胞转染HDAC1 shRNA 24 h后就已经出现明显的生长抑制, 48 h后达到抑制高峰, 维持至96 h细胞抑制作用未出现下降趋势. 本文主要从细胞凋亡和细胞周期阻滞角度对其作用机制方面进行探讨. 流式细胞仪检测结果显示HDAC1转染48 h后, SW480细胞发生G0/G1和G2/M期阻滞, 其中以G0/G1期阻滞更为明显. 真核细胞的细胞周期行进受细胞周期依赖性蛋白激酶亚单位和周期素调节亚单位调控, 时相性激活是细胞周期调控机制的核心, 在细胞周期行进的不同时期, 由不同Cyclin蛋白的表达驱动[25]. Cdk2与Cyclin E结合是S期启动的必要条件. Cyclin E/Cdk2复合体使Rb磷酸化, 释放核转录因子E2F, 激活Cyclin转录, 加速细胞周期跨过G1/S检查点, 进入S期. p21/WAF-1/CIP-1作为细胞增殖的重要调节因子, 是细胞周期内通用性抑制物, 他能与多种CDK/cyclin复合物直接结合, 抑制CDK的激活, 阻滞G1/S的过渡. 现认为p21/CIP1/WAF1在肿瘤细胞中的沉默机制可能由其染色质的表观遗传修饰所致, 其表达可被组蛋白的乙酰化水平所调节[26-29].
为进一步探讨HDAC1基因沉默引起G0/G1期细胞周期阻滞的机制, 我们检测了可能涉及细胞周期调控的几个关键基因的表达. 结果发现, SW480大肠癌细胞转染HDAC1 shRNA 48 h后HDAC1基因的mRNA和蛋白质水平的下降与p21/WAF-1/CIP-1蛋白质水平的升高相一致. 相反, Cdk2与Cyclin E蛋白表达水平上调. 结合以上分析结果, 认为HDAC1 shRNA可能通过上调p21/WAF-1/CIP-1的表达抑制Cdk2与Cyclin E的表达达到阻止细胞周期进程, 影响细胞增殖的目的. 细胞凋亡是由内在基因精确调节的一种程序性死亡, 他同坏死有着本质的区别[30]. 细胞发生凋亡时, 细胞膜的改变之一是磷脂酰丝氨酸从细胞膜的内侧转移到细胞膜的外侧, Annexin V可以特异性结合于细胞膜表面的磷脂酰丝氨酸. PI为膜非通透性核酸嵌入型染料, 不能穿过完整的细胞膜, 仅穿过死细胞膜, 从而使之染色, AnnexinV、PI双标可以区分凋亡和坏死的细胞. 我们采用AnnexinV、PI双标经流式细胞术检测发现, SW480大肠癌细胞转染HDAC1 shRNA 48 h后出现大量凋亡细胞(AnnexinV+PI-). 说明HDAC1基因沉默可能通过细胞凋亡而非直接的细胞毒作用产生抗肿瘤活性.
我们通过转染HDAC1 shRNA体外作用于大肠癌细胞SW480, 初步发现HDAC1基因沉默可以阻滞大肠癌细胞SW480于细胞周期的G0/G1期, 诱导细胞凋亡的发生, 从而抑制细胞的增殖, 为开发HDAC1高选择性HDAC抑制剂应用于大肠癌治疗提供理论依据.
肿瘤的发生是多因素、多阶段和多种基因改变协同作用的过程. 在这个过程中, 涉及许多癌基因的激活和/或抑癌基因的失活. 而在基因的表达调控中, 表观遗传修饰有着重要的作用, 其中组蛋白乙酰化是修饰的主要方式之一. 当某些组蛋白去乙酰化酶分子过度表达时, 组蛋白乙酰化水平降低, 染色质结构发生改变, 抑制某些抑癌基因的转录, 可导致癌症的发生. 因此组蛋白去乙酰化酶已经成为人们抗肿瘤治疗的一个新靶点.
王健生, 副教授, 西安交通大学医学院第一附属医院肿瘤外科
组蛋白去乙酰酶抑制剂作为新一代的抗肿瘤药物, 在国外已成为研究热点.
Archer et al研究表明HDAC抑制剂丁酸钠能够以p21/CIP1/WAF1依赖方式抑制HT-29细胞Cyclin B1的表达水平, 从而导致肿瘤细胞的生长抑制.
本文通过设计合成HDAC1的特异性shRNA, 将其插入至pSilencer载体中, 并将重组后的pSilencer质粒载体经脂质体包裹转染SW480细胞株, 特异性抑制HDAC1基因表达, 研究HDAC1对大肠癌细胞凋亡、增殖、细胞周期分布的影响及其可能机制.
shRNA介导的HDAC1基因沉默能够阻滞大肠癌细胞SW480于细胞周期的G0/G1期, 诱导细胞凋亡的发生, 从而显著抑制细胞的增殖, 为开发HDAC1高选择性HDAC抑制剂应用于大肠癌治疗提供理论依据.
本研究创新性较强, 实验设计合理, 具有一定的科学意义.
编辑: 李军亮 电编:郭海丽
| 2. | Gallinari P, Di Marco S, Jones P, Pallaoro M, Steinkuhler C. HDACs, histone deacetylation and gene transcription: from molecular biology to cancer therapeutics. Cell Res. 2007;17:195-211. [PubMed] |
| 3. | Bi G, Jiang G. The molecular mechanism of HDAC inhibitors in anticancer effects. Cell Mol Immunol. 2006;3:285-290. [PubMed] |
| 4. | Mei S, Ho AD, Mahlknecht U. Role of histone deacetylase inhibitors in the treatment of cancer (Review). Int J Oncol. 2004;25:1509-1519. [PubMed] |
| 5. | Kuendgen A, Gattermann N. Valproic acid for the treatment of myeloid malignancies. Cancer. 2007;110:943-954. [PubMed] |
| 6. | Bug G, Schwarz K, Schoch C, Kampfmann M, Henschler R, Hoelzer D, Ottmann OG, Ruthardt M. Effect of histone deacetylase inhibitor valproic acid on progenitor cells of acute myeloid leukemia. Haematologica. 2007;92:542-545. [PubMed] |
| 7. | Ahn MY, Jung JH, Na YJ, Kim HS. A natural histone deacetylase inhibitor, Psammaplin A, induces cell cycle arrest and apoptosis in human endometrial cancer cells. Gynecol Oncol. 2008;108:27-33. [PubMed] |
| 8. | Mukhopadhyay NK, Weisberg E, Gilchrist D, Bueno R, Sugarbaker DJ, Jaklitsch MT. Effectiveness of trichostatin A as a potential candidate for anticancer therapy in non-small-cell lung cancer. Ann Thorac Surg. 2006;81:1034-1042. [PubMed] |
| 9. | Bluethner T, Niederhagen M, Caca K, Serr F, Witzigmann H, Moebius C, Mossner J, Wiedmann M. Inhibition of histone deacetylase for the treatment of biliary tract cancer: a new effective pharmacological approach. World J Gastroenterol. 2007;13:4761-4770. [PubMed] |
| 10. | Karagiannis TC, El-Osta A. Will broad-spectrum histone deacetylase inhibitors be superseded by more specific compounds? Leukemia. 2007;21:61-65. [PubMed] |
| 11. | Nakagawa M, Oda Y, Eguchi T, Aishima S, Yao T, Hosoi F, Basaki Y, Ono M, Kuwano M, Tanaka M. Expression profile of class I histone deacetylases in human cancer tissues. Oncol Rep. 2007;18:769-774. [PubMed] |
| 12. | Roth SY, Denu JM, Allis CD. Histone acetyltran-sferases. Annu Rev Biochem. 2001;70:81-120. [PubMed] |
| 13. | Mahlknecht U, Hoelzer D. Histone acetylation modifiers in the pathogenesis of malignant disease. Mol Med. 2000;6:623-644. [PubMed] |
| 14. | Duvic M, Vu J. Vorinostat: a new oral histone dea-cetylase inhibitor approved for cutaneous T-cell lymphoma. Expert Opin Investig Drugs. 2007;16:1111-1120. [PubMed] |
| 15. | Komatsu N, Kawamata N, Takeuchi S, Yin D, Chien W, Miller CW, Koeffler HP. SAHA, a HDAC inhibitor, has profound anti-growth activity against non-small cell lung cancer cells. Oncol Rep. 2006;15:187-191. [PubMed] |
| 16. | Coradini D, Speranza A. Histone deacetylase inhibitors for treatment of hepatocellular carcinoma. Acta Pharmacol Sin. 2005;26:1025-1033. [PubMed] |
| 17. | Kulp SK, Chen CS, Wang DS, Chen CY, Chen CS. Antitumor effects of a novel phenylbutyrate-based histone deacetylase inhibitor, (S)-HDAC-42, in prostate cancer. Clin Cancer Res. 2006;12:5199-5206. [PubMed] |
| 18. | Bali P, Pranpat M, Swaby R, Fiskus W, Yamaguchi H, Balasis M, Rocha K, Wang HG, Richon V, Bhalla K. Activity of suberoylanilide hydroxamic Acid against human breast cancer cells with amplification of her-2. Clin Cancer Res. 2005;11:6382-6389. [PubMed] |
| 19. | Archer SY, Johnson J, Kim HJ, Ma Q, Mou H, Daesety V, Meng S, Hodin RA. The histone deacetylase inhibitor butyrate downregulates cyclin B1 gene expression via a p21/WAF-1-dependent mechanism in human colon cancer cells. Am J Physiol Gastrointest Liver Physiol. 2005;289:G696-G703. [PubMed] |
| 20. | Calonghi N, Cappadone C, Pagnotta E, Boga C, Bertucci C, Fiori J, Tasco G, Casadio R, Masotti L. Histone deacetylase 1: a target of 9-hydroxystearic acid in the inhibition of cell growth in human colon cancer. J Lipid Res. 2005;46:1596-1603. [PubMed] |
| 21. | Gitlin L, Karelsky S, Andino R. Short interfering RNA confers intracellular antiviral immunity in human cells. Nature. 2002;418:430-434. [PubMed] |
| 22. | Jacque JM, Triques K, Stevenson M. Modulation of HIV-1 replication by RNA interference. Nature. 2002;418:435-438. [PubMed] |
| 23. | Brummelkamp TR, Bernards R, Agami R. A system for stable expression of short interfering RNAs in mammalian cells. Science. 2002;296:550-553. [PubMed] |
| 24. | Elbashir SM, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. Nature. 2001;411:494-498. [PubMed] |
| 26. | Chen YX, Fang JY, Zhu HY, Lu R, Cheng ZH, Qiu DK. Histone acetylation regulates p21WAF1 expression in human colon cancer cell lines. World J Gastroenterol. 2004;10:2643-2646. [PubMed] |
| 27. | Fang JY, Chen YX, Lu J, Lu R, Yang L, Zhu HY, Gu WQ, Lu LG. Epigenetic modification regulates both expression of tumor-associated genes and cell cycle progressing in human colon cancer cell lines: Colo-320 and SW1116. Cell Res. 2004;14:217-226. [PubMed] |
| 28. | Ocker M, Schneider-Stock R. Histone deacetylase inhibitors: signalling towards p21cip1/waf1. Int J Biochem Cell Biol. 2007;39:1367-1374. [PubMed] |
| 29. | Zopf S, Neureiter D, Bouralexis S, Abt T, Glaser KB, Okamoto K, Ganslmayer M, Hahn EG, Herold C, Ocker M. Differential response of p53 and p21 on HDAC inhibitor-mediated apoptosis in HCT116 colon cancer cells in vitro and in vivo. Int J Oncol. 2007;31:1391-1402. [PubMed] |
| 30. | Dini L, Coppola S, Ruzittu MT, Ghibelli L. Multiple pathways for apoptotic nuclear fragmentation. Exp Cell Res. 1996;223:340-347. [PubMed] |