修回日期: 2008-02-28
接受日期: 2008-03-28
在线出版日期: 2008-04-08
目的: 检测错配修复基因hMLH1、hMSH2和hMLH3在胰腺癌中的甲基化和表达状态, 探讨错配修复缺陷在胰腺癌发病中的作用.
方法: 采用甲基化特异性PCR(MSP)检测hMLH1、hMSH2和hMLH3的甲基化状态, 逆转录PCR(RT-PCR)检测hMLH1、hMSH2和hMLH3的表达状态.
结果: 在胰腺癌组织中hMLH1、hMSH2和hMLH3的甲基化频率分别为28.6%、46.4%和39.3%; 在癌旁正常组织中分别为3.6%、10.7%和12.5%, 上述各基因在胰腺癌中的甲基化频率均显著高于癌旁正常组织(hMLH1: χ2 = 12.97, P<0.01; hMSH2: χ2 = 17.50, P<0.01; hMLH3: χ2 = 10.47, P<0.01).在胰腺癌组织中分别有25.0%、50.0%和33.9%没有检出hMLH1、hMSH2和hMLH3表达. 在癌旁正常组织中分别有7.1%、8.9%和16.1%, 各基因在胰腺癌中的表达缺失频率均显著高于癌旁正常组织(hMLH1: χ2 = 6.62, P<0.05; hMSH2: χ2 = 22.73, P<0.01; hMLH3: χ2 = 4.76, P<0.05).hMLH1在胰腺癌细胞系PANC-1和CFPAC-1检出甲基化和表达消失. hMSH2在PC-3检出甲基化和表达消失. hMLH3在PC-3和PANC-1检出甲基化和表达亦消失.
结论: 在胰腺癌错配修复基因缺陷较普遍参与了部分胰腺癌的发病过程.
引文著录: 卜献民, 赵成海, 张宁, 林帅, 高峰, 戴显伟. 胰腺癌组织和细胞系中DNA错配修复基因的甲基化和异常表达. 世界华人消化杂志 2008; 16(10): 1074-1077
Revised: February 28, 2008
Accepted: March 28, 2008
Published online: April 8, 2008
AIM: To determine the methylation and expression status of mismatch repair genes hMLH1, hMSH2 and hMLH3 and explore the role of mismatch repair defect in pancreatic cancer.
METHODS: Methylation status of hMLH1, hMSH2 and hMLH3 was detected by methylation-specific polymerase chain reaction (MSP), and the expression of hMLH1, hMSH2 and hMLH3 were determined by reverse-transcription PCR (RT-PCR).
RESULTS: The frequencies of methylation for hMLH1, hMSH2 and hMSH3 were 28.6%, 46.4% and 39.3%, respectively in pancreatic cancer tissues, and 3.6%, 10.7% and 12.5% in cancer-adjacent normal tissues, and there were significant differences between cancer and normal tissues (hMLH1: χ2 = 12.97, P < 0.01; hMSH2: χ2 = 17.50, P < 0.01; hMLH3: χ2 = 10.47, P < 0.01). The frequencies of expression loss for hMLH1, hMSH2 and hMSH3 were 25.0%, 50.0% and 33.9%, respectively in pancreatic cancer tissues, and 7.1%, 8.9% and 16.1% in cancer-adjacent normal tissues, and there were also marked differences between cancer and normal tissues (hMLH1: χ2 = 6.62, P < 0.05; hMSH2: χ2 = 22.73, P < 0.01; hMLH3: χ2 = 4.76, P < 0.05). Methylations of hMLH1, hMSH2 and hMLH3 were found in PANC-1, PC-3 and CFPAC-1 cell lines, while expression loss of hMLH1 was observed in PANC-1 and CFPAC-1 cell lines, that of hMSH2 in PC-3 cell line and hMLH3 was observed in PC-3 and PANC-1 cell lines.
CONCLUSION: Mismatch repair defect is common in pancreatic cancer and plays a role in pancreatic carcinogenesis.
- Citation: Bu XM, Zhao CH, Zhang N, Lin S, Gao F, Dai XW. Hypermethylation and aberrant expression of DNA mismatch repair gene in pancreatic cancer. Shijie Huaren Xiaohua Zazhi 2008; 16(10): 1074-1077
- URL: https://www.wjgnet.com/1009-3079/full/v16/i10/1074.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i10.1074
DNA错配修复基因是一类重要的看门基因(caretaker gene), 能够识别并及时纠正DNA复制过程中错配的碱基对, 以保证复制的正确性和基因的稳定性. 目前已发现的错配修复基因包括MutS家族的hMSH2及MutL家族的hMLH1和hMLH3等. 这些基因在DNA聚合酶的协助下, 可将错配的碱基切除并修复. DNA错配修复缺陷常导致DNA复制过程中出现的错误无法得到纠正, 使整个基因组变得不稳定而出现微卫星不稳定(microsatellite instability, MSI)现象, 最终诱导肿瘤发生.
胰腺癌是一种恶性程度高、预后差的消化系统肿瘤. 越来越多的研究证明, 胰腺癌的发病是一个多阶段、多基因参与的过程. Kondo et al[1]和Yamamoto et al[2]均在研究中发现部分胰腺癌患者存在MSI, 提示错配修复缺陷参与了这些肿瘤的发病过程. 我们对hMLH1、hMSH2和hMLH3等多个错配修复基因在胰腺癌中的表达情况及甲基化状态进行研究, 以探讨错配修复缺陷在胰腺癌发病中的作用.
中国医科大学附属盛京医院胰腺癌及相应的癌旁正常组织标本各56例. 标本获取后即冷冻于液氮之中, 并保存在-80℃条件下备用. 采用HE染色确定肿瘤标本主要由肿瘤组织构成, 癌旁正常组织没有肿瘤细胞浸润及正常胰腺组织没有炎性细胞浸润. 人类胰腺癌细胞系PC-3、PANC-1和CFPAC-1购自于南京凯基(KeyGen)生物科技发展公司. 人类胰腺癌细胞系PC-3、PANC-1和CFPAC-1细胞培养于RPMI 1640培养基中, 其中含有100 mL/L胎牛血清, 青霉素(100 kU/L)及链霉素(100 mg/L) , 于37℃下置于含有50 mL/L CO2的培养箱中.
采用饱和氯化钠法进行DNA提取. 采用RNAiso试剂(TaKaRa公司)进行RNA提取, 具体步骤参照其说明书进行. 逆转录PCR反应(RT-PCR): 采用随机9引物和AMV逆转录酶进行逆转录反应. 反应体系共10 μL, 逆转录反应条件: 30℃, 10 min; 42℃, 25 min; 99℃, 5 min; 5℃, 5 min, 共1个循环. PCR总反应体系40 μL, 反应条件为: 94℃, 2 min, 1个循环. 94℃, 30 s; 60℃, 30 s; 72℃, 2 min, 共30个循环. 各基因引物序列参见文献[3]. 甲基化特异性PCR反应(MSP): 采用genmed基因甲基化检测试剂盒进行hMLH1, hMSH2和hMSH3基因甲基化状态检测. 具体步骤包括转化实验和甲基化特异性PCR检测. PCR反应条件为: 95℃, 5 min, 1个循环. 95℃, 30 s; 60℃, 30 s; 72℃, 30 s, 共35个循环. 甲基化检测包括检测甲基化反应(M)和未甲基化反应(U)两方面. 各基因引物序列参见文献[2].
统计学处理 肿瘤组织与瘤旁正常组织之间比较采用χ2检验, P<0.05为有统计学意义.
在56例胰腺癌组织标本中, 有16例(28.6%)检出hMLH1甲基化, 26例(46.4%)检出hMSH2甲基化, 22例(39.3%)检出hMLH3甲基化. 在其相应的癌旁组织中, 分别有2例(3.6%)、6例(10.7%)和7例(12.5%)检出hMLH1、hMSH2和hMLH3甲基化. 上述各基因在肿瘤标本中的甲基化频率均显著高于癌旁正常组织(hMLH1: χ2 = 12.97, P<0.01; hMSH2: χ2 = 17.50, P<0.01; hMLH3: χ2 = 10.47, P<0.01). 在胰腺癌组织标本中, 分别有14例(25.0%)、28例(50.0%)和19例(33.9%)没有检出hMLH1、hMSH2和hMLH3表达. 在其相应的癌旁正常组织标本中分别有4例(7.1%)、5例(8.9%)和9例(16.1%)没有检出hMLH1、hMSH2和hMLH3表达. 上述各基因在肿瘤标本中的表达缺失频率均显著高于癌旁正常组织(hMLH1: χ2 = 6.62, P<0.05; hMSH2: χ2 = 22.73, P<0.01; hMLH3: χ2 = 4.76, P<0.05).
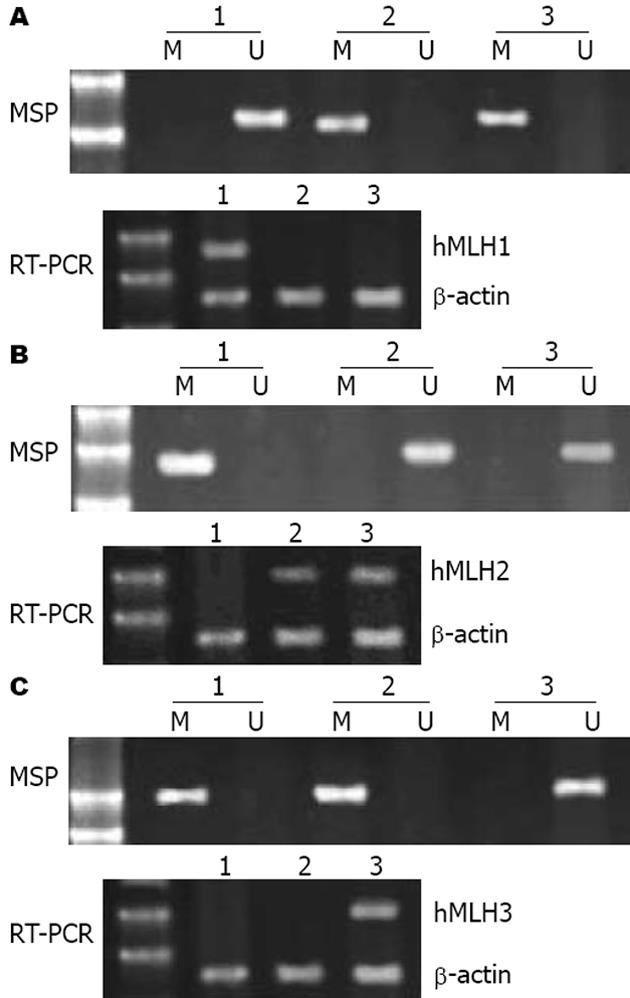
hMLH1在PANC-1和CFPAC-1检出甲基化, 表达消失, 在PC-3中未检出甲基化, 同时检出表达(图1A). hMSH2在PC-3检出甲基化和表达消失, 而在PANC-1和CFPAC-1未检出甲基化, 同时检出表达(图1B). hMLH3在PC-3和PANC-1检出甲基化和表达亦消失, 在CFPAC-1中未检出甲基化, 同时检出表达(图1C).
胰腺癌的发生是一个复杂的过程, 其中涉及到一系列遗传和表观遗传(epigenetic)的改变, 导致细胞失控性生长. 基因突变在胰腺癌中比较常见, 其常通过激活癌基因或沉默抑癌基因而诱导肿瘤的发生发展. 除了基因突变以外, 表观遗传改变, 例如异常甲基化, 是导致基因表达沉默的另一种重要途径. 现在越来越多的基因在胰腺癌中被发现存在启动子甲基化及其所致的表达沉默[4-10]. 这些基因在胰腺癌中甲基化频率往往明显高于癌旁正常组织和胰腺良性肿瘤, 提示基因甲基化不是胰腺细胞恶性转变的结果, 而相反可能是细胞出现恶性转变的原因.
DNA错配修复基因能够识别并及时纠正DNA复制过程中错配的碱基对, 以保证复制的正确性和基因的稳定性. 目前已发现的错配修复基因包括MutS家族的hMSH2、hMSH3及hMSH6和MutL家族的hMLH1、hPMS1、hPMS2和hMLH3. 这些基因通过互相配合能够识别并结合错配的DNA序列, 在DNA聚合酶的协助下, 将错配的碱基切除并修复. 由基因突变或过甲基化引起的DNA错配修复缺陷常导致DNA复制过程中出现的错误无法得到纠正而不断积累, 使整个基因组变得不稳定, 一些癌基因和抑癌基因突变频率不断增加, 最终诱导肿瘤发生. 目前已发现错配修复缺陷参与了人类多种肿瘤的发病过程, 包括结直肠癌[11-12]、胃癌[13-14]、乳腺癌[15-16]、子宫内膜癌[17-18]及卵巢癌[19-20]. 一些研究显示, hMLH1启动子过甲基化及其导致的表达减少或消失存在于15%-30%的胃癌患者中, 提示错配修复基因启动子过甲基化及其导致的失活与胃癌等肿瘤的发病有关. 由于在错配修复基因存在缺陷的情况下, DNA聚合酶复制错误引起的DNA序列微卫星长度异常变化和基因组的不稳定性常得不到修复, 因此错配修复缺陷参与的肿瘤通常具有MSI这一表型特征. Yamamoto et al在研究中发现MSI阳性与hMLH1启动子甲基化相关. 但Kondo et al对7例高微卫星不稳定胰腺癌的研究却显示hMLH1表达正常, 并不存在hMLH1甲基化现象. 这些研究提示胰腺癌MSI阳性可能与其他的错配修复缺陷参与有关, 或者是多个错配修复基因缺陷综合作用的结果.
我们的研究发现28.6%胰腺癌组织标本检出hMLH1甲基化, 46.4%胰腺癌组织标本检出hMSH2甲基化, 39.3%胰腺癌组织标本检出hMLH3甲基化. 上述各基因在肿瘤标本中的甲基化频率均显著高于癌旁正常组织. 同时发现25.0%胰腺癌组织标本没有检出hMLH1表达, 50.0%胰腺癌组织标本没有检出hMSH2表达, 33.9%没有检出hMLH3表达. 上述3种基因在肿瘤标本中的表达缺失频率亦均显著高于癌旁正常组织. 在对胰腺癌细胞系的研究中发现, hMLH1在PANC-1和CFPAC-1检出甲基化, 表达消失. hMSH2在PC-3检出甲基化, 表达消失. hMLH3在PC-3和PANC-1检出甲基化, 表达亦消失. 以上结果显示, 错配修复基因缺陷在胰腺癌是一种较为普遍的现象, 同时错配修复基因缺陷参与了部分胰腺癌的发病过程.
DNA错配修复基因能够识别并及时纠正DNA复制过程中错配的碱基对, 以保证复制的正确性和基因的稳定性. DNA错配修复缺陷常导致DNA复制过程中出现的错误无法得到纠正, 使整个基因组变得不稳定而诱导肿瘤发生.
李淑德, 主任医师, 中国人民解放军第二军医大学长海医院消化内科;周国雄, 主任医师, 南通大学附属医院消化内科
本研究对hMLH1、hMSH2和hMLH3等多个错配修复基因在胰腺癌中的表达情况及甲基化状态进行了综合研究, 以探讨错配修复缺陷在胰腺癌发病中的作用.
去甲基化治疗将成为胰腺癌治疗的一种潜在方法.
本研究方法成熟, 统计正确, 数据可靠, 结果明确, 讨论客观, 对临床工作有较大的指导价值.
编辑: 李军亮 电编:吴鹏朕
| 1. | Kondo E, Furukawa T, Yoshinaga K, Kijima H, Semba S, Yatsuoka T, Yokoyama T, Fukushige S, Horii A. Not hMSH2 but hMLH1 is frequently silenced by hypermethylation in endometrial cancer but rarely silenced in pancreatic cancer with microsatellite instability. Int J Oncol. 2000;17:535-541. [PubMed] [DOI] |
| 2. | Yamamoto H, Itoh F, Nakamura H, Fukushima H, Sasaki S, Perucho M, Imai K. Genetic and clinical features of human pancreatic ductal adenocarcinomas with widespread microsatellite instability. Cancer Res. 2001;61:3139-3144. [PubMed] |
| 3. | Zekri AR, Sabry GM, Bahnassy AA, Shalaby KA, Abdel-Wahabh SA, Zakaria S. Mismatch repair genes (hMLH1, hPMS1, hPMS2, GTBP/hMSH6, hMSH2) in the pathogenesis of hepatocellular carcinoma. World J Gastroenterol. 2005;11:3020-3026. [PubMed] [DOI] |
| 4. | Muscarella P, Melvin WS, Fisher WE, Foor J, Ellison EC, Herman JG, Schirmer WJ, Hitchcock CL, DeYoung BR, Weghorst CM. Genetic alterations in gastrinomas and nonfunctioning pancreatic neuroendocrine tumors: an analysis of p16/MTS1 tumor suppressor gene inactivation. Cancer Res. 1998;58:237-240. [PubMed] |
| 5. | Ueki T, Toyota M, Sohn T, Yeo CJ, Issa JP, Hruban RH, Goggins M. Hypermethylation of multiple genes in pancreatic adenocarcinoma. Cancer Res. 2000;60:1835-1839. [PubMed] |
| 6. | Sato N, Ueki T, Fukushima N, Iacobuzio-Donahue CA, Yeo CJ, Cameron JL, Hruban RH, Goggins M. Aberrant methylation of CpG islands in intraductal papillary mucinous neoplasms of the pancreas. Gastroenterology. 2002;123:365-372. [PubMed] [DOI] |
| 7. | House MG, Guo M, Iacobuzio-Donahue C, Herman JG. Molecular progression of promoter methylation in intraductal papillary mucinous neoplasms (IPMN) of the pancreas. Carcinogenesis. 2003;24:193-198. [PubMed] [DOI] |
| 8. | Dammann R, Schagdarsurengin U, Liu L, Otto N, Gimm O, Dralle H, Boehm BO, Pfeifer GP, Hoang-Vu C. Frequent RASSF1A promoter hypermethylation and K-ras mutations in pancreatic carcinoma. Oncogene. 2003;22:3806-3812. [PubMed] [DOI] |
| 9. | Kuroki T, Tajima Y, Kanematsu T. Role of hypermethylation on carcinogenesis in the pancreas. Surg Today. 2004;34:981-986. [PubMed] [DOI] |
| 10. | Martin ST, Sato N, Dhara S, Chang R, Hustinx SR, Abe T, Maitra A, Goggins M. Aberrant methylation of the Human Hedgehog interacting protein (HHIP) gene in pancreatic neoplasms. Cancer Biol Ther. 2005;4:728-733. [PubMed] [DOI] |
| 11. | Sinicrope FA, Rego RL, Halling KC, Foster N, Sargent DJ, La Plant B, French AJ, Laurie JA, Goldberg RM, Thibodeau SN. Prognostic impact of microsatellite instability and DNA ploidy in human colon carcinoma patients. Gastroenterology. 2006;131:729-737. [PubMed] [DOI] |
| 12. | Fang JY, Lu R, Mikovits JA, Cheng ZH, Zhu HY, Chen YX. Regulation of hMSH2 and hMLH1 expression in the human colon cancer cell line SW1116 by DNA methyltransferase 1. Cancer Lett. 2006;233:124-130. [PubMed] [DOI] |
| 13. | Bacani J, Zwingerman R, Di Nicola N, Spencer S, Wegrynowski T, Mitchell K, Hay K, Redston M, Holowaty E, Huntsman D. Tumor microsatellite instability in early onset gastric cancer. J Mol Diagn. 2005;7:465-477. [PubMed] [DOI] |
| 14. | Li JH, Shi XZ, Liu M, Wang Y, Yu ZH, Xu GW, Lu S. Methylation of hMLH1 gene promoter in gastric carcinoma with microsatellite instability. Ai Zheng. 2005;24:273-277. [PubMed] |
| 15. | Murata H, Khattar NH, Gu L, Li GM. Roles of mismatch repair proteins hMSH2 and hMLH1 in the development of sporadic breast cancer. Cancer Lett. 2005;223:143-150. [PubMed] [DOI] |
| 16. | Koster F, Schroer A, Fischer D, Horn AK, Diedrich K, Friedrich M. Immunohistochemistry of proteins for DNA mismatch repair in correlation to prognostic factors of mammarian cancer. Oncol Rep. 2007;17:1223-1227. [PubMed] [DOI] |
| 17. | Helmle KE, Otto CJ, Constantinescu G, Honore LH, Andrew SE. Variable MLH1 promoter methylation patterns in endometrial carcinomas of endometrioid subtype lacking DNA mismatch repair. Int J Gynecol Cancer. 2005;15:1089-1096. [PubMed] [DOI] |
| 18. | Takano K, Ichikawa Y, Ueno E, Ohwada M, Suzuki M, Tsunoda H, Miwa M, Uchida K, Yoshikawa H. Microsatellite instability and expression of mismatch repair genes in sporadic endometrial cancer coexisting with colorectal or breast cancer. Oncol Rep. 2005;13:11-16. [PubMed] [DOI] |
| 19. | Song H, Ramus SJ, Quaye L, DiCioccio RA, Tyrer J, Lomas E, Shadforth D, Hogdall E, Hogdall C, McGuire V. Common variants in mismatch repair genes and risk of invasive ovarian cancer. Carcinogenesis. 2006;27:2235-2242. [PubMed] [DOI] |
| 20. | Helleman J, van Staveren IL, Dinjens WN, van Kuijk PF, Ritstier K, Ewing PC, van der Burg ME, Stoter G, Berns EM. Mismatch repair and treatment resistance in ovarian cancer. BMC Cancer. 2006;6:201. [PubMed] [DOI] |