修回日期: 2006-11-01
接受日期: 2006-11-28
在线出版日期: 2007-02-18
目的: 构建羟基喜树碱耐药结肠癌细胞株SW1116/HCPT细胞的酵母双杂交cDNA文库.
方法: 用TRIzol从SW1116/HCPT细胞提取总RNA, 采用CLONTECH SMART (switching mechanism at 5'end of RNA transcript)技术和同源重组(homologous recombination)的方法构建SW1116/HCPT细胞的cDNA文库.
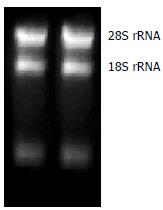
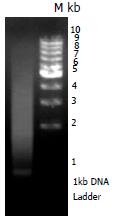
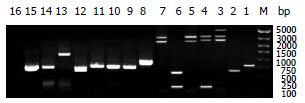
结果: 提取的RNA的A260/A280为1.98, 甲醛变性琼脂糖凝胶电泳示出28S rRNA、18S rRNA及5S rRNA 3条特异性带, 28S与18S带浓度及亮度比值约为2. 文库ds cDNA smear较长分布在0.1-5 kb, 成长瀑布条带, 而且在接近1 kb的区域有密集的带子, 为高丰度表达基因. 依据生长菌落计数, 共获得(1.2-1.9)×106个转化子, 重组率达93.7%, 文库插入片段大小为0.2-5 kb.
结论: 成功构建了SW1116/HCPT cDNA文库.
引文著录: 靳西凤, 冉志华, 陈翔, 萧树东. 结肠癌羟基喜树碱耐药细胞SW1116/HCPT酵母双杂交cDNA文库的构建和鉴定. 世界华人消化杂志 2007; 15(5): 500-504
Revised: November 1, 2006
Accepted: November 28, 2006
Published online: February 18, 2007
AIM: To construct a yeast two-hybrid cDNA library of hydroxycamptothecin-resistant human colon cancer cell line SW1116/HCPT in yeast cells.
METHODS: The total RNA of SW1116/HCPT cells was extracted using TRIzol method. Clontech SMART (switching mechanism at 5′end of RNA transcript) technique and homologous recombination mediated approach were used to construct cDNA library of SW1116/HCPT cells.
RESULTS: The ratio of A260 to A280 value of the extracted RNA was 1.98. A total of 3 bands (28S rRNA, 18S rRNA and 5S rRNA) were exhibited by agar gel electrophoresis, and the ratio of 28S rRNA and 18S rRNA was about 2. The ds cDNA smear in cDNA library was between 0.1-5 kb in length, with high expression at the 1-kb region. A total of (1.2-1.9)×106 recombinants were obtained from the cDNA library, and the recombination rate was 93.7%. The amplified polymerase chain reaction (PCR) fragments were between 0.2 and 5 kb in length.
CONCLUSION: The yeast two-hybrid cDNA library of SW1116/HCPT is successfully constructed by Clontech SMART technique in yeast cell.
- Citation: Jin XF, Ran ZH, Chen X, Xiao SD. Construction and characterization of yeast two-hybrid cDNA library of hydroxycamptothecin-resistant human colon cancer cell line SW1116/HCPT in yeast cells. Shijie Huaren Xiaohua Zazhi 2007; 15(5): 500-504
- URL: https://www.wjgnet.com/1009-3079/full/v15/i5/500.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i5.500
肿瘤的多药耐药性(multidrug resistance, MDR)是一个十分复杂的生物学过程, 影响因素、参与机制众多, 也是造成化疗失败的重要原因. 本研究组前期通过药物浓度递增诱导法, 建立了人结肠癌羟基喜树碱耐药细胞株(hydroxycamptothecin-resistant human colon cancer cell line SW1116/HCPT)[1]. 为了更好的探讨MDR现象和机制, 我们构建SW1116/HCPT细胞的酵母双杂交cDNA文库, 为进一步阐明羟基喜树碱耐药结肠癌细胞内的耐药蛋白之间的相互作用提供坚实的基础.
HCPT耐药细胞株SW1116/HCPT为本课题组前期建立, cDNA文库构建试剂盒(含酵母菌AH109, 质粒pGADT7-Rec)购自Clontech公司. 其中含SMART ⅢTM Oligo寡核苷酸: 5'-AAGCAGT GGTATCA ACGCAG AGTGGCC ATTATGGCCGGG-3'; CDSⅢ 引物: 5'-ATTCTAGAGG CCGAGGCG GCCGACATG-d (T)30VN-3'; LD-PCR上游引物: 5'-TTCCACCCAAGC AGTGGTAGAGGCCGAGGCGGCCGACA-3'和LD-PCR下游引物: 5'-GTATCGATGCCCACCCTCTAGAGGCCGAGGCGGCCGACA-3'. 文库扩增的上游引物5'-AGCAGTGGTATCAACGCAGAG-3'和下游引物: 5'-GCGGGGTTTTT CAGTATCTACGA-3'由上海生工合成. TRIzol购自Invitrogen公司. 酵母质粒抽提试剂盒购自Omega公司. 此外, YPDA培养基和SD/-Leu营养选择性培养基分别购自Oxford和Clontech公司.
1.2.1 SW1116/HCPT细胞培养: 用含100 mL/L胎牛血清的RPMI1640(Gibco BRL)完全培养液, 细胞置于含50 mL/L CO2、950 mL/L空气的CO2孵箱中, 在37℃、95%湿度条件下培养. 取对数生长期的细胞提取总RNA.
1.2.2 细胞总RNA提取: 按照TRIzol提供的操作说明书进行, 提取的总RNA用30 μL DEPC水溶解. 从样品中取2 μL稀释成400 μL, 以DEPC水作对照, 在Ultraspec 2000型紫外/可见分光光度计上测定A260和A280比值, 另取2 μL样品进行甲醛变性琼脂糖凝胶电泳. 同时进行RNA保温试验以验证RNA溶液中有无RNA酶污染.
1.2.3 cDNA第一链的合成: 在无菌的0.25 mL PCR薄壁管中加入1 μL 总RNA, 1.0 μL 10 μmol/L的CDSⅢ引物, 去离子水加至4 μL混匀, 稍离心, 72℃孵育2 min, 冰浴2 min. 轻轻混匀, 在室温下加入2.0 μL 第一链缓冲液, 1 μL 20 mmol/L DTT, 1.0 μL 10 mmol/L dNTP混合物, 1.0 μL的MMLV逆转录酶, 混匀, 稍离心, 42℃反应10 min, 加入1.0 μL 10 μmol/L SMART Ⅲ, 于42℃反应1 h. 75℃, 10 min终止第一链合成, 冷却至室温, 再加入1.0 μL RNase H, 37℃温育20 min, 以消化RNA.
1.2.4 LD-PCR合成: 在无菌的0.5 mL PCR薄壁管中按操作说明书依次加入: 第一链cDNA, 无菌双蒸水, 10×Advantage 2 PCR 缓冲液, 10 μmol/L的50×dNTP, PCR上、下游引物, 10×GC-Melt Solution, 50×Advantage 2 polymerase Mix. 在PTC240型PCR仪上进行: 95℃ 30 s先变性后再进行95℃ 10 s, 68℃ 6 min(后一次循环的延伸时间在前一次循环时间上加5 s)二温度PCR反应, 共20个循环: 最后一循环68℃ 5 min. 取5 μL产物进行12 g/L琼脂糖凝胶电泳.
1.2.5 双链cDNA(ds cDNA)的纯化: 纯化方法依照BD CHROMA SPINTM+T E-400 Columns (Clontech公司)说明书进行. 纯化柱颠倒数次以完全重悬凝胶基质, 纯化柱保持直立, 断去break-away端, 放入一灭菌的1.5 mL离心管中, 去掉顶盖. 1864 r/min离心5 min, 以平衡缓冲液, 重建基床, 纯化柱放入一新的1.5 mL离心管中, 将cDNA加到其内胶床平面的中心. 1864 r/min离心5 min, 离心管中即为纯化的ds cDNA.
1.2.6 AH109感受态的制备: PEG/LiAc法制备酵母感受态. 大致如下: 刮取少量酵母AH109贮存液接种于YPDA琼脂平板上, 30℃倒置培养直至克隆长出(2-3 d). 接种一个克隆(直径2-3 mm)于装有3 mL YPDA的15 mL无菌离心管中, 剧烈振荡以完全分散菌落, 30℃振荡孵育8 h. 将100 μL培养物转移到50 mL的YPDA培养基中, 30℃, 230-250 r/min振荡孵育16-20 h, 待A600为0.15-0.3时, 室温1864 r/min离心5 min收获酵母. 弃上清, 重新悬浮沉淀于100 mL YPDA, 30℃孵育3-5 h (A600为0.4-0.5), 室温1864 r/min离心5 min. 弃上清, 重新悬浮沉淀于60 mL三蒸水, 室温1864 r/min离心5 min. 弃上清, 重新悬浮沉淀于3 mL 1.1×TE/LiAc溶液, 分装在两个1.5 mL离心管(每管1.5 mL), 高速离心15 s. 弃上清, 重新悬浮沉淀于600 μL 1.1×TE/LiAc溶液.
1.2.7 同源重组及转化: 在7 mL离心管中加入ds cDNA (20 μL)、pGADT7- Rec (0.5 g/L 6 μL), 鲑鱼精DNA(取20 μL鲑鱼精DNA于离心管中100℃加热5 min, 置于冰上冷却, 在加样前重复几次, 使其变性彻底). 加入600 μL经上述处理的酵母AH109感受态细胞, 轻轻混匀. 加入2.5 mL PEG/LiAc溶液, 轻轻混匀, 30℃孵育45 min, 每15 min混匀一次. 加入160 μL DMSO, 混匀, 于42℃水浴20 min, 每10 min混匀一次. 1864 r/min离心5 min, 弃上清, 重新悬浮于3 mL YPD Plus溶液, 30℃振荡孵育90 min. 1864 r/min离心5 min, 弃上清, 重新悬浮于1 mL 9 g/L NaCl溶液. 然后分涂在10个直径为100 mm的SD/Leu选择性平板上. 30℃倒置培养3-6 d.
1.2.8 文库质粒DNA的评价: 取共转化产物按1:10、1:100、1:1000、1:0000稀释, 分别涂100 μL稀释液至100 mm SD/-Leu平板, 30℃倒置培养3-6 d. 计数平板菌落. 依据公式计算转化效率: 克隆数目(cfu)×总转化混合物体积(μL)/稀释混合物涂板体积×稀释系数×转化所用质粒量(μg). 随机挑取16个生长在上述SD/-Leu平板上的克隆分别接种于5 mL SD/-Leu液体培养基中, 30℃振荡培养16 h, 直至培养物饱和. 使用Yeast Plasmid Mini Kit (Omega公司)提取质粒DNA, 用文库扩增引物扩增插入片段, 并计算重组率.
提取的RNA之A260/A280为1.98. 甲醛变性琼脂糖凝胶电泳示, 28S rRNA、18S rRNA及5S rRNA 3条特异性带, 28S与18S带浓度及亮度比值约为2, 而且保温试验后RNA电泳结果如图1所示, 因此RNA质量较优可用于后续研究.
可见文库ds cDNA smear较长分布在0.1-5 kb, 成长瀑布条带, 而且在接近1 kb的区域有密集的带子, 为高丰度表达基因(图2).
依据生长菌落计数, 共获得(1.2-1.9)×106个转化子. 从平板上随机挑取16个单克隆, 提取质粒, 用文库扩增引物扩增插入片段, 该文库的cDNA插入片段在0.2-5 kb之间, 其中泳道3, 4, 5, 7, 12有2或3条带子, 可能是挑取单克隆时菌落不纯所致, 亦可能是由于质粒中插入了不同的片段所致, 由菌落PCR推算得知重组率为93.7% (图3).
目前, 构建全长cDNA文库的方法主要有以下几种: Oligo-capping法[2], CAP ture法[3], SMART法[4-5], Cap-Select法[6], Cap-jumping法[7]以及Cap -trapper法[8]等. 所有的这些方法都着眼于真核生物mRNA 5'端的帽子结构, 但又各有其独到之处. 总的说来, cDNA文库构建大致经分离总RNA, 纯化mRNA, 反转录成cDNA, 然后与两端带有限制性酶切位点的人工接头相连接, 并将其插入到载体中, 转染大肠杆菌, 得到cDNA文库. 而本课题组直接选用SMART技术构建耐药细胞株cDNA文库. Clontech公司提供的SMART[9]技术在构建cDNA文库时, 不需对总RNA中的mRNA进行分离纯化, 可以直接以微量的总RNA (50 ng)作为反转录的模板, 从而可避免因mRNA在分离纯化时的多步骤所造成mRNA的降解作用, 可以有效保证cDNA的完整性. 而且利用此方法所获得的cDNA全长比例较高.
在构建cDNA文库时, 将两端带有与线性穿梭表达质粒pGADT7-Rec (该质粒含L EU2筛选标志)两端相同序列的cDNA一起共转化酵母细胞, 即可在细胞内发生同源重组, 从而形成完整的环状文库质粒, 在相应的营养缺陷培养基中很容易筛选得到文库质粒. 与传统的cDNA文库构建方法相比, SMART方法省去了酶切、纯化、连接等环节, 减少了工作量, 提高了建库的效率. 但是, SMART方法也有其自身明显的缺点: PCR扩增具有偏性, 不利于长片段序列的扩增, 使得一些长片段全长基因丢失, 很难得到大于4 kb片段和低丰度全长基因, 造成了文库冗余性强. 另外一个缺点是: 寡聚脱氧鸟嘌呤核苷酸(dG)加尾效率低, 导致文库中基因信息丢失, 文库缺乏代表性. 因此, 人们采用Little-cycle SMART 和Large-size SMART这两种基于SMART改进的技术构建出质量较好的文库. Large-size SMART[10]在进行LD-PCR扩增合成ds cDNA前, 先将第一链产物按片段大小分出3-6 个等级, 再分别PCR 扩增和克隆. 这样就避免了PCR 扩增和载体连接对片段大小产生偏爱性的缺点, 使长片段的基因也能得到等效扩增和克隆. 而Little-cycle SMART技术是将PCR循环数减少, 因此减小了PCR扩增对长片段的不利影响, 提高了克隆长片段全长基因的可能性, 使得文库的代表性大大提高[9].
酵母菌是遗传工程中用于表达真核细胞基因较理想的受体细胞, 用酵母菌作为受体菌克隆真核基因比大肠杆菌有许多优点, 因此本研究采用酵母细胞来构建cDNA文库. 利用其具有较高的同源重组酶活性, 且酵母细胞用LiAc处理后, 其转化效率大大提高, 酵母细胞往往具有不同的可用来筛选的表型标志(营养缺陷表型), 易于将转化子筛选. 而且, 酵母细胞没有质粒不相容性, 一次转化时几个具有相同复制起始点的质粒可以同时转入一个酵母细胞中[11]. 为后续的酵母双杂交技术筛选[12-14]目的蛋白奠定了坚实的基础. 哺乳动物细胞含有10 000-30 000种不同的mRNA. 按丰度可分为低丰度、中丰度和高丰度3种, 其中低丰度mRNA占总mRNA 30% 左右, 每一细胞拥有的拷贝<14, 要克隆低丰度mRNA, 可按Clarke和Carbon公式[15]对文库大小进行估计: N = ln (1-P)/ln (1-1/n)(P为克隆到特定cDNA的概率, n为一种稀有mRNA在总mRNA中所占的比例, N = 所需克隆数), 若要求以99%概率得到一低丰度的克隆, 所需的文库克隆数为1.7×105. 我们所建文库已达到这一标准, 因此用此文库筛选目的基因是适当的, 可以用于后续研究.
肿瘤细胞的多药耐药性是造成化疗失败的主要原因, 各种耐药相关蛋白的表达和相互作用是肿瘤发生多药耐药现象的直接基础, 其确切机制尚不清楚. 本文构建SW1116/HCPT全长cDNA文库目的是利用酵母双杂交技术筛选与结肠癌羟基喜树碱耐药相关的蛋白质、探讨耐药蛋白质间的相互作用及其作用方式, 另外也可测表达序列标签ESTs (Expressed Sequcence Tags), 丰富SW1116/HCPT基因数据库, 同时也可以制作基因芯片.
肿瘤的多药耐药性(MDR)是一个十分复杂的生物学过程, 影响因素、参与机制众多, 也是造成化疗失败的重要原因. 为了更好的探讨MDR现象和机制, 本文构建了SW1116/HCPT细胞的酵母双杂交 cDNA文库.
全长cDNA文库的构建是功能基因组研究的一种经济、快速、有效的途径, 目前国内外构建 cDNA文库的方法主要有括Oligo-Capping, CAPture,SMART,CAP-trapper, CapSelect和CAPjumping等. 本文采用SMART技术构建cDNA文库, 节省了基因克隆和功能鉴定的时间, 同时也为后续的研究奠定了坚实的基础.
本文在国内外首 次构建了结肠癌羟基喜树碱耐药细胞cDNA文库,目的是通过酵母双杂交技术寻找可能与耐药蛋白相关蛋白相互作用的蛋白质, 并探讨其作用方式.
SMART技术与经典的cDNA文库构建方法相比显然 高效、快速、方便, 可直接以微量的总RNA (50 ng)作为反转录的模板, 从而可避免因 mRNA在分离纯化时的多步骤所造成mRNA的降解作用, 可以有效保证cDNA的完整性. 同时, 此方法所获得的cDNA全长比例较高, 因此应用价值也较大.
本文在前期工作的基础上, 构建SW1116/HCPT细胞的酵母双杂交 cDNA文库, 为进一步阐明羟基喜树碱耐药结肠癌细胞内的耐药蛋白之间的相互作用提供试验基础, 条理分明, 结论明确, 实 验 证据充足, 具有一定的科学意义.
电编: 李琪 编辑:王晓瑜
| 2. | Maruyama K, Sugano S. Oligo-capping: a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides. Gene. 1994;138:171-174. [PubMed] |
| 3. | Edery I, Chu LL, Sonenberg N, Pelletier J. An efficient strategy to isolate full-length cDNAs based on an mRNA cap retention procedure (CAPture). Mol Cell Biol. 1995;15:3363-3371. [PubMed] |
| 4. | Chenchik A, Moqadam F, Siebert P. A Laboratory Guide to RNA: Isolation, Analysis and Synthesis. New York: Wiley-Liss 1996; 273-321. |
| 5. | Du XJ, Wang JX, Liu N, Zhao XF, Li FH, Xiang JH. Identification and molecular characterization of a peritrophin-like protein from fleshy prawn (Fenneropenaeus chinensis). Mol Immunol. 2006;43:1633-1644. [PubMed] |
| 6. | Schmidt WM, Mueller MW. CapSelect: a highly sensitive method for 5' CAP-dependent enrichment of full-length cDNA in PCR-mediated analysis of mRNAs. Nucleic Acids Res. 1999;27:e31. [PubMed] |
| 7. | Efimov VA, Chakhmakhcheva OG, Archdeacon J, Fernandez JM, Fedorkin ON, Dorokhov YL, Atabekov JG. Detection of the 5'-cap structure of messenger RNAs with the use of the cap-jumping approach. Nucleic Acids Res. 2001;29:4751-4759. [PubMed] |
| 8. | Carninci P, Kvam C, Kitamura A, Ohsumi T, Okazaki Y, Itoh M, Kamiya M, Shibata K, Sasaki N, Izawa M. High-efficiency full-length cDNA cloning by biotinylated CAP trapper. Genomics. 1996;37:327-336. [PubMed] |
| 9. | Zhu YY, Machleder EM, Chenchik A, Li R, Siebert PD. Reverse transcriptase template switching: a SMART approach for full-length cDNA library construction. Biotechniques. 2001;30:892-897. [PubMed] |
| 10. | Wellenreuther R, Schupp I, Poustka A, Wiemann S. SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones. BMC Genomics. 2004;5:36. [PubMed] |
| 12. | 张 黎颖, 成 军, 邓 红, 郭 江, 郭 风劲, 王 巧侠. 酵母双杂交技术筛选白细胞cDNA文库中新基因NS2TP蛋白结合蛋白基因. 世界华人消化杂志. 2005;13:1688-1691. [DOI] |
| 13. | Huang YP, Zhang SL, Cheng J, Wang L, Guo J, Liu Y, Yang Y, Zhang LY, Bai GQ, Gao XS. Screening of genes of proteins interacting with p7 protein of hepatitis C virus from human liver cDNA library by yeast two-hybrid system. World J Gastroenterol. 2005;11:4709-4714. [PubMed] |