修回日期: 2007-09-27
接受日期: 2007-09-28
在线出版日期: 2007-10-28
目的: 探讨胃癌中caspase 9基因的表达及其单核苷酸多态位点的分布.
方法: 应用Western-blot方法检测70例胃癌患者及对应癌旁正常组织中caspase 9的表达情况. 应用限制性片段长度多态性技术结合测序, 分析70例胃患者及100例正常人caspase 9基因rs1052576位点基因型. 应用列联表法统计分析患者组和对照组SNP位点基因型及等位基因频率.
结果: 51.4%(36/70)的胃癌组织caspase 9基因表达明显下调, 与癌旁正常组织相比差异有统计学意义(P<0.05). 位于caspase 9基因第5外显子的SNP位点rs1052576的基因型频率和等位基因频率在两组人群中的分布差异有统计学意义. 患者组G等位基因频率明显高于对照组(22.9% vs 13.0%, P<0.05), AG基因型频率在有淋巴结转移的患者中明显高于无淋巴结转移的患者(P<0.05).
结论: caspase 9基因在胃癌中低表达, rs1052576多态位点G等位基因和胃癌的发生相关, AG基因型和淋巴结转移有关.
引文著录: 方长清, 刘树立, 娄毅, 李建华. 胃癌组织中caspase 9的表达及其多态性变化. 世界华人消化杂志 2007; 15(30): 3190-3193
Revised: September 27, 2007
Accepted: September 28, 2007
Published online: October 28, 2007
AIM: To explore the expression of caspase 9 in gastric cancer, and single nucleotide polymorphism (SNP) distribution of caspase 9 in gastric cancer patients and healthy individuals.
METHODS: Western blotting was used to analyze the expression of caspase 9 in 70 gastric cancer patients and normal gastric tissues. We investigated the distribution of a caspase 9 gene SNP. We analyzed the genotype rs1052576 in 70 patients and 100 healthy individuals by polymerase chain reaction-restriction fragment length polymorphism. Statistical analysis was applied to analyze SNP genotype frequency and allele frequency in patients and the control group.
RESULTS: In comparison with normal gastric tissues, caspase 9 gene expression was obviously down-regulated in 51.4% (36/70) of gastric cancer tissues. rs1052576 was located in exon 5 of the caspase 9 gene showed a significant difference between the two groups, and the G allele frequency in gastric cancer patients was higher than that in healthy controls (22.9% vs 13.0%, P < 0.05). AG genotype frequency in patients with lymph node metastasis was higher than that in patients with no lymph node metastasis (P < 0.05).
CONCLUSION: The caspase 9 gene is associated with gastric cancer oncogenesis. rs1052576 located in exon 5 of the caspase 9 gene is associated with gastric cancer. AG genotype is associated with lymph node metastasis.
- Citation: Fang CQ, Liu SL, Lou Y, Li JH. Expression of the caspase 9 gene and its polymorphism distribution in gastric cancer. Shijie Huaren Xiaohua Zazhi 2007; 15(30): 3190-3193
- URL: https://www.wjgnet.com/1009-3079/full/v15/i30/3190.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i30.3190
胃癌是我国最常见的消化道肿瘤, 占恶性肿瘤死亡的第一位. 胃癌的发生、发展是一个多因素、多基因作用的复杂过程. 目前已经发现很多和胃癌发生相关的基因, 如RAS基因家族、ERBB基因家族、MYC基因家族, p53等[1-2]. 凋亡和肿瘤的发生发展有密切的关系, 也是肿瘤耐药和产生化疗、放疗副作用的重要原因. caspase 9基因定位于1p36.3-p36.1, 参与线粒体介导的凋亡途径[3-4]. 为探讨caspase 9基因与胃癌的分子遗传学联系, 我们研究caspase 9基因在胃癌中的表达, 进一步选择caspase 9基因内已知单核苷酸多态(SNP), 检测其在70例胃癌患者及100例正常人中的分布情况, 分析其与NSCLC遗传易感性的关系.
选用中国医科大学附属第一医院普外科2000-2002年手术切除的胃腺癌组织70例, 标本的使用经伦理委员会批准并经患者本人知情同意. 其中男47例, 女23例, 年龄29-64(平均45.2)岁. 高分化9例, 中分化8例, 低分化53例, 淋巴结转移阴性24例, 阳性46例. 对应远癌胃黏膜组织均取自胃癌行胃切除的近侧断端胃黏膜, 所有断端均距离癌组织5 cm以上, 组织学检查为基本正常胃黏膜.
1.2.1 Westernblot分析caspase 9表达: 提取胃癌及正常胃组织的蛋白质后, 考马斯亮蓝法定量样品蛋白浓度, 把样品制成相同浓度, 加入样品缓冲液, 沸水煮5 min. 100 g/L SDS-PAGE凝胶电泳后转至PVDF膜. 取下PVDF膜TBS浸泡10 min, 封闭(50 g/L脱脂奶粉TBS 1 h), TBS洗5 min 2次, 然后加一抗(1:100), 4℃过夜, TBS洗5 min 2次, 加二抗2 h(1:2000), TBS洗5 min 2次. 将PVDF膜浸入碱性磷酸酶显色液1-2 min, 蒸馏水冲洗, 观察结果.
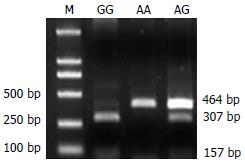
1.2.2 SNP的选取及引物设计: 在http://www.ncbi.nlm.nih.gov/SNP数据库选择caspase 9基因内已知SNP位点. rs1052576位于caspase 9基因第5外显子. 明确两侧基因组序列及SNP是否引起酶切位点改变. 应用Primer3软件设计引物(Sangon公司合成). rs1052576引物序列: CCTCCTGAGTAGCTGGGATT/AAGGGT GTTTTGGACACAAG, 长度464 bp, 酶切后片段长度307 bp/157 bp(BstUⅠ). 应用常规饱和酚-氯仿法提取胃癌患者及正常人外周静脉血基因组DNA.
1.2.3 目的片段扩增和酶切鉴定: 反应液总体积25 μL, 其中基因组DNA约40 ng, 10×反应缓冲液(MgCl2 15 mmol/L) 2.5 μL, dNTP(2.0 mmol/L) 2.0 μL, 上下游引物(20 μmol/L)各0.5 μL, Taq酶16.67 nkat. 循环参数: rs1052576: 94℃变性8 min, 94℃50 s、58℃ 50 s、72℃ 50 s 35个循环, 72℃延伸5 min. 所有PCR产物均经20 g/L琼脂糖凝胶电泳检测确认. BstUⅠ酶切鉴定rs1052576基因型, 等位基因为G时能被BstUⅠ酶识别. 酶切反应体系20 μL, 其中PCR产物8 μL, BstUⅠ 83.35 nkat, 10×缓冲液2 μL. 37℃水浴过夜, 15 g/L琼脂糖凝胶电泳鉴定基因型.
统计学处理 应用Arlequin软件(http://anth-ropologie.unige.ch/arle-quin/methods.html)检测病例组与对照组中的Hardy-Weinberg平衡, 应用SPSS10.0软件统计分析患者组和对照组中基因型频率、等位基因频率以及caspase 9基因表达率是否存在差异.
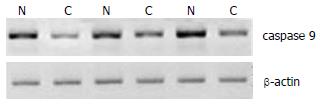
9基因表达 免疫印记结果表明, 正常胃黏膜组织中均有caspase 9的表达, 而51.4%(36/70)胃癌组织caspase 9表达明显下调(图1), 与癌旁正常组织相比差异有统计学意义.
经Arlequin软件分析, 对照组和患者组中2个SNP位点基因型频率及等位基因频率均符合Hardy-Weinberg平衡(图2). 患者组及正常对照组SNP位点基因型频率及等位基因频率有区别. rs1052576位点患者组G等位基因频率高于对照组(χ2 = 9.52, P<0.01). 患者组与对照组相比, 3种基因型分布频率存在差异(χ2 = 10.40, P<0.01), AA基因型频率降低, AG基因型频率增高(P<0.01, 表1). 进一步分析发现rs1052576基因型频率与患者淋巴结转移有关(表2).
| 分组 | n | 基因型频率 | 等位基因频率 | |||
| GG | AG | AA | G | A | ||
| 患者组 | 70 | 16 | 39 | 15 | 71 | 69 |
| (22.9) | (55.7) | (21.4) | (52.1) | (47.9) | ||
| 对照组 | 100 | 13 | 42 | 45 | 68 | 132 |
| (13.0) | (42.0) | (45.0) | (34.0) | (66.0) | ||
| 临床特征 | rs1052576 | χ2 | P | ||
| GG | AG | AA | |||
| 男 | 10 | 28 | 9 | 0.89 | 0.64 |
| 女 | 6 | 11 | 6 | ||
| 高分化 | 2 | 3 | 4 | 3.82 | 0.43 |
| 中分化 | 2 | 4 | 2 | ||
| 低分化 | 12 | 32 | 9 | ||
| 淋巴结转移 | |||||
| (+) | 12 | 29 | 5 | 8.88 | 0.01 |
| (-) | 4 | 10 | 10 | ||
胃癌是我国最常见的消化道肿瘤, 全国平均年死亡率16/10万, 高发区达60/10万. 胃癌的发生、发展是一个多因素、多基因作用的复杂过程. 对细胞凋亡的耐受是肿瘤细胞逃避化疗药物治疗和免疫介导破坏的一个潜在机制. 对于大多数抗肿瘤药物, 细胞凋亡始于细胞色素C/Apaf-1/caspase 9途径[5-6]. 失去激活细胞凋亡途径可以导致细胞对多种化疗药物的耐受. 在dATP和ATP的作用下, Apaf-1/细胞色素C复合物通过CARD-CARD结构域相互作用与caspase 9前体结合, 从而激活caspase 9, 即成熟caspase 9从凋亡小体中释放出来[7-9]. 一旦与Apaf-1结合, caspase 9就会被细胞色素C和dATP激活, 进一步激活caspase 3, 从而引起细胞凋亡. caspase 3的激活依赖于caspase 9.
caspase 9基因定位于1p36.3-p36.1, 编码的蛋白产物分子量约为46 kDa, 广泛表达于人类的正常组织, 如神经组织、肌肉组织、肝脏、脾脏、胃和肺等[10]. 有研究表明, 在胃癌、皮肤黑色素瘤和白血病中都发现caspase 9基因转录和翻译水平的下调[11-12], 目前国内尚未有该基因在胃癌中的表达研究. 我们应用免疫印记方法发现在70例胃癌中有36例出现了caspase 9基因表达的明显下调, 在癌旁正常对照胃黏膜组织却未发现明显改变, 二者对比差异有统计学意义, 提示凋亡基因caspase 9基因与胃癌的发生相关. 另有研究表明, 非小细胞肺癌中caspase 9基因的单核苷酸多态位点rs1052576的等位基因频率和基因型频率的分布与正常人群存在差异[13]. 因此, 我们进一步研究该单核苷酸多态在胃癌中的分布情况, 利用PCR-RFLP方法对70例胃癌和100例正常人进行了rs1052576位点的基因型分析. 结果发现, rs1052576位点患者组G等位基因频率高于对照组(χ2 = 9.52, P<0.01). 患者组与对照组相比, 3种基因型分布频率存在差异(χ2 = 10.40, P<0.05), AA基因型频率降低, AG基因型频率增高. 进一步分析rs1052576基因型频率在胃癌患者中的分布, 发现3种基因型的分布与胃癌患者的性别及分化程度无关, 与淋巴结转移相关(χ2 = 8.88, P = 0.01), 提示rs1052576(Gln221Arg)位点位于caspase 9基因第5外显子, 编码的氨基酸位于CASc结构域. rs1052576位点的G等位基因与胃癌的发生相关, 可能是由于蛋白质空间结构的不同, 影响其在线粒体介导的凋亡caspase 3的激活, 进而阻碍正常的细胞凋亡过程, 引起胃癌的发生. 因此, 在胃癌易感人群中筛查rs1052576位点的G等位基因有利于胃癌的早期诊断与预防. rs1052576位点AG基因型与淋巴结转移有关, 可能作为判断患者病情的新指标.
胃癌的发生发展涉及到很多凋亡基因的异常. 我们将进一步研究caspase 9在胃癌的作用机制, 随着研究的深入可能为其早期临床基因诊断及患者预后的判断提供新的指标, 为基因治疗提供新的靶点.
凋亡和肿瘤的发生发展有着密切的关系, 也是肿瘤耐药和产生化疗、放疗副作用的重要原因. caspase 9基因定位于1p36.3-p36.1, 其参与线粒体介导的凋亡途径.
本文应用免疫印记方法发现在70例胃癌中有36例出现caspase 9基因表达的明显下调, 进一步分析rs1052576基因型频率在胃癌患者中的分布, 发现3种基因型的分布与胃癌患者的性别及分化程度无关, 与淋巴结转移相关.
研究表明, 在胃癌、皮肤黑色素瘤和白血病中都发现了caspase 9基因转录和翻译水平的下调, 非小细胞肺癌中caspase 9基因的单核苷酸多态位点rs1052576的等位基因频率和基因型频率的分布与正常人群存在差异.
凋在胃癌易感人群中筛查rs1052576位点的G等位基因有利于胃癌的早期诊断与预防. rs1052576位点AG基因型与淋巴结转移有关, 可能作为判断患者病情的新指标.
本文选题新颖, 分析有据, 数据可信, 有较好的学术价值.
编辑: 程剑侠 电编:何基才
| 1. | Correa P, Schneider BG. Etiology of gastric cancer: what is new? Cancer Epidemiol Biomarkers Prev. 2005;14:1865-1868. [PubMed] |
| 2. | Kountouras J, Zavos C, Chatzopoulos D. New concepts of molecular biology on gastric carcinogenesis. Hepatogastroenterology. 2005;52:1305-1312. [PubMed] |
| 3. | Soengas MS, Capodieci P, Polsky D, Mora J, Esteller M, Opitz-Araya X, McCombie R, Herman JG, Gerald WL, Lazebnik YA. Inactivation of the apoptosis effector Apaf-1 in malignant melanoma. Nature. 2001;409:207-211. [PubMed] |
| 4. | Fu WN, Bertoni F, Kelsey SM, McElwaine SM, Cotter FE, Newland AC, Jia L. Role of DNA methylation in the suppression of Apaf-1 protein in human leukaemia. Oncogene. 2003;22:451-455. [PubMed] |
| 5. | Cecconi F, Alvarez-Bolado G, Meyer BI, Roth KA, Gruss P. Apaf1 (CED-4 homolog) regulates pro-grammed cell death in mammalian development. Cell. 1998;94:727-737. [PubMed] |
| 6. | Fortin A, Cregan SP, MacLaurin JG, Kushwaha N, Hickman ES, Thompson CS, Hakim A, Albert PR, Cecconi F, Helin K. APAF1 is a key transcriptional target for p53 in the regulation of neuronal cell death. J Cell Biol. 2001;155:207-216. [PubMed] |
| 7. | Robles AI, Bemmels NA, Foraker AB, Harris CC. APAF-1 is a transcriptional target of p53 in DNA damage-induced apoptosis. Cancer Res. 2001;61:6660-6664. [PubMed] |
| 8. | Ho AT, Li QH, Hakem R, Mak TW, Zacksenhaus E. Coupling of caspase-9 to Apaf1 in response to loss of pRb or cytotoxic drugs is cell-type-specific. EMBO J. 2004;23:460-472. [PubMed] |
| 9. | Zou H, Henzel WJ, Liu X, Lutschg A, Wang X. Apaf-1, a human protein homologous to C. elegans CED-4, participates in cytochrome c-dependent activation of caspase-3. Cell. 1997;90:405-413. [PubMed] |
| 10. | Lemaire C, Godefroy N, Costina-Parvu I, Rincheval V, Renaud F, Trotot P, Bouleau S, Mignotte B, Vayssiere JL. Caspase-9 can antagonize p53-induced apoptosis by generating a p76(Rb) truncated form of Rb. Oncogene. 2005;24:3297-3308. [PubMed] |
| 11. | Frank O, Brors B, Fabarius A, Li L, Haak M, Merk S, Schwindel U, Zheng C, Muller MC, Gretz N. Gene expression signature of primary imatinib-resistant chronic myeloid leukemia patients. Leukemia. 2006;20:1400-1407. [PubMed] |
| 12. | Kuwahara D, Tsutsumi K, Oyake D, Ohta T, Nishikawa H, Koizuka I. Inhibition of caspase-9 activity and Apaf-1 expression in cisplatin-resistant head and neck squamous cell carcinoma cells. Auris Nasus Larynx. 2003;30 Suppl:S85-88. [PubMed] |
| 13. | Lou Y, Fang CQ, Li JH. A study on the expression of CASP9 gene and its polymorphism distribution in non-small cell lung cancer. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2007;24:59-62. [PubMed] |