修回日期: 2007-08-19
接受日期: 2007-08-28
在线出版日期: 2007-08-28
对肿瘤研究的不断深入使人们认识到几乎所有人类肿瘤都是由表观遗传学异常与基因改变共同引起并促进其演变的. 表观遗传学改变主要包括DNA甲基化改变和组蛋白修饰, 这两种调控模式本身及之间的相互作用网络可导致印记丢失, 染色质重塑, 非必需重复序列的转录、基因的异常活化以及抑制肿瘤发生和演进基因的异常沉默等, 在异常的克隆性增生和癌性增生的级联事件起始中发挥重要作用, 在酵母模型中的研究更深入的理解了肿瘤干细胞的定义以及引起肿瘤的相关分子机制. 表观遗传学的研究为进一步了解肿瘤的各种特性, 优化肿瘤的早期诊断, 改善肿瘤的预后提供了新的策略.
引文著录: 郑丹, 刘彬彬, 刘银坤. 肿瘤表观遗传学研究进展. 世界华人消化杂志 2007; 15(24): 2631-2637
Revised: August 19, 2007
Accepted: August 28, 2007
Published online: August 28, 2007
A growing body of evidence suggests that almost all tumors result from both epigenetic abnormality and gene mutation, which then promote tumor progression. Epigenetic abnormality mainly includes changes in DNA methylation and the modification of histone. These two regulatory modes and the corresponding interacting network between them can lead to the loss of imprinting, chromatin remodeling, transcription of repeat sequences, abnormal activation of genes, and inactivation of tumor suppressors. Furthermore, they also have a vital role in aberrant cell proliferation and in the earliest stages of tumorigenesis. Research into yeast models provides a basis for a better understanding and definition of cancer stem cells and the relative molecular mechanisms that induce tumors. Research in the field of tumor epigenetics shows promise as a way to understand the various characteristics of tumors, to optimize the early diagnosis of tumors, and to improve the prognosis for patients with tumors.
- Citation: Zheng D, Liu BB, Liu YK. Research progress in the field of tumor epigenetics. Shijie Huaren Xiaohua Zazhi 2007; 15(24): 2631-2637
- URL: https://www.wjgnet.com/1009-3079/full/v15/i24/2631.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i24.2631
表观遗传是指表型状态发生改变而基因型未发生变化, 有可逆潜能, 但是在细胞分化期间通常保持稳定, 是一种对肿瘤演进具有重要作用的可遗传的基因表达模式. 他在整体上可以影响染色体的包装, 局部则影响与肿瘤发生、发展相关的重要基因的转录. 最新的研究表明, 肿瘤表观遗传改变在干细胞的基因表达上发挥举足轻重的作用, 通过DNA甲基化和组蛋白修饰协同作用促进肿瘤发生.
研究表明, 几乎所有类型的人类肿瘤中都存在表观遗传上的异常, 这种改变可导致DNA结构功能的改变、非必需重复序列的转录、基因的异常活化、染色体复制调控机制的破坏以及抑制肿瘤发生和演进基因(其中涉及到经典的抑癌基因, 肿瘤演进过程中的转录因子, 组织重塑基因, DNA修复基因, 细胞周期调控基因, 抗凋亡基因以及在肿瘤发生中阻止异常发育通路活化的基因)的异常沉默等. 在癌基因中进行的沉默基因的随机筛查表明在任何的肿瘤中都可能隐藏着异常沉默的基因[1]. 通常基因的沉默只涉及到少数的基因, 但最近的报道表明, 在结肠癌中, 基因的沉默涉及到染色体臂上的广泛区域[2]. 这种大范围的基因沉默使基因群更易于发生染色质改变以及伴随的大量基因的转录抑制. 然而抑癌基因功能的丢失更大程度上是由表观遗传的异常而不是基因的变化引起的[3], 染色质的凝集和修饰也控制基因表达的模式. 在哺乳动物细胞中, 存在一个动态的调控模式来调节基因CpG岛胞嘧啶的甲基化修饰、核小体重塑和一系列的组蛋白修饰, 以保证DNA的正确组装从而调节转录抑制与激活之间的平衡. 基因组DNA的高度甲基化是转录抑制的显著特征, 高甲基化DNA处于一种紧密结构中, 这种结构维持其抑制状态[4]. 而处于转录活跃状态的基因启动子和一系列具有协同作用的调控基因则维持在一个较开放的转录构型状态. 这种抑制或开放的状态与染色质的状态相关, 其中涉及到组蛋白不同程度激活或抑制的修饰[5-6]. 最近在基因组范围内对染色体标记的描述又有了新的进展, 人们认为染色体标记可能是同细胞谱系定型阶段相伴随的, 或者可能是在这些阶段发挥控制作用[7]. 肿瘤表观遗传改变会影响干细胞表型的维持从而诱发肿瘤的生成; 他还与衰老, 应激反应等诱导细胞发生异常克隆的肿瘤诱因相关. 这些信息将有利于理解表观遗传学改变在异常的克隆性增生和癌性增生的级联事件的起始中发挥的重要作用.
肿瘤表观遗传学异常的提出源于在肿瘤中发现了遗传印记的异常. 遗传印记是指来自父母双方的等位基因在通过精子和卵子传递给子代时发生了修饰, 使带有亲代印记的等位基因具有不同的表达特性. 正常的基因印记受DNA甲基化和组蛋白乙酰化和甲基化修饰的调控, 以保证父系基因的决定地位, 即一对等位基因中一个发生转录而另外一个受到抑制. 肿瘤中一些基因丢失其遗传印记后会导致异常情况发生, 比如两个等位基因在肿瘤中共表达. 印记丢失引起的肿瘤发生可以在胚胎衍生的过程中找到明确的证据: 胚胎在只具有母系来源的染色体的情况下将形成卵巢畸胎瘤, 而在只具有父系染色体的情况下则形成了葡萄胎. 在成人肿瘤中研究最明确的就是类胰岛素生长因子2(IGF2)基因, 当他的等位基因发生共表达后则会产生一个过度强效的生长因子[8]. IGF2的印记丢失可在结肠癌的患者的正常结肠上皮中发现, 与不断增高的结肠癌风险相关[9]. 另一个例子是Wilm's瘤, IGF2的印记丢失后产生了一种异常的未分化细胞, 这种细胞可以不断的增生并且不受相关的修复和调控基因的影响, 从而引发肾癌. IGF2基因印记丢失的机制包括H19基因启动子甲基化的异常增多而导致其转录沉默以及位于同一个染色体远端的增强子发生转移等[10].
最近Holm et al[11]通过破坏DNA甲基化转移酶DNMT1暂时诱导基因组DNA脱甲基化, 从而建立了一个基因印记丢失的小鼠模型. 从这些小鼠获得的成纤维细胞可在免疫缺陷型小鼠身上形成肿瘤而且还具有体外永生的特性. 当这些细胞在转入H-Ras癌基因后可以被完全诱导同步化. Holm认为单独的印记丢失就可以使细胞形成癌性增生, 细胞通过印记丢失降低了其转化为永生性细胞的域值. 在没有基因突变的情况下, 可遗传基因表达模式的变化导致干细胞的异常增生, 并进一步诱发肿瘤的发生、
发展.
肿瘤表观遗传学异常以DNA甲基化模式的改变为主, 包括整体基因组的低甲基化和启动子的高甲基化, 目前倍受关注的就是启动子区域CpG岛的高甲基化导致的抑癌基因转录沉默, 这也很可能是癌性增生的最初表现. Issa et al[12]证实了CpG岛甲基化表型(CpG island methylator phenotype, CIMP)的存在, 其特征是同时存在的多种基因具有肿瘤特异性的CpG岛甲基化. 常见的抑癌基因p16ink4A就是由于启动子区域CpG岛的高甲基化而丧失功能[13]. 来自基因敲除小鼠的数据显示该基因的丢失可以延长干细胞的寿命[14]. p16ink4A的丢失以及随之产生的基因不稳定状态使早期的癌性增生易于发生[15].
与肿瘤异常甲基化状态相关的DNA甲基化转移酶DNMTs, 通过共价的形式在CpG岛的胞嘧啶上结合一个甲基基团. 使用5-aza-2-deoxycytidine(胞嘧啶类似物)抑制DNMT的活性后, 可延缓小鼠肺癌模型中的癌性增生[16]. 在胃, 膀胱, 非白血性白血病, 大肠, 肺等各种组织类型的肿瘤中DNMT蛋白和活动水平都提高. 哺乳动物的DNA甲基化模式多来自于对正常小鼠的研究. 任何一种Dnmt基因(Dnmt1, Dnmt3a, Dnmt3b)的纯合型缺失对小鼠都是致死性的[17]. 研究表明Dnmt3a, Dnmt3b的缺失阻止了从头甲基化, 而Dnmt1缺失导致大量DNA去甲基化, 反映了Dnmt1的维持甲基化的作用[18]. 然而在结肠癌和其他肿瘤细胞中同时运用基因敲除和RNA干扰技术方法去除Dnmt1[19]后, 发现严重的Dnmt1的缺失却只使整体DNA甲基化水平产生了微弱的减少, 沉默的抑癌基因也未检测到有重新表达[20]. Yan et al[21]的研究发现了MDA-MB-231和Hs578t这2株乳腺癌细胞需要DNMT1来维持雌激素受体的甲基化和抑制状态. 这种对DNMT1不同需要量的机制可能涉及到人类肿瘤细胞中的DNMTs之间在维持DNA甲基化时具有协同效应. 从DNMT1-/-的大肠癌细胞HCT116中敲除从头甲基化酶DNMT3b, 可导致95%的基因组5-甲基化胞嘧啶去甲基化, 全部启动子去甲基化以及沉默基因的重新表达[22]. 尽管DNMT1对于未甲基化的物质几乎没有从头甲基化活性, 但其过度表达依然导致了人类成纤维细胞中的CpG岛序列的从头甲基化. 未甲基化的人类CpG岛是DNMT1从头甲基化活性的良好的底物[23]. 因此DNMT1很可能在肿瘤细胞中有引发CpG岛的异常高甲基化的能力.
组蛋白是核小体的重要组成部分. 核小体由147个DNA碱基对包裹在核心组蛋白(H2A, H2B, H3, H4)上组成, 核小体中组蛋白和147个DNA碱基的位置以及组蛋白的修饰(包括赖氨酸乙酰化, 赖氨酸和精氨酸甲基化, 丝氨酸和苏氨酸磷酸化, 谷氨酸ADP核糖基化, 赖氨酸泛素化)对维持基因的表达模式和染色体正常的结构功能发挥作用, 以此来调控正常的表观遗传. 核小体的这些组分处于一个精细的稳定状态, 任何外界微小变化就会对细胞表型和转录模式产生巨大的影响. 在酵母中, 组蛋白SIR2单个拷贝的变化就可以引起一个区带的基因发生转录沉默, 如果发生在染色体着丝点将会影响更多的基因[24].
对于DNA包装来说, 组蛋白修饰和DNA甲基化之间有密切的联系. 研究表明选择型组蛋白去乙酰化酶HDACsⅠ和Ⅱ、各种DNMTs的复合体、特定甲基化CpG序列结合的蛋白质家族MBDs之间相互关联[25]. MBDs具有特异结合基因启动子的能力, 和DNA高甲基化和肿瘤中异常沉默的基因相关. 转录活跃的基因由乙酰化组蛋白包围, 转录抑制的基因就由去乙酰化组蛋白包围[26]. 在调控基因转录时甲基化和赖氨酸乙酰化虽然相互协作, 但甲基化是占主导地位的. 例如必须在抑制Ⅰ和Ⅱ型HDACs之前首先用去甲基化因素减少甲基化, 才能有效的激活转录[1]; 去甲基化也可导致MBDs蛋白以及HDACs从启动子里释放.
组蛋白乙酰基转移酶(HATs)和组蛋白去乙酰化酶(HDACs)相互作用调节组蛋白乙酰化的平衡, 同时组蛋白甲基化转移酶(HMTs)和组蛋白去甲基化酶有效的调控着组蛋白甲基化[27]. 组蛋白赖氨酸乙酰化与转录激活相关联, 而这些残基的甲基化则同时与激活以及抑制状态有关[28]. 组蛋白乙酰化和甲基化模式通过不同的效应蛋白而体现, 含有bromo结构域的蛋白质, 可以识别乙酰化的赖氨酸残基和chromo结构域, 从而结合到甲基化的赖氨酸残基上, 作用于组蛋白并引起基因转录. 赖氨酸甲基化有单甲基化, 双甲基化以及三甲基化3种不同的形式, 他们显著扩大了组蛋白复合体的密码信息. 比如, H3K9乙酰化和H3K4甲基化是表达的活跃标志; 而H3K9和H3K27的单、双和三甲基化的大量出现则是转录抑制相关的组蛋白甲基化的标志. H3K9的三甲基化和异染色质的高度紧密的结构相关, H3K9双甲基化和常染色质的沉默相关, H3K27甲基化则与♀哺乳动物的X染色体的失活相关[26]. 从果蝇属到人类H3K27甲基化均由一种蛋白质复合体PcG(polycomb protein complexes)定位, PcG参与调节基因的长期沉默, 在癌基因沉默的起始也具有重要作用[29]. 这种复合体至少有4个组群, 包括维持复合体PRC1(RNF2, HPC, ECR, BMI1组成)和起始复合体(从PRC2到PRC4). PRC2复合物中EZH2的SET结构域可以催化H3K27和H1K26的甲基化, 而在所有的高甲基化的DNA和癌基因的启动子区域都可以发现H3K27甲基化[26]. EZH2的表达水平和前列腺癌和乳腺癌的发生及预后相关[30]. PRC2的另一个复合物SUZ12, 在结肠癌, 乳腺癌以及肝癌中表达上调[15]. 也有报道称PRC1的组分Bmil在肿瘤中表达增加并且导致造血系统和脑部的异常的克隆增生[31]. 最近一些研究还认为EZH2和Bmil作用还与DNMTs相关[32], 导致DNA高甲基化和基因沉默.
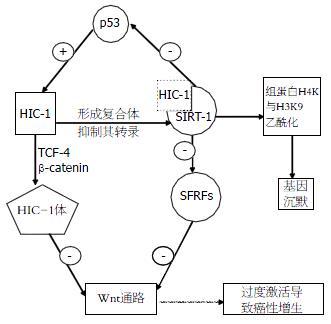
表观遗传学改变在干细胞的异常克隆性增生以及肿瘤的易感性等方面发挥关键性作用. 对慢性炎症导致肿瘤的研究人们发现肿瘤的表观遗传调控具有一个潜在性的网络(图1). 正常组织对细胞应激和损伤的应答是暂时的, 通过反馈通路调控对应激的抵抗力, 细胞更新, 组织修复和凋亡反应之间相互协调. 其中凋亡反应取决于p53基因介导的细胞周期检查点机制, 增加能够产生DNA损伤的活性氧而抑制细胞存活[33]. 然而慢性暴露于应激状态下则引起上述应答反应的异常延长从而可能导致异常的克隆增生. 这种异常的应答反应涉及SIRT1的持续性增加, 抑癌基因p53的转录抑制以及抑癌基因HIC1的沉默. SIRT1是一个多功能的应激敏感蛋白, 是组蛋白去乙酰化酶(HDACs)Ⅲ的一种, 其去乙酰化活性参与了p53基因的翻译后修饰从而下调了p53基因的转录活性[34]. HIC1基因是锌指结构域的一个成员, 包含转录抑制物POZ家族. 他本身也是一个p53基因的转录激活目标[35]. HIC1和SIRT1组成一个复合体并向SIRT1启动子聚集, 抑制SIRT1的转录[36].
实验证明, HIC1基因纯合型敲除的小鼠具有多种致命的上皮缺陷, 在胚胎性成纤维细胞中SIRT1的水平增高8倍; 杂合型小鼠则具有肿瘤易感性. 这表明HIC1基因功能的表观遗传丢失可以增加SIRT1, 从而降低p53基因对正常细胞的作用. 研究发现增加SIRT1家族中的Sir2基因的一个拷贝, 就可以延长有机体包括从新杆状线虫到哺乳动物的细胞寿命. HIC1缺陷的小鼠和人类肿瘤细胞在对DNA损伤引发的凋亡应答过程中, 都有p53的减少和SIRT1的增加. HIC1基因表达异常可以抑制凋亡反应并延长细胞寿命, 从而使早期异常克隆性增生易于发生.
SIRT1还可以进一步引起其他的表观遗传学改变. 如SIRT1去乙酰化可导致染色体末端着丝点和配对区域基因的转录沉默, 并且维持这种抑制的状态[37]. Pruitt et al[38]诱导降低SIRT1的活性, 发现可以使这些沉默基因的重新表达. 这一过程涉及到了Wnt发育信号通路[39]. Wnt发育信号通路对成人细胞更新时干细胞的扩增以及维持具有重要作用. 在结肠癌中, 由于对抗Wnt信号通路的SFRPs基因家族(编码细胞表面的膜蛋白)[40]首先发生了基因沉默[41], 随后当下游通路基因(如APC和β-catenin)突变时因缺少拮抗作用而使Wnt通路被过度激活, 从而促进了肿瘤的发展[42]. 在肿瘤细胞中实验性的敲除SIRT1可以使已经沉默的对抗Wnt通路的基因(SFRPs)的重新激活, 从而逆转了在乳腺癌和结肠癌细胞中的Wnt通路的过度激活. 值得一提的是HIC1基因的表观遗传沉默也涉及到其他路径, HIC1基因沉默后可导致Wnt通路的过度激活. HIC1结合TCF-4后可以募集β-catenin到紧凑的核结构上, 构成HIC1体, 就会妨碍驱动正常的Wnt通路的正常核β-catenin-TCF复合体的形成[43]. 另一方面, SIRT1和PcG复合物在肿瘤中异常的基因沉默维持过程中可能共同发挥重要作用. SIRT1作用的具体机制以及SIRT1在建立和维持DNA甲基化的整体作用越来越被人们重视[44].
关于"肿瘤干细胞", 过去的观点认为成熟的干细胞发生异常的克隆增生, 这导致了细胞异质性的不断增强, 因此很多肿瘤就呈现出一系列的演进步骤[45]. 新的观点强调肿瘤增生是由肿瘤干细胞引起的, 肿瘤干细胞内的一些分子可以促进肿瘤的发展. 例如, 特定干细胞基因Oct4的过表达可以促进肠和其他器官的上皮细胞的肿瘤迅速发生[46].
最近一些报道表明胚胎干细胞可以通过染色体的组建来保持自身的状态. 在鼠和人的胚胎干细胞中, 细胞干细胞化的转录因子, 包括OCT4, NANOG和SOX2, 都定位在一个限制性的启动子区域[7]. 这些转录因子、细胞增殖调控基因、组织分化调控基因等都普遍和干细胞的多潜能性相关. 他们在干细胞中大部分保持一种低表达的状态, 他们近侧的启动子区包含PcG蛋白和重要的抑制标志-三甲基化的H3K23. 胚胎发育状态的稳定依赖于基因的表达模式. 例如当基因需要表达的时候, 这种PcG的影响可以被逆转或消失[5]. 直到细胞后续的成熟阶段或在成熟细胞的分化阶段, PcG复合物的作用可能重新发挥, 保持长时间的基因沉默[47]. PcG蛋白保持一个精确协调的、可塑的基因表达模式, 其诱导的染色体标记在胚胎发育过程中能够保持干细胞和分化发育细胞表型之间的平衡.
PcG在正常的干细胞中的最开始的标记很可能涉及到一类非编码RNA. 从植物到酵母的研究证实了是由这类非编码RNA参与而触发了转录抑制[48]. 在酵母中这种RNA可以作用于组蛋白修饰性酶, 特别是催化H3K9和H3K27的酶类参与并触发了转录抑制. 最近的研究认为HDACs在酵母转录沉默最初建立的过程中扮演重要的角色[49], 同时一些研究发现dsRNA作用于基因的启动子区可以重建哺乳动物模型中的基因沉默[50]. 在这种类型的沉默中可以发现在组蛋白H3K9上抑制性标记明显增多[51]. 这种dsRNA依赖的转录抑制(RdTS)是否直接和DNA的甲基化相关还有待进一步的证实. 但RdTS和组蛋白修饰之间的有力的联系预示着RdTS可能是一种内源性的机制, 细胞通过这个机制建立表观遗传调控.
总之, 尽管肿瘤表观遗传学的研究进展十分迅速, 仍有许多分子机制和人类肿瘤发生演进的关系有待更深入的了解. 表观遗传学的研究为进一步了解肿瘤的各种特性, 优化肿瘤的早期诊断, 丰富肿瘤的治疗方式, 改善肿瘤的预后提供了新的策略, 具有重要的临床治疗意义. 无论从基础研究还是从临床研究的角度, 表观遗传学状态的建立和平衡维持都将我们对肿瘤的研究带进了一个崭新的时代, 也为我们指明了一个新的有潜力的方向.
几乎所有人类肿瘤都是由表观遗传学异常与基因改变共同引起并促进其演变的. 与染色质相关的异常变化常出现在恶性肿瘤产生之前. DNA甲基化改变和组蛋白修 饰, 这两种表观遗传学改变及之间相互作用网络可导致印记丢失, 染色质重塑, 基因的异常活化以及沉 默等, 在异常的克隆性增生和癌性增生的级联事件起始中发挥重要作用.
目前倍受关注的就是由一系列甲基化修饰和组蛋白修饰导致的启动子区域CpG岛的高甲基化导致的抑癌基因转录沉默和癌基因低甲基化导致的过度激活. 这也很可能是癌性增生的最初表现. 最近人们认为染色体标记可能是同细胞谱系定型阶段相伴随的, 或者可能是在这些阶段发挥控制作用.
最近Guillermo Barreto et al报道了Gadd45a(生长抑制和DNA损伤诱导蛋白45alpha), 一种涉及基因组稳定, DNA修复和细胞生长抑制的核蛋白, 在促进DNA去甲基化过程中发挥重要作用. Gadd45a通过DNA修复解除基因表观遗传学沉默, 其过表达激活去甲基化信号质粒(methylation-silenced reporter plasmids)同时促进整体基因组去甲基化.
本文阐述了肿瘤表观遗传改变在干细胞的基因表达中举足轻重的作用, 通过DNA甲基化和组蛋白修饰协同作用促进肿瘤发生. 同时强调了“肿瘤干细胞”的新观点, 认为肿瘤增生是由肿瘤干细胞引起的, 肿瘤干细胞内的一些分子可以促进肿瘤的发展. 本文还进一步系统阐述了早期肿瘤表观遗传改变调控的潜在性网络.
对于染色质结构, 组蛋白修饰, DNA甲基化研究的深入, 以及肿瘤表观遗传学改变的可逆性, 为人们提供了新的肿瘤的治疗方法和预防策略. 异常的表观遗传学修饰可能成为肿瘤治疗的新靶点, 同时异常 DNA甲基化模式提供了三个强有力的诊断应用: 分级标志物、敏感检测标志物、风 险评估标志物.
染色质重塑: 指在能量驱动下核小体的置换或重新排列, 暴露基因转录启动子区中的顺式作用元件, 增加了转录因子和启动子的可接近性从而导致基因活化和转录. 他主要有2种类型: 一是依赖ATP的物理修饰; 二是共价化学修饰:多发生在组蛋白N末端尾巴, 尤其组蛋白H3 和H4 的修饰,包括乙酰化、磷酸化、甲基化和泛素化等, 他们控制着基因转录和其他染色质调控过程.
本文论述了肿瘤表观遗传学的研究进展, 题目和内容相应, 摘要简明扼要, 文章层次分明, 内容新颖, 反映了表观遗传学方面的进展, 参考文献较新, 而且文献来源杂志级别较高, 有较高的学术价值.
编辑: 程剑侠 电编:张敏
| 1. | Suzuki H, Gabrielson E, Chen W, Anbazhagan R, van Engeland M, Weijenberg MP, Herman JG, Baylin SB. A genomic screen for genes upregulated by demethylation and histone deacetylase inhibition in human colorectal cancer. Nat Genet. 2002;31:141-149. [PubMed] |
| 2. | Frigola J, Song J, Stirzaker C, Hinshelwood RA, Peinado MA, Clark SJ. Epigenetic remodeling in colorectal cancer results in coordinate gene suppression across an entire chromosome band. Nat Genet. 2006;38:540-549. [PubMed] |
| 3. | Jones PA, Baylin SB. The fundamental role of epigenetic events in cancer. Nat Rev Genet. 2002;3:415-428. [PubMed] |
| 4. | Robertson KD. DNA methylation and human disease. Nat Rev Genet. 2005;6:597-610. [PubMed] |
| 5. | Bernstein BE, Mikkelsen TS, Xie X, Kamal M, Huebert DJ, Cuff J, Fry B, Meissner A, Wernig M, Plath K. A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell. 2006;125:315-326. [PubMed] |
| 6. | Boyer LA, Plath K, Zeitlinger J, Brambrink T, Medeiros LA, Lee TI, Levine SS, Wernig M, Tajonar A, Ray MK. Polycomb complexes repress developmental regulators in murine embryonic stem cells. Nature. 2006;441:349-353. [PubMed] |
| 7. | Lee TI, Jenner RG, Boyer LA, Guenther MG, Levine SS, Kumar RM, Chevalier B, Johnstone SE, Cole MF, Isono K. Control of developmental regulators by Polycomb in human embryonic stem cells. Cell. 2006;125:301-313. [PubMed] |
| 8. | Feinberg AP, Ohlsson R, Henikoff S. The epigenetic progenitor origin of human cancer. Nat Rev Genet. 2006;7:21-33. [PubMed] |
| 9. | Sakatani T, Kaneda A, Iacobuzio-Donahue CA, Carter MG, de Boom Witzel S, Okano H, Ko MS, Ohlsson R, Longo DL, Feinberg AP. Loss of imprinting of Igf2 alters intestinal maturation and tumorigenesis in mice. Science. 2005;307:1976-1978. [PubMed] |
| 10. | Kato Y, Sasaki H. Imprinting and looping: epigenetic marks control interactions between regulatory elements. Bioessays. 2005;27:1-4. [PubMed] |
| 11. | Holm TM, Jackson-Grusby L, Brambrink T, Yamada Y, Rideout WM 3rd, Jaenisch R. Global loss of imprinting leads to widespread tumorigenesis in adult mice. Cancer Cell. 2005;8:275-285. [PubMed] |
| 12. | Issa JP. CpG island methylator phenotype in cancer. Nat Rev Cancer. 2004;4:988-993. [PubMed] |
| 13. | Belinsky SA. Gene-promoter hypermethylation as a biomarker in lung cancer. Nat Rev Cancer. 2004;4:707-717. [PubMed] |
| 14. | Janzen V, Forkert R, Fleming HE, Saito Y, Waring MT, Dombkowski DM, Cheng T, DePinho RA, Sharpless NE, Scadden DT. Stem-cell ageing modified by the cyclin-dependent kinase inhibitor p16INK4a. Nature. 2006;443:421-426. [PubMed] |
| 15. | Reynolds PA, Sigaroudinia M, Zardo G, Wilson MB, Benton GM, Miller CJ, Hong C, Fridlyand J, Costello JF, Tlsty TD. Tumor suppressor p16INK4A regulates polycomb-mediated DNA hypermethylation in human mammary epithelial cells. J Biol Chem. 2006;281:24790-24802. [PubMed] |
| 16. | Belinsky SA, Nikula KJ, Baylin SB, Issa JP. Increased cytosine DNA-methyltransferase activity is target-cell-specific and an early event in lung cancer. Proc Natl Acad Sci U S A. 1996;93:4045-4050. [PubMed] |
| 17. | Okano M, Bell DW, Haber DA, Li E. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell. 1999;99:247-257. [PubMed] |
| 18. | Gruenbaum Y, Cedar H, Razin A. Substrate and sequence specificity of a eukaryotic DNA methylase. Nature. 1982;295:620-622. [PubMed] |
| 19. | Ting AH, Jair KW, Schuebel KE, Baylin SB. Differential requirement for DNA methyltransferase 1 in maintaining human cancer cell gene promoter hypermethylation. Cancer Res. 2006;66:729-735. [PubMed] |
| 20. | Suzuki H, Watkins DN, Jair KW, Schuebel KE, Markowitz SD, Chen WD, Pretlow TP, Yang B, Akiyama Y, Van Engeland M. Epigenetic inactivation of SFRP genes allows constitutive WNT signaling in colorectal cancer. Nat Genet. 2004;36:417-422. [PubMed] |
| 21. | Yan L, Nass SJ, Smith D, Nelson WG, Herman JG, Davidson NE. Specific inhibition of DNMT1 by antisense oligonucleotides induces re-expression of estrogen receptor-alpha (ER) in ER-negative human breast cancer cell lines. Cancer Biol Ther. 2003;2:552-556. [PubMed] |
| 22. | Suzuki M, Sunaga N, Shames DS, Toyooka S, Gazdar AF, Minna JD. RNA interference-mediated knockdown of DNA methyltransferase 1 leads to promoter demethylation and gene re-expression in human lung and breast cancer cells. Cancer Res. 2004;64:3137-3143. [PubMed] |
| 23. | Jair KW, Bachman KE, Suzuki H, Ting AH, Rhee I, Yen RW, Baylin SB, Schuebel KE. De novo CpG island methylation in human cancer cells. Cancer Res. 2006;66:682-692. [PubMed] |
| 24. | Kimura A, Umehara T, Horikoshi M. Chromosomal gradient of histone acetylation established by Sas2p and Sir2p functions as a shield against gene silencing. Nat Genet. 2002;32:370-377. [PubMed] |
| 25. | Burgers WA, Fuks F, Kouzarides T. DNA methyltransferases get connected to chromatin. Trends Genet. 2002;18:275-277. [PubMed] |
| 26. | McGarvey KM, Fahrner JA, Greene E, Martens J, Jenuwein T, Baylin SB. Silenced tumor suppressor genes reactivated by DNA demethylation do not return to a fully euchromatic chromatin state. Cancer Res. 2006;66:3541-3549. [PubMed] |
| 27. | Yamane K, Toumazou C, Tsukada Y, Erdjument-Bromage H, Tempst P, Wong J, Zhang Y. JHDM2A, a JmjC-containing H3K9 demethylase, facilitates transcription activation by androgen receptor. Cell. 2006;125:483-495. [PubMed] |
| 28. | Briggs SD, Xiao T, Sun ZW, Caldwell JA, Shabanowitz J, Hunt DF, Allis CD, Strahl BD. Gene silencing: trans-histone regulatory pathway in chromatin. Nature. 2002;418:498. [PubMed] |
| 29. | Pirrotta V, Gross DS. Epigenetic silencing mechanisms in budding yeast and fruit fly: different paths, same destinations. Mol Cell. 2005;18:395-398. [PubMed] |
| 30. | Matsukawa Y, Semba S, Kato H, Ito A, Yanagihara K, Yokozaki H. Expression of the enhancer of zeste homolog 2 is correlated with poor prognosis in human gastric cancer. Cancer Sci. 2006;97:484-491. [PubMed] |
| 31. | Bruggeman SW, Valk-Lingbeek ME, van der Stoop PP, Jacobs JJ, Kieboom K, Tanger E, Hulsman D, Leung C, Arsenijevic Y, Marino S. Ink4a and Arf differentially affect cell proliferation and neural stem cell self-renewal in Bmi1-deficient mice. Genes Dev. 2005;19:1438-1443. [PubMed] |
| 32. | Vire E, Brenner C, Deplus R, Blanchon L, Fraga M, Didelot C, Morey L, Van Eynde A, Bernard D, Vanderwinden JM. The Polycomb group protein EZH2 directly controls DNA methylation. Nature. 2006;439:871-874. [PubMed] |
| 33. | Lu H, Ouyang W, Huang C. Inflammation, a key event in cancer development. Mol Cancer Res. 2006;4:221-233. [PubMed] |
| 34. | Langley E, Pearson M, Faretta M, Bauer UM, Frye RA, Minucci S, Pelicci PG, Kouzarides T. Human SIR2 deacetylates p53 and antagonizes PML/p53-induced cellular senescence. EMBO J. 2002;21:2383-2396. [PubMed] |
| 35. | Guerardel C, Deltour S, Pinte S, Monte D, Begue A, Godwin AK, Leprince D. Identification in the human candidate tumor suppressor gene HIC-1 of a new major alternative TATA-less promoter positively regulated by p53. J Biol Chem. 2001;276:3078-3089. [PubMed] |
| 36. | Chen WY, Wang DH, Yen RC, Luo J, Gu W, Baylin SB. Tumor suppressor HIC1 directly regulates SIRT1 to modulate p53-dependent DNA-damage responses. Cell. 2005;123:437-448. [PubMed] |
| 37. | Suka N, Luo K, Grunstein M. Sir2p and Sas2p opposingly regulate acetylation of yeast histone H4 lysine16 and spreading of heterochromatin. Nat Genet. 2002;32:378-383. [PubMed] |
| 38. | Pruitt K, Zinn RL, Ohm JE, McGarvey KM, Kang SH, Watkins DN, Herman JG, Baylin SB. Inhibition of SIRT1 reactivates silenced cancer genes without loss of promoter DNA hypermethylation. PLoS Genet. 2006;2:e40. [PubMed] |
| 39. | Gregorieff A, Clevers H. Wnt signaling in the intestinal epithelium: from endoderm to cancer. Genes Dev. 2005;19:877-890. [PubMed] |
| 40. | Lacher MD, Siegenthaler A, Jager R, Yan X, Hett S, Xuan L, Saurer S, Lareu RR, Dharmarajan AM, Friis R. Role of DDC-4/sFRP-4, a secreted frizzled-related protein, at the onset of apoptosis in mammary involution. Cell Death Differ. 2003;10:528-538. [PubMed] |
| 41. | Akino K, Toyota M, Suzuki H, Mita H, Sasaki Y, Ohe-Toyota M, Issa JP, Hinoda Y, Imai K, Tokino T. The Ras effector RASSF2 is a novel tumor-suppressor gene in human colorectal cancer. Gastroenterology. 2005;129:156-169. [PubMed] |
| 42. | Baylin SB, Ohm JE. Epigenetic gene silencing in cancer - a mechanism for early oncogenic pathway addiction? Nat Rev Cancer. 2006;6:107-116. [PubMed] |
| 43. | Valenta T, Lukas J, Doubravska L, Fafilek B, Korinek V. HIC1 attenuates Wnt signaling by recruitment of TCF-4 and beta-catenin to the nuclear bodies. EMBO J. 2006;25:2326-2337. [PubMed] |
| 44. | Kuzmichev A, Margueron R, Vaquero A, Preissner TS, Scher M, Kirmizis A, Ouyang X, Brockdorff N, Abate-Shen C, Farnham P. Composition and histone substrates of polycomb repressive group complexes change during cellular differentiation. Proc Natl Acad Sci U S A. 2005;102:1859-1864. [PubMed] |
| 45. | Valk-Lingbeek ME, Bruggeman SW, van Lohuizen M. Stem cells and cancer; the polycomb connection. Cell. 2004;118:409-418. [PubMed] |
| 46. | Hochedlinger K, Yamada Y, Beard C, Jaenisch R. Ectopic expression of Oct-4 blocks progenitor-cell differentiation and causes dysplasia in epithelial tissues. Cell. 2005;121:465-477. [PubMed] |
| 47. | Ringrose L, Paro R. Epigenetic regulation of cellular memory by the Polycomb and Trithorax group proteins. Annu Rev Genet. 2004;38:413-443. [PubMed] |
| 48. | Cam H, Grewal SI. RNA interference and epigenetic control of heterochromatin assembly in fission yeast. Cold Spring Harb Symp Quant Biol. 2004;69:419-427. [PubMed] |
| 49. | Yamada T, Fischle W, Sugiyama T, Allis CD, Grewal SI. The nucleation and maintenance of heterochromatin by a histone deacetylase in fission yeast. Mol Cell. 2005;20:173-185. [PubMed] |