修回日期: 2007-02-01
接受日期: 2007-02-13
在线出版日期: 2007-05-18
目的: 构建能表达靶向肾母细胞瘤过度表达基因(NOV)的小干扰RNA(siRNA)的重组质粒, 研究NOV对大鼠肝星状细胞(HSC)活化、增殖、凋亡、分泌细胞外基质(ECM)的影响.
方法: 以NOV为目的基因, 以质粒psiRNA-hH1neo为载体, 构建能在真核细胞中表达的靶向NOV的siRNA的重组质粒psiRNA (psiRNA1, 2, 3)和阴性对照重组质粒pconsiRNA. 限制性酶切和测序鉴定构建成功后, 脂质体介导重组质粒转染HSC, 依据转染质粒的不同将HSC分为psiRNA1组、psiRNA2组、psiRNA3组、阴性对照pconsiRNA组, 并以未转染重组质粒的HSC为空白对照组. 半定量RT-PCR检测HSC的NOV、α-平滑肌肌动蛋白(α-SMA)、Ⅰ型胶原、Ⅲ型胶原mRNA表达情况, Western blot检测α-SMA蛋白表达, MTT法检测细胞增殖, 流式细胞仪分析细胞凋亡.
结果: 限制性酶切和测序鉴定表明成功构建了NOVsiRNA表达质粒; 与阴性对照组相比, 转染外源重组质粒psiRNA2的HSC内NOV、α-SMA的mRNA表达水平下降(73.0% vs 23.2%, 51.4% vs 15.1%, 均P<0.05), Ⅰ型胶原、Ⅲ型胶原mRNA表达水平明显下降(59.8% vs 17.0%, 37.1% vs 6.6%, P<0.05), α-SMA蛋白表达水平下降. 与空白对照组相比, 转染外源重组质粒psiRNA2的HSC内HSC增殖活性显著降低(24 h: 0.172±0.005 vs 0.318±0.018, P<0.05; 48 h: 0.296±0.004 vs 0.472±0.029, P<0.05; 72 h: 0.432±0.024 vs 0.672±0.050, P<0.05). psiRNA1组、psiRNA3组的HSC内NOV mRNA、α-SMA、Ⅰ型胶原、Ⅲ型胶原的表达及HSC增殖活性均无明显下降(均P>0.05).
结论: NOV可促进HSC增殖、活化及分泌细胞外基质, 提示NOV可以作为肝纤维化基因治疗的一个新的靶位点.
引文著录: 徐冬, 林菊生, 任精华, 陈琼, 姚津剑, 何星星. 肾母细胞瘤过度表达基因对大鼠肝星状细胞生物学行为的影响. 世界华人消化杂志 2007; 15(14): 1602-1608
Revised: February 1, 2007
Accepted: February 13, 2007
Published online: May 18, 2007
AIM: To construct recombinant plasmids that can express small interfering RNA (siRNA) targeting nephroblastoma overexpressed gene (NOV) and investigate its effect on the biological behaviors of hepatic stellate cells (HSCs).
METHODS: Hairpin siRNA templates targeting NOV gene were synthesized and cloned into plasmid vector psiRNA-H1neo. Three vector-derived siRNAs (denoted psiRNA1, 2 and 3) and one mocking pconsiRNA (as control) were constructed. The recombinant NOV siRNA plasmids were constructed and identified using restrictive enzyme analysis and DNA sequencing, and then transfected into HSCs by lipofectamine. HSCs were divided into group psiRNA1, psiRNA2, psiRNA3 and pconsiRNA, transfected with the corresponding recombinant plasmids. Blank group consisted of HSCs contaning no plasmids. The expression of NOV and α-smooth muscle actin (α-SMA) were detected by semi-quantitative reverser transcription-polymerase chain reaction (RT-PCR) and Western blot. The mRNA expression of type Ⅰ collagen and type Ⅲ collagen were detected by semi-quantitative RT-PCR. Cell proliferation was assayed by MTT method and cell apoptosis by flow cytometry.
RESULTS: Restrictive enzyme analysis and DNA sequencing revealed the successful construction of siRNA expression plasmids. Compared with negative control group, extrogenous recombinant plasmid psiRNA2 reduced the mRNA levels of NOV (decreased rate: 73.0% vs 23.2%, P < 0.05), type Ⅰ collagen (decreased rate: 59.8% vs 17.0%, P < 0.05), type Ⅲ collagen (decreased rate: 37.1% vs 6.6%, P < 0.05), and inhibit the expression of α-SMA at mRNA level (decreased rate: 51.4% vs 15.1%, P < 0.05) and protein level. Compared with non-transfection group, extrogenous recombinant plasmid psiRNA2 significantly decreased the proliferating activity of HSC (24 h: 0.172 ± 0.005 vs 0.318 ± 0.018, P < 0.05; 48 h: 0.296 ± 0.004 vs 0.472 ± 0.029, P < 0.05; 72 h: 0.432 ± 0.024 vs 0.672 ± 0.050, P < 0.05). No obvious changes were found in psiRNA1 group and psiRNA3 group (all P > 0.05).
CONCLUSION: NOV can increase the secretion of extracellular matrix (ECM) and promote the proliferation and activation of HSCs. NOV may be a novel target for gene therapy of liver fibrosis.
- Citation: Xu D, Lin JS, Ren JH, Chen Q, Yao JJ, He XX. Effect of nephroblastoma overexpressed gene on biological behaviors of rat hepatic stellate cells. Shijie Huaren Xiaohua Zazhi 2007; 15(14): 1602-1608
- URL: https://www.wjgnet.com/1009-3079/full/v15/i14/1602.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i14.1602
肝纤维化是肝硬化的前期病理阶段, 是肝脏受到慢性损伤时, 细胞外基质(extracellular matrix, ECM)可逆性沉积的创伤愈合过程. 肝星状细胞(hepatic stellate cell, HSC)的活化和增殖是肝纤维化发生的关键环节[1]. 肾母细胞瘤过度表达基因(nephroblastoma overexpressed gene, NOV)是一种胰岛素样生长因子(insulin like growth factor, IGF)结合蛋白(IGF binding proteins, IGFBPs), 属于CCN家族的成员[2-3]. Lee et al[4]研究发现活化的HSC中NOV基因表达上升. 但NOV对HSC的作用目前尚无研究. RNA干扰(RNA interference, RNAi)是一种由小干扰RNA(small interfering RNA, siRNA)诱发的特定基因沉默[5], 基于此的RNAi技术在基因功能研究及基因治疗中发挥了重要作用. 本实验构建靶向NOV的siRNA的表达载体, 观察其对NOV表达和HSC活化、增殖及表达细胞外基质的影响, 以此来研究NOV基因对大鼠HSC生物学行为的影响.
质粒DNA提取纯化试剂盒、胶回收试剂盒购自北京博大泰克生物基因技术有限责任公司; RNA提取试剂TRIzol购自上海华舜生物工程有限公司; BbsⅠ和AseⅠ内切酶、Taq酶购自Fermentas公司; T4DNA连接酶购于TaKaRa公司; M-MLV购自Promega公司; 真核细胞转染试剂Lipofectamine2000为Invitrogen公司产品; 噻唑蓝购自Amersco公司; 小鼠抗α-平滑肌肌动蛋白(α-smooth muscle actin, α-SMA)抗体购自武汉博士德生物工程有限公司, 小鼠抗GAPDH mAb购自武汉三鹰生物科技有限公司, 辣根酶标记山羊抗小鼠IgG(H+L)购自武汉凌飞科技有限公司; 其他试剂均为国产分析纯. HSC由福建医科大学翁山耕教授惠赠[6]. 真核表达载体psiRNA-hH1neo由本实验室留存.
1.2.1 靶向NOV基因的siRNA表达载体的构建: 根据RNAi软件分析, 设计寡核苷酸片段, 以consiRNA为阴性对照, 序列如表1. 单链寡核苷酸片段由上海生工生物工程技术服务有限公司合成, 经过退火、连接、转化、筛选、AseⅠ酶切鉴定后, 并进一步DNA测序证实这些阳性克隆的正确性.
| 名称 | 序列 | 目标起始位置 |
| ConsiRNA正义链 | 5'-TCCCAGCCTCCAAACTGTGAGTCTCTCCACCAGAGACTCACAGTTTGGAGGCTT-3' | |
| ConsiRNA反义链 | 5'-CAAAAAGCCTCCAAACTGTGAGTCTCTGGTGGAGAGACTCACAGTTTGGAGGCT-3' | |
| siRNA1正义链 | 5'-TCCCAGTTCCAGAGGGAGACAACTGTCCACCACAGTTGTCTCCCTCTGGAACTT-3' | 292 |
| siRNA1反义链 | 5'-CAAAAAGTTCCAGAGGGAGACAACTGTGGTGGACAGTTGTCTCCCTCTGGAACT-3' | |
| siRNA2正义链 | 5'-TCCCAGAAATCCATCCACCTACAGTTCCACCAACTGTAGGTGGATGGATTTCTT-3' | 795 |
| siRNA2反义链 | 5'-CAAAAAGAAATCCATCCACCTACAGTTGGTGGAACTGTAGGTGGATGGATTTCT-3' | |
| siRNA3正义链 | 5'-TCCCAGCTGCTGTACGCCCTTCAATACCACCTATTGAAGGGCGTACAGCAGCTT-3' | 878 |
| siRNA3反义链 | 5'-CAAAAAGCTGCTGTACGCCCTTCAATAGGTGGTATTGAAGGGCGTACAGCAGCT-3' | |
1.2.2 HSC培养与形态学观察: 大鼠HSC系rHSC-99是从正常Wistar大鼠分离出来的一个HSC系. 细胞于37℃快速复苏, 接种于玻璃培养瓶中, 于50 mL/L二氧化碳、950 mL/L潮湿空气的二氧化碳培养箱里培养, 24 h细胞完全贴壁后换液, 以后每3-4 d换1次培养液, 每隔4-8 h用OlympusCKX41相关倒置显微镜进行观察, 待细胞长满单层, 经胰蛋白酶消化并传代.
1.2.3 细胞瞬时转染: 细胞分5组: HSC分别用重组质粒psiRNA1, psiRNA2, psiRNA3和pconsiRNA转染细胞, 设未转染组为空白对照. 转染前1 d, 用胰蛋白酶消化液消化细胞并计数, 将HSC细胞按每孔3×105浓度接种到6孔细胞培养板, 同时按每孔3×103浓度接种到96孔细胞培养板, 应用真核细胞转染试剂Lipofectamine2000转染, 6孔板, 每孔转染4.0 µg重组质粒, 96孔板每孔转染0.2 µg重组质粒.
1.2.4 MTT法检测HSC增殖: 96孔细胞培养板内每组设8个复孔, 同时另设8个复孔不加细胞作为调零孔, 均在转染24, 48和72 h后测定. 测定时分别在每孔加入5 g/L噻唑蓝20 mL, 再培养4 h, 弃去上清后加入二甲基亚砜(DMSO) 200 µL, 振荡10 min后, 用酶标仪(波长490 nm)测定HSC吸光度(A).
1.2.5 RNA抽提及RT-PCR分析: 6孔细胞培养板内细胞转染后48 h, 按TRIzol说明书抽提细胞总RNA, 溶于20 µL DEPC-H2O, 测A260/A280值, 计算RNA浓度. 取2 µg RNA, 按Promega公司M-MLV说明书操作行逆转录. 反应条件: 42℃ 60 min, 95℃ 5 min, 以所得cDNA为模板行PCR, 按Fermentas公司Taq酶说明书操作, 引物序列如下(表2).
| 引物名称 | 引物序列 | 扩增片段长度(bp) | 退火温度(℃) |
| NOV上游引物 | 5'-ATGGTTCCAGAGGGAGACAA-3' | 647 | 52.8 |
| NOV下游引物 | 5'-CCTGGCAAACACTGAAACTC-3' | ||
| α-SMA上游引物 | 5'-TGTGCTGGACTCTGGAGATG-3' | 292 | 54 |
| α-SMA下游引物 | 5'-GATCACCTGCCCATCAGG-3' | ||
| GAPDH上游引物 | 5'-TGCACCACCAACTGCTTAG-3' | 177 | 53 |
| GAPDH下游引物 | 5'-GGATGCAGGGATGATGTTC-3' | ||
| Ⅰ型胶原上游引物 | 5'-GAGGCATAAAGGGTCATCGTG-3' | 715 | 54 |
| Ⅰ型胶原下游引物 | 5'-GAACCTTCGCTTCCATACTCG-3' | ||
| Ⅲ型胶原上游引物 | 5'-CATTGCTGGAGTTGGAGGTG-3' | 704 | 50 |
| Ⅲ型胶原下游引物 | 5'-GTCTCATGGCCTTGCGTGTT-3' |
反应条件: PCR循环94℃ 45 s, 退火温度30 s, 72℃ 45 s, 30个循环; 72℃ 7 min. 扩增产物于20 g/L琼脂糖凝胶中电泳, 应用JS2380自动图像凝胶分析仪(上海培清公司), 分析每条带的光密度值和所占的百分比, 并和GAPDH比较, 计算各组细胞的基因表达差异.
1.2.6 蛋白质抽提及Western blot分析: 收集6孔板内细胞用冰PBS漂洗3次, 离心, 弃上清液, 加蛋白裂解液[含50 mmol/L Tris-HCl (pH7.5), 150 mmol/L NaCl, 1 mL/L TritonX-100, 10 g/L NaDeoxycholate, 1 g/L SDS, 2 mmol/L EDTA, 1 mmol/L PMSF及蛋白酶抑制剂]抽提细胞蛋白, 用Braford法测蛋白浓度. 取40 µg蛋白样本放沸水震荡水浴5 min, 以100 g/L SDS-聚丙烯酰胺凝胶电泳分离a-SMA和GAPDH蛋白, 转蛋白至硝酸纤维素膜; 50 g/L脱脂奶粉封闭2 h; 一抗[a-SMA小鼠mAb(1∶100稀释); GAPDH小鼠mAb(1∶1000稀释)]4℃孵育过夜; TBS-T(含2 mL/L Tween20, 20 mmol/L Tris-HCl, 150 mmol/L NaCl, pH7.4)洗膜3次, 每次10 min; 二抗(辣根过氧化物酶标记羊抗小鼠IgG 1∶2000稀释)37℃孵育1 h, TBS-T洗膜3次, 每次10 min, 应用DAB显色, 处理后观查结果. 应用JS2380自动图像凝胶分析仪(上海培清公司)对DAB显色结果进行扫描分析, 以GAPDH为内参照.
1.2.7 siRNA对HSC凋亡的影响: 收集转染后的细胞, 用预冷的PBS液洗涤2次, 然后用-20℃预冷的700 mL/L乙醇固定过夜, PBS重悬, 用10 mg/L PI和5 g/L RNase在室温下避光进行DNA染色, 采用FACS流式细胞仪检测细胞凋亡, 数据采用ModFit软件分析.
统计学处理 数据资料用mean±SD表示, 采用SPSS11.0进行方差分析, 以P<0.05表示差异有统计学意义.
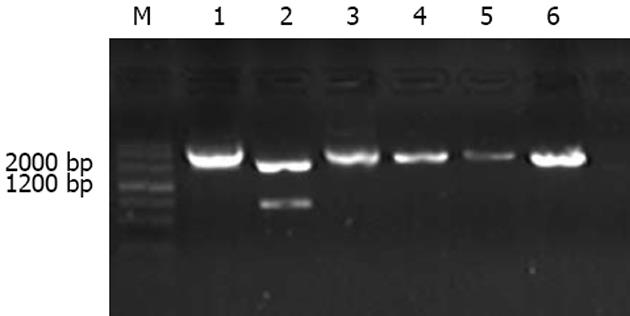
经双酶切纯化的线性质粒psiRNA-hH1neo与退火形成的双链siRNA插入片段按一定的比例连接, 连接后转入DH5α, 经卡那霉素板行蓝白斑筛选, 筛选出的阳性克隆抽提质粒, 内切酶AseⅠ酶切分析鉴定为psiRNA1, psiRNA2, psiRNA3, pconsiRNA(图1), 结果显示与预期完全一致, 测序结果进一步显示目的片段插入psiRNA-hH1neo载体中, 所测序列与合成序列吻合(测序结果未显示).
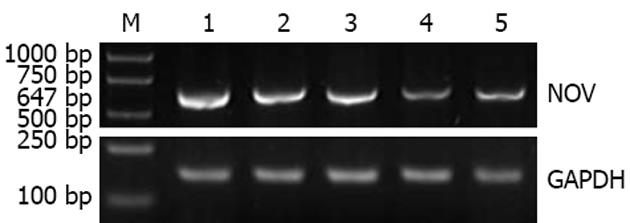
RT-PCR检测NOV mRNA表达, 经计算机灰度扫描及与内参比较分析显示, 与空白对照组相比, 在转染psiRNA1, psiRNA2, psiRNA3, pconsiRNA后48 h HSC内NOV mRNA分别下降37.8%, 73.0%, 42.5%, 23.2%. 与阴性对照组相比, psiRNA2组内NOV mRNA明显下降(P<0.05), 而psiRNA1组、psiRNA3组内NOV mRNA下降不明显(P>0.05)(图2).
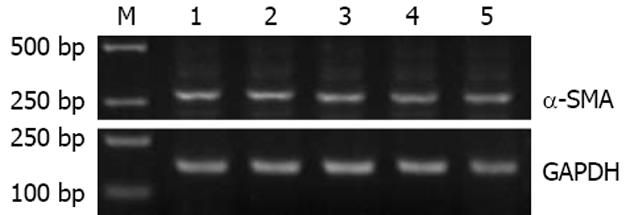
RT-PCR及Western blot结果经计算机灰度扫描并与内参照(GAPDH)比较分析显示, 与空白对照组相比, 在转染psiRNA1, psiRNA2, psiRNA3, pconsiRNA后48 h HSC内α-SMA mRNA分别下降25.7%, 51.4%, 27.2%, 15.1%(图3). 转染psiRNA2组α-SMA蛋白表达较空白对照组下降40%(图4). 与阴性对照组相比, psiRNA2组内α-SMA mRNA明显下降(P<0.05), 而psiRNA1组、psiRNA3组内α-SMA mRNA下降不明显(均P>0.05).
转染psiRNA2组细胞增殖活性显著低于空白对照组(P<0.05), 且在转染后24 h时siRNA2抑制细胞增殖活性的能力最强; 转染pconsiRNA、psiRNA1和psiRNA3组细胞增殖活性与空白对照组相比没有显著改变(均P>0.05)(表3).
流式细胞术分析表明, 与空白对照组相比, 转染pconsiRNA, psiRNA1, psiRNA3和psiRNA2的HSC凋亡均不显著.
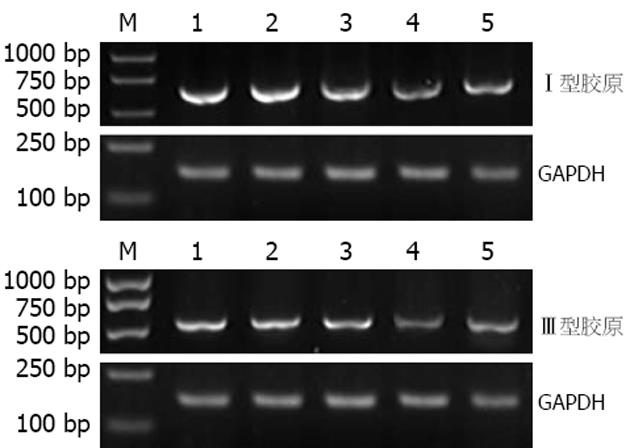
RT-PCR结果经计算机灰度扫描并与内参照(GAPDH)比较分析显示, 与空白对照组相比, 在转染psiRNA1, psiRNA2, psiRNA3, pconsiRNA后48 h Ⅰ型胶原mRNA分别下降19.4%, 59.8%, 34.1%, 17.0%, Ⅲ型胶原mRNA分别下降17.2%, 37.1%, 9.2%, 6.6%. 与阴性对照组相比, psiRNA2组内Ⅰ, Ⅲ型胶原mRNA明显下降(P<0.05), 而psiRNA1组、 psiRNA3组内Ⅰ, Ⅲ型胶原mRNA下降不明显(均P>0.05)(图5).
肝纤维化是慢性肝病发展到肝硬化的必经阶段, 在致病因素消除后可以发生逆转[7], 其发病机制的研究是肝病攻关的热点课题之一. HSC在肝纤维化发生、发展中起主导作用. 在各种致病因素作用下, 静止期HSC被激活并增殖, 转化为肌成纤维样母细胞, 合成和分泌ECM上调, 同时合成和释放大量的基质金属蛋白酶抑制因子, 使间质胶原酶等蛋白酶活性降低, ECM降解减少, 最终导致ECM积聚而发生肝纤维化乃至肝硬化[1].
NOV是一种低亲和力胰岛素样生长因子结合蛋白, 其效应有胰岛素样生长因子依赖性和胰岛素样生长因子非依赖性[8]. 目前研究发现NOV可调节细胞内钙离子水平[9-10], 并对某些肿瘤有抑制生长作用. Lee et al[4]研究发现在活化的HSC中NOV基因表达上升. Northern analysis表明NOV mRNA在培养的鼠HSC中表达上升. 并观察发现在CCL4诱导的及胆管结扎导致的鼠肝纤维化模型中NOV mRNA上调, RT-PCR表明与正常肝组织相比, 人NOV mRNA在纤维化的肝组织中增加. 在人或鼠的肝纤维化中NOV蛋白的表达主要位于导管增生区或纤维隔HSC中. 但NOV在活化的HSC中的作用尚不明了.
RNAi现象是指内源性或外源性双链RNA (dsRNA)介导细胞内的mRNA发生特异性降解, 导致靶基因的表达沉默, 产生相应的功能表型缺失[5]. RNAi具有自我扩增性, 能够在细胞间传代, 甚至将效应传到下一代[11]. 最近研究表明, 无论在体内还是体外RNAi比反义寡核苷酸更具有优势效应[12-13].
早期siRNA多为化学合成, 目前的研究中较多的使用基于载体表达的方法制备siRNA. 根据研究物种的不同, 可选用特异的RNA多聚酶识别的启动子, 目前已用于哺乳动物RNAi研究的载体常用的启动子有T7, H1, U5, U6启动子等. 这些表达载体, 在瞬时稳定地转染了哺乳动物细胞后, 可以持续的表达siRNA. 这一类基于载体制备的siRNA能够在细胞内持续表达, 因此可以比直接转染siRNA更持久[14-16]. 可将插入序列设计成含基因特异序列的反转重复序列, 他在体内表达后自发形成小发卡RNA(smallhairpinRNAs, shRNA), shRNA继而被加工成siRNA样分子, 执行基因特异的沉默[17].本实验构建了针对NOV的siRNA的真核表达载体, 并把其转染入HSC细胞内, 结果证实这些载体可以在转染的HSC中高效表达, RT-PCR结果显示siRNA2抑制靶基因表达的效果最明显, 而siRNA1和siRNA3抑制效果不明显, 这可能是因为靶基因上siRNA作用位点选择不当, 并且不是所有的siRNA都能够接近靶序列RNA, 有些序列包埋于靶RNA的二级结构或折叠区域, 而另一些则与蛋白质形成紧密的复合物, 阻碍了对靶基因序列的识别[18].
肝星状细胞活化过程中伴随着α-平滑肌肌动蛋白表达增加[19], 检测α-平滑肌肌动蛋白表达是鉴定HSC激活或判定HSC活化状态的一种有效方法. 本实验中转染psiRNA2的HSC与转染其他质粒的相比α-SMA的mRNA及蛋白表达明显下降, 表明siRNA2能明显抑制HSC的活化, 由此可推论NOV在HSC的活化中可以起一定的促进作用.
Ⅰ型胶原及Ⅲ型胶原是细胞外基质的主要成分. 胶原蛋白在正常肝脏内约占蛋白总量的5%-20%, 肝纤维化时可增加至50%左右. 肝纤维化时发现有5种主要胶原蛋白, 其中以Ⅰ、Ⅲ型为主, 肝纤维化早期以Ⅲ型为主, 后期以Ⅰ型为主[20-21]. 在肝纤维化过程中Ⅰ、Ⅲ型胶原是肝纤维间隔的重要来源. RT-PCR结果显示siRNA2沉默NOV基因对大鼠HSCⅠ型胶原、Ⅲ型胶原mRNA表达有明显抑制作用. 这表明NOV基因可促进活化的HSC表达细胞外基质.
Maillard et al[22]研究发现, 在前列腺细胞转化过程, 随着NOV表达增多, 细胞的增殖能力增强. Glukhova et al[23]在肾细胞癌的研究中发现随着NOV表达上升, 癌细胞的分级增高, 并且在SCID小鼠中的成瘤能力增强. 本实验MTT结果证实了siRNA2降低HSC增殖活性, 表明NOV基因能增强HSC增殖能力. 但Gupta et al[24]研究发现在G59神经胶质瘤细胞系中导入NOV基因降低了细胞的增殖能力和致瘤性. 同时NOV基因表达在不同类型的组织中可以不同, 如在骨肉瘤中表达升高, 在尤文氏肉瘤中不确定, 而在横纹肌肉瘤中则降低[25], 在尤文氏肉瘤中随着NOV表达增多, 细胞的增殖能力减弱, 但转移及侵袭性增强[26]. 这些研究结果表明NOV的生物学效应也许与效应细胞种类有关. 本实验表明NOV可促进HSC的活化及增殖, 增加Ⅰ型胶原、Ⅲ型胶原mRNA的表达, NOV可以作为肝纤维化基因治疗的一个靶位点.
肝纤维化是慢性肝病发展到肝硬化的必经阶段, 其发病机制的研究是肝病攻关的热点课题之一. 而肝星状细胞在肝纤维化发生、发展中起主导作用. 对肝星状细胞活化过程中相关基因表达的变化进行研究可以有助于更好了解肝纤维化的致病机制, 为肝纤维化新的治疗方法提供理论依据.
肾母细胞瘤过度表达基因(NOV)表达产物是一种低亲和力胰岛素样生长因子结合蛋白, 在正常HSC中无表达, 近来研究发现在活化的HSC中NOV基因表达, 并且随着HSC活化程度的上升NOV表达上升, 但NOV在活化的HSC中的作用尚不明了. 需要进一步研究.
肝星状细胞活化在肝纤维化过程起主导作用. 肝星状细胞活化、凋亡相关基因是目前肝纤维化研究中的热点. 本文研究了NOV与肝星状细胞生物学性状之间的关系, 发现NOV可促进肝星状细胞的活化, 增强肝星状细胞的增殖能力, 增加Ⅰ型胶原、Ⅲ型胶原mRNA的表达.
本研究结果表明肾母细胞瘤过度表达基因(NOV)能促进肝星状细胞的活化、增殖, 增加细胞外基质的表达, 表明NOV基因在肝纤维化发展中起促进作用. 本实验为NOV在肝星状细胞中作用机制的进一步研究提供了实验依据, 为肝纤维化的研究提供了一条新思路, 同时为肝纤维化的基因治疗提供了新靶点.
肾母细胞瘤过度表明基因(NOV): 人NOV基因定位于常染色体8q24.1, 其编码产物是一种低亲和力胰岛素样生长因子结合蛋白, 属于CCN家族的成员, 目前研究发现其能调节细胞内钙离子水平, 并能抑制某些肿瘤的生长.
本文研究了肾母细胞瘤过度表达基因对大鼠肝星状细胞生物学行为的影响, 内容新颖, 具有一定的意义.
电编: 张敏 编辑:张焕兰
| 1. | Safadi R, Friedman SL. Hepatic fibrosis-role of hepatic stellate cell activation. MedGenMed. 2002;4:27. [PubMed] |
| 2. | Kim HS, Nagalla SR, Oh Y, Wilson E, Roberts CT Jr, Rosenfeld RG. Identification of a family of low-affinity insulin-like growth factor binding proteins (IGFBPs): characterization of connective tissue growth factor as a member of the IGFBP superfamily. Proc Natl Acad Sci U S A. 1997;94:12981-12986. [PubMed] [DOI] |
| 3. | Brigstock DR. The connective tissue growth factor/cysteine-rich 61/nephroblastoma overexpressed (CCN) family. Endocr Rev. 1999;20:189-206. [PubMed] [DOI] |
| 4. | Lee SH, Seo GS, Park YN, Sohn DH. Nephroblas-toma overexpressed gene (NOV) expression in rat hepatic stellate cells. Biochem Pharmacol. 2004;68:1391-1400. [PubMed] [DOI] |
| 7. | Friedman SL. Molecular mechanisms of hepatic fibrosis and principles of therapy. J Gastroenterol. 1997;32:424-430. [PubMed] [DOI] |
| 8. | Joliot V, Martinerie C, Dambrine G, Plassiart G, Brisac M, Crochet J, Perbal B. Proviral rearrange-ments and overexpression of a new cellular gene (nov) in myeloblastosis-associated virus type 1-induced nephroblastomas. Mol Cell Biol. 1992;12:10-21. [PubMed] [DOI] |
| 9. | Li CL, Martinez V, He B, Lombet A, Perbal B. A role for CCN3 (NOV) in calcium signalling. Mol Pathol. 2002;55:250-261. [PubMed] [DOI] |
| 10. | Lombet A, Planque N, Bleau AM, Li CL, Perbal B. CCN3 and calcium signaling. Cell Commun Signal. 2003;1:1. [PubMed] [DOI] |
| 11. | Paroo Z, Corey DR. Challenges for RNAi in vivo. Trends Biotechnol. 2004;22:390-394. [PubMed] [DOI] |
| 12. | Bertrand JR, Pottier M, Vekris A, Opolon P, Maksimenko A, Malvy C. Comparison of antisense oligonucleotides and siRNAs in cell culture and in vivo. Biochem Biophys Res Commun. 2002;296:1000-1004. [PubMed] [DOI] |
| 13. | Aoki Y, Cioca DP, Oidaira H, Kamiya J, Kiyosawa K. RNA interference may be more potent than antisense RNA in human cancer cell lines. Clin Exp Pharmacol Physiol. 2003;30:96-102. [PubMed] [DOI] |
| 14. | Sui G, Soohoo C, Affar el B, Gay F, Shi Y, Forrester WC, Shi Y. A DNA vector-based RNAi technology to suppress gene expression in mammalian cells. Proc Natl Acad Sci U S A. 2002;99:5515-5520. [PubMed] [DOI] |
| 15. | Brummelkamp TR, Bernards R, Agami R. A system for stable expression of short interfering RNAs in mammalian cells. Science. 2002;296:550-553. [PubMed] [DOI] |
| 16. | Paddison PJ, Caudy AA, Hannon GJ. Stable suppression of gene expression by RNAi in mammalian cells. Proc Natl Acad Sci U S A. 2002;99:1443-1448. [PubMed] [DOI] |
| 17. | Yu JY, DeRuiter SL, Turner DL. RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells. Proc Natl Acad Sci U S A. 2002;99:6047-6052. [PubMed] [DOI] |
| 18. | Yoshinari K, Miyagishi M, Taira K. Effects on RNAi of the tight structure, sequence and position of the targeted region. Nucleic Acids Res. 2004;32:691-699. [PubMed] [DOI] |
| 19. | Rockey DC, Boyles JK, Gabbiani G, Friedman SL. Rat hepatic lipocytes express smooth muscle actin upon activation in vivo and in culture. J Submicrosc Cytol Pathol. 1992;24:193-203. [PubMed] |
| 20. | Van Eyken P, Sciot R, Desmet VJ. Expression of the novel extracellular matrix component tenascin in normal and diseased human liver. An immunohistochemical study. J Hepatol. 1990;11:43-52. [PubMed] [DOI] |
| 21. | Gulubova MV, Stoyanov HD, Julianov AE, Vasilev IV, Stoyanova II, Hadjipetkov PB. Immunohisto-chemical detection of collagen type III and IV in relation with transformation of Ito cells in liver sinusoids of patients with reactive biliary hepatitis. Acta Histochem. 1999;101:213-228. [PubMed] [DOI] |
| 22. | Maillard M, Cadot B, Ball RY, Sethia K, Edwards DR, Perbal B, Tatoud R. Differential expression of the ccn3 (nov) proto-oncogene in human prostate cell lines and tissues. Mol Pathol. 2001;54:275-280. [PubMed] [DOI] |
| 23. | Glukhova L, Angevin E, Lavialle C, Cadot B, Terrier-Lacombe MJ, Perbal B, Bernheim A, Goguel AF. Patterns of specific genomic alterations associated with poor prognosis in high-grade renal cell carcinomas. Cancer Genet Cytogenet. 2001;130:105-110. [PubMed] [DOI] |
| 24. | Gupta N, Wang H, McLeod TL, Naus CC, Kyurkchiev S, Advani S, Yu J, Perbal B, Weichselbaum RR. Inhibition of glioma cell growth and tumorigenic potential by CCN3 (NOV). Mol Pathol. 2001;54:293-299. [PubMed] [DOI] |
| 25. | Manara MC, Perbal B, Benini S, Strammiello R, Cerisano V, Perdichizzi S, Serra M, Astolfi A, Bertoni F, Alami J. The expression of ccn3(nov) gene in musculoskeletal tumors. Am J Pathol. 2002;160:849-859. [PubMed] [DOI] |