修回日期: 2006-01-09
接受日期: 2006-01-14
在线出版日期: 2006-02-28
目的: 筛选HCV p7病毒蛋白反式调节的新靶基因p7TP3, 克隆p7TP3基因及其不同剪切体基因序列, 并进行功能分析, 探讨丙型肝炎病毒(HCV)p7病毒蛋白在HCV致病过程中的作用.
方法: 依据我们构建的真核表达载体pcDNA 3.1(-)-p7转染肝母细胞瘤细胞系HepG2, 提取总RNA, 逆转录为cDNA后进行基因表达谱芯片分析, 利用生物信息学技术获得新基因p7TP3及其可变剪切体的编码序列, 设计特异性引物, 并对其进行克隆化研究.
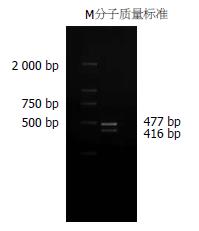
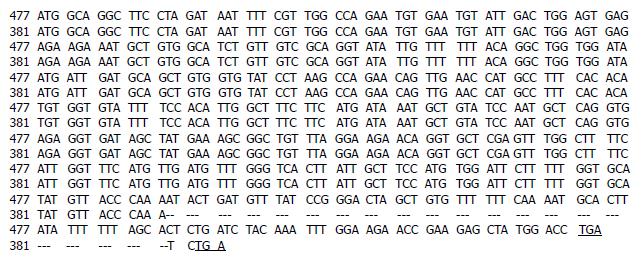
结果: 经测序鉴定获得新基因p7TP3的编码序列, 并发现了p7TP3的不同剪切体, 对p7TP3基因组进行分析, 获得剪切体的编码序列, 并进行了克隆化研究及生物信息学分析. p7TP3(416 bp)编码蛋白质序列比p7TP3(477 bp)少TQNTDVYPGLAVFFQNALIFFSTLIY 26个氨基酸残基, 其余序列相同.
结论: HCV p7蛋白是一种典型的病毒基因组编码的具有反式调节作用的蛋白. 利用分子生物学技术与生物信息学分析, 发现并鉴定了HCV p7TP3反式调节作用的新的靶基因p7TP3及其剪切体, 为阐明HCV p7蛋白的反式调节作用及其机制开辟了新的研究方向.
引文著录: 陶明亮, 成军, 王春花, 郭江, 袁菊, 靳亚平, 毛羽. HCV p7蛋白反式调节新基因3及其剪切体克隆化和生物信息学分析. 世界华人消化杂志 2006; 14(6): 576-580
Revised: January 9, 2006
Accepted: January 14, 2006
Published online: February 28, 2006
AIM: To identify and clone a new gene p7TP3 and its spliced variant transregulated by hepa-titis C virus p7 protein, and to explore the role of HCV p7 protein in the pathogenesis of HCV infection.
METHODS: Based on the construction of expressive vector pcDNA3.1(-)-p7, hepatoblastoma cells HepG2 were transfected with plasmid DNA of pcDNA3.1(-)-p7, and then the total RNA was extracted from the cells. Reverse transcribed cDNA was subjected for microarray assay. The coding sequence of the new gene and its spliced variant were obtained by bioinformatic methods. Polymerase chain reaction (PCR) was conducted to amplify p7TP3 gene and its spliced variant using specific primer. The structure was detected and the function was predicted by bioinformatics methods.
RESULTS: Sequence analysis confirmed that the coding sequence of p7TP3 gene and its spliced variant were cloned successfully and bioinformatically analyzed. p7TP3 (416 bp) with Mr 14 165.4 is identical to p7TP3 (477 bp) with Mr 17 935.7 except a deletion of 26 amino acid residue (TQNTDVYPGLAVFFQNALIFFSTLIY).
CONCLUSION: HCV p7 protein is a potential transregulator. A novel gene and its spliced variant have been identified as the new target transregulated by HCV p7 protein.
- Citation: Tao ML, Cheng J, Wang CH, Guo J, Yuan J, Jin YP, Mao Y. Cloning and bioinformatic analysis of new gene p7TP3 and its spliced variant transregulated by hepatitis C virus p7 protein. Shijie Huaren Xiaohua Zazhi 2006; 14(6): 576-580
- URL: https://www.wjgnet.com/1009-3079/full/v14/i6/576.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i6.576
丙型肝炎病毒(HCV)是含外膜蛋白的单股正链RNA病毒, 属黄病毒科肝炎病毒类. 全世界范围内约有1.7亿人被感染, 在大多数感染人群中HCV表现为持续性感染, 并可导致肝纤维化(LC), 甚至肝细胞癌(HCC)[1]. HCV基因组编码一个约3 010个氨基酸残基组成的多蛋白, 被病毒和细胞蛋白酶切割产生至少10个病毒产物[2-3]. p7蛋白是存在于HCV结构蛋白和非结构蛋白之间的63个氨基酸残基(aa)组成的一个小蛋白, 他在脂质膜中形成六聚体阳离子通道, 研究发现金刚烷胺和长烷基链的亚氨基糖衍生物可抑制p7形成的阳离子通道, 从而对HCV的治疗发挥重要作用[4-7]. 我们利用基因芯片技术, 对p7表达载体转染的肝母细胞瘤细胞系HepG2的基因表达谱变化进行了比较, 发现了p7蛋白可以反式调节一些基因的表达. 其中包括未知功能基因, 命名为p7TP3. 我们在研究新基因p7TP3过程中, 意外发现了p7TP3基因的不同剪切体, 对p7TP3基因组进行分析, 确定了p7TP3及其剪切体的编码序列, 并进行了克隆化研究和生物信息学分析.
肝母细胞瘤细胞系HepG2和感受态大肠杆菌DH5α为本室保存, Taq酶购自Promega公司, pGEM-T载体购自Promega公司. 玻璃奶回收试剂盒购自博大公司. DNA序列测定由北京奥科公司完成. 利用基因芯片技术对p7蛋白的上调、下调基因进行分析, 结果表明, 1种基因的表达水平显著上调, 22种基因的表达水平显著下调, 下调基因中含有2个未知功能的新基因, 利用美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)及其同源基因序列的搜索(BLASTN)发现这两个新基因与GenBank中注册的已知功能基因序列没有同源性, 依据GenBank数据库EST中的同源基因片段进行电子拼接, 根据Kozak规则和终止密码子下游的多聚腺苷酸信号序列, 确定完整的编码基因序列, 分别命名为HCV p7TP2(p7蛋白反式调节蛋白2)、p7TP3.
根据p7TP3的全长编码基因, 设计上下游引物. 上游引物5'-GAA TTC CA ATG GCA GGC TTC CTA GA-3', 下游引物5'-GGA TCC GGT CCA TAG CTC TTC GGT TC-3'. 以反转录产物为模板进行PCR, PCR参数如下: 95 ℃ 5 min预变性, 94 ℃ 30 s变性, 58 ℃ 45 s退火, 72 ℃ 90 s延伸, 共35个循环, 72 ℃再延伸10 min. 克隆目的片段将PCR产物在10 g/L琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与pGEM-T载体连接, 转化DH5α感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养16 h. 挑取白色菌落, 增菌. 提取质粒进行限制性酶切分析鉴定发现两个大小不一致的酶切片段, 挑选相应菌落测序. 将测序得到的两个核苷酸序列进行分析, 其中一个与我们预测的p7TP3序列完全一致, 而另一个序列与p7TP3部分同源. 利用美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)进行搜索, 确定了两个序列的染色体定位并对其编码的蛋白质功能进行了预测.
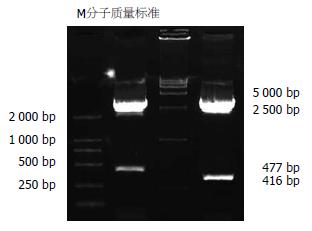
以HepG2细胞系cDNA文库为模板, PCR反应后经10 g/L琼脂糖凝胶电泳可见长度为477 bp和416 bp的两种电泳条带(图1). PCR产物与T-载体连接, 转化大肠杆菌, 提取质粒进行酶切鉴定(图2).
2.2 将p7TP3(477 bp)与p7TP3(416 bp)编码序列进行比较(图3).
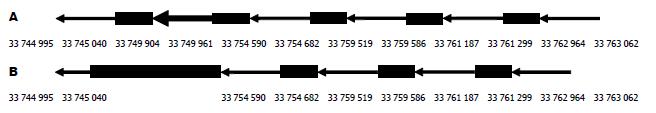
NCBI中BLASTN核酸序列同源性分析以及基因组序列的内含子⁄外显子分析, (http://www.ens-embl.org/index.html)在线分析p7TP3及其剪切体在染色体中定位以及内含子⁄外显子位点分析. 发现二者均定位于21号染色体, 但其内含子和外显子结构不同, p7TP3(477 bp)与p7TP3(416 bp)基因定位于33 763 062-33 744 995 nt, p7TP3(416 bp)基因与p7TP3(477 bp)相比缺少33749961- 33749904的外显子序列.
经Protparam tool在线软件分析, p7TP3(477 bp)基因编码的蛋白的Mr为17 935.7, 理论pⅠ5.23; p7TP3(416 bp)基因编码的蛋白的Mr为14 165.4, 理论pⅠ5.51. p7TP3(477 bp)与p7TP3(416 bp)编码蛋白质均在19-21SeR具有潜在的蛋白激酶C磷酸化位点; 在62-67GVfsTL、108-113GSliAS、119-124GAyvTQ具有潜在的N-豆蔻酰基化位点; 在81-83RGD具有潜在的细胞黏附序列位点. 应用ProtScale软件对二者的编码蛋白质分别进行了α螺旋、β折叠和无规则卷曲等二级结构预测, 发现他们除在羧基末端稍有差异外, 其蛋白质二级结构基本相同. 经Predicting Antigenic Peptides(http://bio.dfci.edu/harvardTools/antigenic.pl)预测蛋白质抗原决定簇, p7TP3(416 bp)编码蛋白质抗原决定簇的No7序列比p7TP3(477 bp)少TQNTDVYPGLAVFFQNALIFFSTLIY 26个氨基酸残基, 其余序列相同(表1).
| No | Start Position | Sequence | End Position |
| 1 | 10 | WPECECID | 17 |
| 2 | 23 | NAVASVVAGILFF | 35 |
| 3 | 41 | MIDAAVVYPK | 50 |
| 4 | 53 | QLNHAFHTCGVF | 72 |
| STLAFFMI | |||
| 5 | 85 | YESGCLG | 91 |
| 6 | 95 | ARVWLFIGF | 103 |
| 7 | 106 | MFGSLIASMWILFGA | 148 |
| YVTQNTDVYPGLAV | |||
| FFQNALIFFSTLIY |
病毒基因组的复制和表达调节与真核细胞类似, 主要是转录水平的调节. 然而在许多情况下病毒还必须借助感染的宿主细胞的一些复制和表达的辅助元件完成其生活周期及致病过程[8]. 在细胞的不同发育阶段和生理状态下, 基因通过不同的拼接方式产生不同的mRNA和翻译产物, 使得细胞所表达的基因类型、表达水平、表达组织和细胞特异性、表达的先后顺序都是不同的. 基因可通过如下几种方式产生多个转录体如不同的转录起始位点, 可变剪切, 选择不同的加尾信号位点, RNA编辑等. 多种生物中存在可变剪切现象[9-11], 可变剪切主要有四种方式: (1)剪切产物缺失一个或几个外显子; (2)剪切产物保留一个或几个内含子作为外显子的编码序列; (3)外显子中存在5'-剪切位点或3'-剪切位点; (4)内含子中存在5'-剪切位点或3'-剪切位点, 从而使部分内含子变为编码序列. RNA剪切是真核生物在长期进化过程中形成的, 他也是基因表达调节的重要环节[12-14].
随着以人的基因克隆化为主的不同生物类型基因克隆化研究的进展, 已经积累了大量的不同生物的基因序列、蛋白质的氨基酸残基序列, 并据此建立了庞大的数据库系统. 对于这些数据的分析, 必须依靠生物信息学技术不断发展, 为这些资料和数据的分析建立了一些有效平台. 对于一种新基因生物学和医学意义的认识, 可以从多方面入手, 其中对于这种蛋白的表达及其在细胞中对于基因表达谱的影响研究, 也是重要的突破口. 从目前的研究技术来看, 以基因芯片技术和抑制性消减杂交(SSH)技术最为有效. 来源于同一个基因的剪切体有时具有相反的生物学功能, 成军 et al[15]利用基因芯片技术分别对于NS5ATP2(615 bp)与NS5ATP2(216 bp)反式调节的肝细胞的靶基因类型进行分析, 然后对于不同剪切型NS5ATP2(615 bp)与NS5ATP2(216 bp)反式调节基因类型的异同进行比较分析, 研究不同剪切型的生物学功能差别, 发现他们的生物学功能具有相同或相似的一面. 我们在克隆p7TP3基因的cDNA过程中, 发现了一个p7TP3(477 bp)的mRNA剪切变异体p7TP3(416 bp), 经生物信息学分析, 我们发现p7TP3(477 bp)与p7TP3(416 bp)编码的蛋白质均是跨膜蛋白, 推测其亚细胞定位可能在胞质膜; 使用ProtScale软件对他们编码的蛋白质分别进行了α螺旋、b折叠和无规则卷曲等二级结构预测, Predicting Antigenic Peptides软件预测他们编码蛋白质抗原决定簇, 发现他们的生物学功能非常相似, 但是这种生物信息学分析有其局限性, 还必须以实际的研究结果进一步证实.
从分子生物学角度来看, 丙型肝炎病毒发病机制就是肝炎病毒生物大分子与肝细胞相互作用及其后果, 主要内容可以分成3个层次: (1)肝细胞中调节肝炎病毒复制和表达的蛋白质因子及相应的信号转导; (2)肝炎病毒蛋白与肝细胞中蛋白之间的相互作用以及后果; (3)肝炎病毒蛋白的表达对于肝细胞基因表达谱和表达水平的反式调节作用. 其中丙型肝炎病毒的反式调节机制在其肝炎病毒发病机制中发挥了重要作用. 反式调节机制至少包括三方面的含义[16-23]: 一方面是肝炎病毒蛋白对于肝细胞基因组表达调节的反式调节即肝炎病毒蛋白与肝细胞基因组启动子DNA结合对于肝细胞基因表达谱产生影响; 第二方面是肝细胞蛋白对于肝炎病毒基因表达的反式调节. 肝炎病毒属于简单的生物类型, 要完成其生活周期必须借助于肝细胞中的蛋白成分, 肝细胞中特有的一些蛋白成分与肝炎病毒基因组DNA/RNA结合对于肝炎病毒基因表达进行调节, 这也是决定肝炎病毒嗜肝特性的重要的分子生物学机制; 第三方面是肝炎病毒蛋白对于肝炎病毒基因组表达的反式调节. 目前关于p7蛋白反式调节的靶基因的结构与功能、表达与调控以及新基因在丙型肝炎病毒致病机制中的作用不明, 但是我们相信, 新基因p7TP3及其剪切体的克隆为研究p7蛋白的生物学功能开辟了新的方向.
p7蛋白是丙型肝炎病毒中介于结构蛋白和非结构蛋白之间的一小蛋白, 由于他在脂质膜上可形成六聚体阳离子通道, 在病毒的自然感染过程中起一定的作用, 然而其具体作用机制尚不清楚. 研究p7蛋白及其相关基因的反式调节机制可能是解决HCV感染的有效途径之一.
本文利用PCR和生物信息学技术分析了p7TP3及其剪切体的基因组序列、编码蛋白的一、二级结构、抗原决定簇等生物学功能差异, 但他们之间的功能差异仍需进一步验证.
新基因p7TP3及其剪切体的克隆和功能比较为阐明p7蛋白的生物学功能及其在HCV感染途径的作用开辟了新的方向.
可变剪切: 由于mRNA的编辑产生了不同的多肽, 进而形成不同蛋白质, 这些蛋白质就互称为剪切体(splice variants)或者可变剪切.
文章发现并鉴定HCV p7TP3(477 bp)及其剪切体p7TP3(416 bp)基因, 为阐明HCV p7蛋白的反式调节作用及其机制开辟了新的研究方向. 该论文具有科学性、创新性和可行性, 能较好反映我国消化病学实验研究的先进水平.
电编: 张敏 编辑:潘伯荣
| 1. | 成 军, 杨 守纯. 现代肝炎病毒分子生物学. 第1版. 北京: 人民军医出版社 1997; 83-88. |
| 2. | Nielsen SU, Bassendine MF, Burt AD, Bevitt DJ, Toms GL. Characterization of the genome and structural proteins of hepatitis C virus resolved from infected human liver. J Gen Virol. 2004;85:1497-1507. [PubMed] |
| 4. | Carrere-Kremer S, Montpellier C, Lorenzo L, Brulin B, Cocquerel L, Belouzard S, Penin F, Dubuisson J. Regulation of hepatitis C virus polyprotein process-ing by signal peptidase involves structural determi-nants at the p7 sequence junctions. J Biol Chem. 2004;279:41384-41392. [PubMed] |
| 5. | Carrere-Kremer S, Montpellier-Pala C, Cocquerel L, Wychowski C, Penin F, Dubuisson J. Subcellular localization and topology of the p7 polypeptide of hepatitis C virus. J Virol. 2002;76:3720-3730. [PubMed] |
| 6. | Ghibaudo D, Cohen L, Penin F, Martin A. Charac-terization of GB virus B polyprotein processing reveals the existence of a novel 13-kDa protein with partial homology to hepatitis C virus p7 protein. J Biol Chem. 2004;279:24965-24975. [PubMed] |
| 8. | 成 军, 刘 妍, 洪 源, 王 琳, 钟 彦伟, 董 菁, 王 刚. 丙型肝炎病毒非结构蛋白5A反式激活基因10的克隆化研究. 世界华人消化杂志. 2003;11:935-938. [DOI] |
| 10. | Slaugenhaupt SA. Genetics of familial dysautono-mia. Tissue-specific expression of a splicing muta-tion in the IKBKAP gene. Clin Auton Res. 2002;12:I15-I19. [PubMed] |
| 11. | Gelfand MS, Dubchak I, Dralyuk I, Zorn M. ASDB: database of alternatively spliced genes. Nucleic Acids Res. 1999;27:301-302. [PubMed] |
| 12. | 王 镜岩, 朱 圣庚, 徐 长法. 生物化学(下册). 第3版. 北京: 高等教育出版社 2002; 484-485. |
| 13. | Stiles JK, Hicock PI, Shah PH, Meade JC. Genomic organization, transcription, splicing and gene regu-lation in Leishmania. Ann Trop Med Parasitol. 1999;93:781-807. [PubMed] |
| 14. | O'Neill JP, Rogan PK, Cariello N, Nicklas JA. Mutat-ions that alter RNA splicing of the human HPRT gene: a review of the spectrum. Mutat Res. 1998;411:179-214. [PubMed] |
| 16. | You LR, Chen CM, Yeh TS, Tsai TY, Mai RT, Lin CH, Lee YH. Hepatitis C virus core protein interacts with cellular putative RNA helicase. J Virol. 1999;73:2841-2853. [PubMed] |
| 19. | 王 建军, 刘 妍, 成 军, 杨 倩, 纪 冬, 党 晓燕, 王 春花. 丙型肝炎病毒非结构蛋白5A反式激活基因6的反式激活基因的筛选. 世界华人消化杂志. 2004;12:54-57. [DOI] |
| 21. | 成 军, 刘 妍, 洪 源, 王 建军, 杨 倩, 王 琳. 乙型肝炎病毒和丙型肝炎病毒反式调节靶基因的抑制性消减杂交和基因芯片分析结果的比较. 世界华人消化杂志. 2004;12:327-331. [DOI] |