修回日期: 2006-10-28
接受日期: 2006-11-10
在线出版日期: 2006-12-28
目的: 探讨中国汉族人群中肿瘤抗原递呈的限速基因LMP2/LMP7单核苷酸多态性与胃癌的遗传易感性关联.
方法: 提取145例胃癌患者及152例健康献血员外周血基因组DNA, 用PCR-RFLP方法检测两组人群中LMP2-codon60/LMP7-codon145两个多态性位点的基因型; 并采用PHASE1.0软件构建这两个多态性位点的个体单倍体型, 以非条件Logistic回归校正混杂因素, 并进行多态性与胃癌患者风险关联的统计学分析.
结果: PCR-RFLP检测结果显示: LMP2-codon60与LMP7-codon145处的多态性在中国人群中普遍存在, LMP2/LMP7多态性位点的各自的等位基因频率分布符合Hardy-Weinberg平衡定律; 最后的统计分析表明: 在145例胃癌患者与152例健康对照人群中LMP2/LMP7两个多态性位点的基因型频率、以及所构建的4个单倍体型频率均没有统计学差异(P值均大于0.05).
结论: 在中国汉族人群中, LMP2/LMP7基因编码区两个位点的多态性以及单倍体型与胃癌易感无相关性.
引文著录: 曹邦伟, 戴悦, 盖郁慧, 柴庆波, 汪欣. LMP2/LMP7基因编码区多态性及单倍体型与胃癌易感无相关性. 世界华人消化杂志 2006; 14(36): 3471-3476
Revised: October 28, 2006
Accepted: November 10, 2006
Published online: December 28, 2006
AIM: To elucidate whether LMP2/LMP7 poly-morphisms or haplotypes were associated with the occurrence of gastric cancer in Han Chinese.
METHODS: The genome DNA was extracted from the peripheral blood of 145 gastric cancer patients and 152 cancer-free controls, and the LMP2-codon60/LMP7-codon145 genotypes were detected by polymerase chain reaction- restriction fragment length polymorphism (PCR-RFLP). The software PHASE1.0 was used to construct the haplotypes of each individual. The unconditional Logistic regression model was applied to analyze the statistical differences of genotypes or haplotypes between the two groups.
RESULTS: PCR-RFLP showed that the polymorphisms were found at both codon60 and codon145 loci of LMP2/LMP7 gene in 297 Chinese individuals, and chi-square test demonstrate that the distributions of LMP2/LMP7 genotypes were in Hardy-Weinberg equilibrium. After statistical analysis, we found the frequencies of LMP2/LMP7 gene polymorphisms and the four haplotypes were not significantly different between gastric cancer patients and cancer-free controls (all P > 0.05).
CONCLUSION: The polymorphisms and haplotypes of LMP2-codon60/LMP7-codon145 gene may not contribute to the genetic susceptibility of gastric cancer in the Han nationality of China.
- Citation: Cao BW, Dai Y, Gai YH, Chai QB, Wang X. No association between LMP2/LMP7 gene polymorphisms or haplotypes and gastric cancer. Shijie Huaren Xiaohua Zazhi 2006; 14(36): 3471-3476
- URL: https://www.wjgnet.com/1009-3079/full/v14/i36/3471.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i36.3471
低分子量蛋白酶体(low molecular weight proteasome, LMP)是于1982年Monaco[1]在分析鼠MHC-Ⅱ类分子的免疫沉淀复合物时得到的产物. 在机体内肿瘤细胞的免疫清除中, 其作用在于能够水解肿瘤抗原成为适合的肽段, 经转运后于与MHC-Ⅰ类分子连接, 并表达于细胞表面为CD8+ T淋巴细胞所识别, 从而清除异己的肿瘤细胞. 因此LMP对于肿瘤细胞的抗原免疫应答有着极为重要的作用[2]. 他包括LMP2和LMP7两个基因, 分别编码产生LMP2与LMP7两种蛋白质产物, 组成一种非溶酶体的蛋白水解酶复合体. LMP2/LMP7基因多态性的重要性在于能够改变其水解后肽段的长度或者裂解片段的模式, 从而能够有利或者不利于与MHC-Ⅰ类分子连接, 继而激发机体对肿瘤细胞的免疫清除[3-4], 因此LMP被认为是肿瘤易感性重要的候选基因. 目前已有研究表明, LMP基因的多态性与肿瘤或者一系列免疫相关性疾病发生有着密切关联[5-8], 但目前国内外的研究尚未有与胃癌易感性关联的报道, 为此我们选取LMP2/LMP7基因中具有氨基酸改变的两个多态性位点, 对中国汉族正常人群及胃癌患者进行了其多态性位点的检测, 并同时进行了易感性的关联分析.
病例组选自2003-05/2005-07在北京大学血液标本库(北大医院医院输血科)留存的胃癌血液样本145例, 其中男82例, 女63例, 平均年龄50.10±8.49岁. 所有病例临床资料完整, 血液标本采集前均未有输全血或其他成分输血记录, 每例患者均经术后组织病理学确诊, 其中胃窦部105例、贲门部31例、胃体部9例, 所有患者均无其他自身免疫系统疾病或者恶性肿瘤合并存在. 对照组为北京大学血液标本库健康人血液样本152例, 其中性别、年龄等均与病例组相匹配, 其中男84例, 女68例, 平均年龄49.53±6.52岁. 两组人群均系中国汉族人群, 个体之间均无血缘关系.
1.2.1 基因组DNA抽提: 采集静脉全血2 mL, 乙二胺四乙酸(EDTA)抗凝, 常规蛋白酶K消化后苯酚/氯仿提取基因组DNA[9], 总共提取胃癌患者、健康正常人外周血DNA 297例, 提取的DNA溶解于TE中, -20 ℃保存.
1.2.2 多态性位点的选择及引物设计: LMP2/LMP7基因位于染色体6p21.3区域, 我们根据文献选择上述两基因各自位于第二外显子的两个SNP位点[10]: LMP2-codon60处多态位点的存在使得所编码的精氨酸(Arg)置换为半胱氨酸(Cys); LMP7-codon145处位点则为谷氨酰胺(Gln)置换为赖氨酸(Lys), 每个多态性位点的详细资料见表1. 在GenBank(GenBank accession number: AF027865)中检索到LMP2/LMP7基因的全长序列, 利用Generunner3.05(http://www.generunner.com)软件, 设计针对LMP2/LMP7中每个位点的PCR扩增引物, 引物由上海申友生物技术有限公司合成, 引物序列见表1.
| 多态位点 | 氨基酸置换 | PCR引物 | 扩增长度 | 复性温度 | 酶切片段(bp) |
| LMP2-codon60 | Arg→Cys | FW: 5'-CTCCACTTTACAGATGCAGA-3' | 330 bp | 56.5 ℃ | HhaⅠ(79+251) |
| R: 5'-ACTTGGTGACTGTTGACTCC-3' | |||||
| LMP7-codon145 | Gln→Lys | FW: 5'-TCATGGCGCTACTAGATGTATG-3' | 351 bp | 57.5 ℃ | BsmⅠ(146+205) |
| R: 5'-AACTCTTTGTCCTAACTTGCAC-3' |
1.2.3 PCR反应体系及反应条件程序: PCR扩增体系及程序如下: ddH2O 40.5 μL, 10×PCR缓冲液(含1.5 mmol/L Mg2+) 5 μL, 10 mmol/L dNTP 1 μL, Taq酶5 U(Promega Company, USA), 上、下游引物各1 μL (20 μmol/L), DNA模板1 μL (500 ng). PCR扩增采用程序: 94 ℃预变性2 min, 94 ℃变性40 s→退火40 s, 温度依次为56.5 ℃(LMP2-codon60)、57.5 ℃(LMP7-codon145)→72 ℃延伸40 s, 共30个循环, 最后在72 ℃条件下延伸10 min(PE Biosystems, USA), PCR产物采用Multiscreen-PCR纯化板(Millipore Company, USA)进行纯化.
1.2.4 RFLP判断病例组LMP2/LMP7基因型: PCR产物经纯化后, 分别采用限制性内切酶HhaⅠ和BsmⅠ(New England Biolabs Beverly, Ma, USA)对LMP2-codon60, LMP7-codon145这两个位点进行酶切基因分型. 每个反应体系为20 μL, 其中DNA约1 μg、内切酶5 U、加各自内切酶相应的10×Buffer 2 μL、最后加ddH2O至20 μL, 分别在37 ℃和65 ℃下水浴4 h. 酶切完成后进行琼脂糖(20 g/L)电泳(Agarose-1000; Gibco BRL, Rockville, MD, USA), 根据电泳条带数判断每一个体LMP2/LMP7的基因型, 并选取3组不同基因型的个体进行结果测序验证.
1.2.5 LMP2/LMP7多态性位点的单倍型构建: 基于LMP2与LMP7两个基因位于同一条染色体, 之间相距约10 kb, 并且相互毗邻, 故而对其两个多态性位点在297个体中的基因型, 利用以贝叶斯算法(Bayesian algorithm)为基础的PHASE软件(Phase1.0)软件进行逐个个体单倍体的构建[11].
统计学处理 以Mann-Whitney检验比较病例组与对照组之间年龄分布的差异; 以Chi-square(χ2-Test)检验两组之间性别、吸烟、饮酒、等位基因位点等的分布差异, 以及验证基因型频率是否符合Hardy-Weinberg平衡定律; 多态性、单倍型与胃癌风险之间的相关性以OR值(odds ratio)及其95%CI(confidence intervals)表示; OR值及其95%CI以非条件Logistic回归模型分析, 均经年龄、性别、吸烟、饮酒等混杂因素校正后取得. 所有的统计检验均为双侧概率检验, P<0.05表示具有统计学意义, 统计学分析使用SPSS11.5软件(SPSS11.5 for Windows, Chicago, IL).
Mann-Whitney检验比较病例组与对照组之间年龄分布的差异P = 0.95, 差异没有显著性; 在病例与对照组的研究中, 性别、吸烟、饮酒等均无统计学差异(P>0.05)(表2).
| 类别 | 病例组 | 对照组 | P | OR (95%CI) |
| 平均年龄(a)(±SD) | 50.10(±8.49) | 49.53(±6.52) | 0.951 | |
| 性别 n(%) | ||||
| 男 | 82(56.55) | 84(55.26) | 0.822 | |
| 女 | 63(43.45) | 68(44.74) | ||
| 吸烟 n(%) | ||||
| 否 | 97(66.90) | 106(69.74) | 0.602 | |
| 是 | 48(33.10) | 46(30.26) | ||
| 饮酒 n(%) | ||||
| 否 | 108(74.48) | 104(68.42) | 0.252 | |
| 是 | 37(25.52) | 48(31.58) | ||
| LMP2 n(%) | ||||
| Arg | 233(80.35) | 245(80.59) | 0.942 | 0.98(0.66-1.48) |
| Cys | 57(19.65) | 59(19.41) | ||
| LMP7 n(%) | ||||
| Gln | 195(67.24) | 203(66.78) | 0.902 | 1.02(0.73-1.44) |
| Lys | 95(32.76) | 101(33.22) |
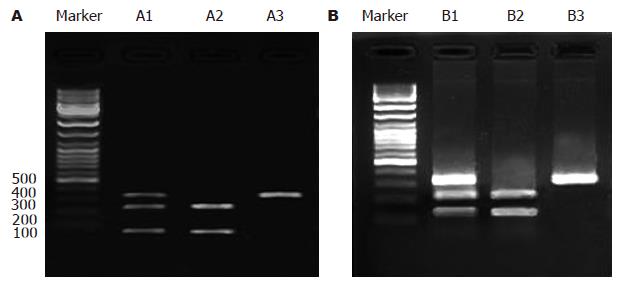
145例胃癌患者与152例健康对照人群中两个多态性位点经PCR-RFLP检测结果显示, LMP2-codon60与LMP7-codon145处的多态性在我们检测的样本中存在(图1); 同时分别进行LMP2/LMP7多态性位点的各自3种基因型数据读取与统计, 经检验P>0.05, 等位基因的频率分布符合Hardy-Weinberg平衡定律. LMP2-codon60位点多态性在正常人群与胃癌患者之间等位基因频率差异没有显著性(P = 0.94), OR = 0.98(95%CI: 0.66-1.48), LMP2-codon145位点多态性在两组人群之间差异亦没有显著性P = 0.90, OR = 1.02(95%CI: 0.73-1.44)(表2). 对于LMP2/LMP7两个点多态性位点, 分别统计其野生型纯合、杂合、多态性纯合基因型频率在正常人群与胃癌患者两组间差异亦没有显著性(P均>0.05)(表3).
针对于病例与对照组LMP/LMP7基因多态性位点的基因型, 用PHASE1.0软件进行每个个体单倍体的构建, 结果共有4种单倍体组合: Arg-Gln, Arg-Lys, Cys-Lys, Cys-Gln(表4). 对每个个体(总297例)逐一进行了--/--, --/Haplotype, Haplotype/Haplotype 3种单倍体型的统计, 鉴于Haplotype/Haplotype纯合单倍体型出现的频率相对较低, 我们的统计分析均将--/Haplotype, Haplotype/Haplotype合并. 结果表明, 4种单倍体型在胃癌与对照组人群之间均无统计学意义(P = 0.87, P = 0.76, P = 0.08, P = 0.79, 表4).
| 单倍体型 | 病例组n(%) | 对照组 n(%) | P1 | OR2(95%CI) |
| A = Arg-Gln | ||||
| 2-/- | 89(61.38) | 92(60.53) | --- | 1.00 |
| -/A+A/A | 56(38.62) | 60(39.47) | 0.87 | 1.04(0.65-1.67) |
| B = Arg-Lys | ||||
| -/- | 114(78.62) | 117(76.97) | --- | 1.00 |
| -/B+B/B | 31(21.38) | 35(23.03) | 0.76 | 1.09(0.63-1.90) |
| C = Cys-Lys | ||||
| -/- | 18(12.41) | 10(6.58) | --- | 1.00 |
| -/C+C/C | 127(87.59) | 142(93.42) | 0.08 | 0.48(0.21-1.09) |
| D = Cys-Gln | ||||
| -/- | 120(82.76) | 128(84.21) | --- | 1.00 |
| -/D+D/D | 25(17.24) | 24(15.79) | 0.79 | 0.92(0.50-1.71) |
LMP2/LMP7基因多态性对于肿瘤发生研究的临床意义在于, LMP基因编码的蛋白酶体水解肿瘤抗原所得到肽段末端氨基酸的性状不同, 其中LMP2能增强酶体对碱性氨基酸残基后肽键的切割, 而不影响中性氨基酸残基后的切割; LMP7则能增强酶体对碱性、中性氨基酸残基后切割, 但不影响酸性残基后切割, 而在基因位点改变后所编码的LMP蛋白酶体水解所得到的肽段羧基末端多为酸性氨基酸残基[12]. 研究发现, 碱性及疏水性氨基酸残基与MHC-Ⅰ类分子结合力强, 而酸性氨基酸残基与MHC-Ⅰ类分子结合力弱, 故LMP2/LMP7基因多态性可决定肽段羧基端氨基酸的性质, 进而对肿瘤抗原处理和递呈过程起到了限制性作用, 使得在后一步由TAP1/TAP2(transporter of antigen presenting, TAP)介导的抗原肽至内质网递呈过程中成为重要的限速步骤, 因而是肿瘤的易感与发生关键因素之一[13-14]. Kang et al[15]曾在其研究中报道LMP2/LMP7在胃癌细胞系的表达是明显减低的, 同时LMP7的低表达与胃癌细胞系中MHC-Ⅰ分子的低表达明显相关. 在以往的一系列的研究中发现, LMP2-codon60或者LMP7-codon145编码位点的多态性与自身免疫性疾病及食管癌的发生有着明显的易感关联[5-8]. 而我们的此次研究发现, LMP2基因codon 60位点(Arg→Cys)在检测的人群中, 无论是等位基因频率(P = 0.94), 还是基因型频率(P = 0.52, P = 0.23)在两组人群中均无显著性差异; 同样, LMP codon145位点(Gln→Lys)多态性在胃癌患者和正常人群中, 等位基因频率(P = 0.90), 基因型频率(P = 0.26, P = 0.38)也没有统计学差异, 因此我们的结果提示, LMP2/LMP7基因的两个多态性位点可能与中国汉族人群中胃癌的发生无遗传易感性关联.
相对于遗传易感因素来说, 每个多态性位点的存在对于疾病的发生与发展并不只是起到一个孤立的作用, 其相互之间亦存在一定的内在关联与作用[16-18], 单倍型的则是这种遗传关联的体现, 所以对于单倍型的研究能够揭示多个SNP与疾病易感的关联[19-21]. 我们进一步的研究进行了LMP2/LMP7基因两个多态性位点的每个个体的单倍型的构建, 并以此进行分析与胃癌的关联. 在我们的研究中, 两个多态性位点共构建的4种单倍体: Arg-Gln, Arg-Lys, Cys-Lys, Cys-Gln, 统计学结果表明, 这4种单倍体对于胃癌的发生均没有易感关联(P>0.05). 因此我们的结果表明, LMP2/LMP7基因的两个SNP不仅单独存在, 而且作为单倍型存在时, 与中国汉族人群中胃癌的发生没有遗传易感性的关联. 虽然我们基于LMP2/LMP7基因多态性对于胃癌遗传易感性的研究没有提供阳性的研究结果, 但本研究的重要意义在于, 在肿瘤抗原的免疫呈递过程中, 作为重要的限速步骤的LMP2/LMP7基因, 其多态性并不与胃癌的发生所相关, 这一点为以后对胃癌发生的其他免疫逃逸机制的遗传学研究提供了一个颇为重要的理论线索, 并为将来进一步寻找胃癌的其他遗传易感性多态位点的研究提供了一个较为明确的参考信息.
肿瘤的免疫逃逸机制是目前肿瘤分子生物学研究的一个热点, LMP2/LMP7基因多态性可改变所编码氨基酸的性质, 对肿瘤抗原处理和递呈过程起到了限制性作用, 从而影响着宿主对肿瘤的易感性. 目前已有研究表明, LMP基因的多态性与肿瘤或者一系列免疫相关性疾病发生有着密切关联
虽然本文研究没有得到阳性的研究结果, 但本研究的重要意义在于, 为以后对胃癌发生的其他免疫逃逸机制的遗传学研究提供了一个颇为重要的理论线索, 为将来进一步寻找胃癌的其他遗传易感性位点的研究提供了一个较为明确的参考信息.
本文结果显示了在肿瘤抗原的免疫呈递过程中, 作为限速步骤的LMP2/LMP7基因的多态性与胃癌的发生不相关, 这为以后对胃癌发生的其他免疫逃逸机制的遗传学研究提供了一个较为重要的实验依据. 实验设计合理, 图表清晰, 表达通顺.
电编: 张敏 编辑: 张焕兰
| 1. | Monaco JJ. A molecular model of MHC class-I-restricted antigen processing. Immunol Today. 1992;13:173-179. [PubMed] |
| 2. | Imanishi T, Kamigaki T, Nakamura T, Hayashi S, Yasuda T, Kawasaki K, Takase S, Ajiki T, Kuroda Y. Correlation between expression of major histocompatibility complex class I and that of antigen presenting machineries in carcinoma cell lines of the pancreas, biliary tract and colon. Kobe J Med Sci. 2006;52:85-95. [PubMed] |
| 3. | Piccinini M, Rinaudo MT, Anselmino A, Ramondetti C, Buccinna B, Fiano V, Ghimenti C, Schiffer D. Characterization of the 20S proteasome in human glioblastomas. Anticancer Res. 2005;25:3203-3210. [PubMed] |
| 4. | Palmowski MJ, Gileadi U, Salio M, Gallimore A, Millrain M, James E, Addey C, Scott D, Dyson J, Simpson E. Role of immunoproteaso-mes in cross-presentation. J Immunol. 2006;177:983-990. [PubMed] |
| 5. | Casp CB, She JX, McCormack WT. Genes of the LMP/TAP cluster are associated with the human autoimmune disease vitiligo. Genes Immun. 2003;4:492-499. [PubMed] |
| 6. | Cao B, Tian X, Li Y, Jiang P, Ning T, Xing H, Zhao Y, Zhang C, Shi X, Chen D. LMP7/TAP2 gene polymorphisms and HPV infection in esophageal carcinoma patients from a high incidence area in China. Carcinogenesis. 2005;26:1280-1284. [PubMed] |
| 7. | Ding HL, Cheng H, Fu ZZ, Deng QL, Yan L, Yan T. The relationship of lmp2 and DR3 genes with susceptibility to type I diabetes mellitus in south China Han population. World J Gastroenterol. 2000;6:111-114. [PubMed] |
| 8. | Dissemond J, Goette P, Moers J, Lindeke A, Goos M, Ferrone S, Wagner SN. Immunoproteasome subunits LMP2 and LMP7 downregulation in primary malignant melanoma lesions: association with lack of spontaneous regression. Melanoma Res. 2003;13:371-377. [PubMed] |
| 9. | Lu XM, Zhang YM, Lin RY, Arzi G, Wang X, Zhang YL, Zhang Y, Wang Y, Wen H. Relationship between genetic polymorphisms of metabolizing enzymes CYP2E1, GSTM1 and Kazakh's esophageal squamous cell cancer in Xinjiang, China. World J Gastroenterol. 2005;11:3651-3654. [PubMed] |
| 10. | Vargas-Alarcon G, Gamboa R, Vergara Y, Rodriguez-Zepeda JM, de la Pena A, Izaguirre R, Zuniga J, Ruiz-Morales JA, Granados J. LMP2 and LMP7 gene polymorphism in Mexican populations: Mestizos and Amerindians. Genes Immun. 2002;3:373-377. [PubMed] |
| 11. | Cox DG, Crusius JB, Peeters PH, Bueno-de-Mesquita HB, Pena AS, Canzian F. Haplotype of prostaglandin synthase 2/cyclooxygenase 2 is involved in the susceptibility to inflammatory bowel disease. World J Gastroenterol. 2005;11:6003-6008. [PubMed] |
| 12. | Lankat-Buttgereit B, Tampe R. The transporter associated with antigen processing: function and implications in human diseases. Physiol Rev. 2002;82:187-204. [PubMed] |
| 13. | Heink S, Fricke B, Ludwig D, Kloetzel PM, Kruger E. Tumor cell lines expressing the proteasome subunit isoform LMP7E1 exhibit immunoproteasome deficiency. Cancer Res. 2006;66:649-652. [PubMed] |
| 14. | Shen YQ, Zhang JQ, Miao FQ, Zhang JM, Jiang Q, Chen H, Shan XN, Xie W. Relationship between the downregulation of HLA class I antigen and clinicopathological significance in gastric cancer. World J Gastroenterol. 2005;11:3628-3631. [PubMed] |
| 15. | Kang JK, Yoon SJ, Kim NK, Heo DS. The expression of MHC class I, TAP1/2, and LMP2/7 gene in human gastric cancer cell lines. Int J Oncol. 2000;16:1159-1163. [PubMed] |
| 16. | Salisbury BA, Pungliya M, Choi JY, Jiang R, Sun XJ, Stephens JC. SNP and haplotype variation in the human genome. Mutat Res. 2003;526:53-61. [PubMed] |
| 17. | Kim KJ, Lee HJ, Park MH, Cha SH, Kim KS, Kim HT, Kimm K, Oh B, Lee JY. SNP identification, linkage disequilibrium, and haplotype analysis for a 200-kb genomic region in a Korean population. Genomics. 2006;88:535-540. [PubMed] |
| 18. | Bahlo M, Stankovich J, Speed TP, Rubio JP, Burfoot RK, Foote SJ. Detecting genome wide haplotype sharing using SNP or microsatellite haplotype data. Hum Genet. 2006;119:38-50. [PubMed] |
| 19. | Li LM, Kim JH, Waterman MS. Haplotype reconstruction from SNP alignment. J Comput Biol. 2004;11:505-516. [PubMed] |
| 20. | Stumpf MP. Haplotype diversity and SNP frequency dependence in the description of genetic variation. Eur J Hum Genet. 2004;12:469-477. [PubMed] |
| 21. | Ke X, Durrant C, Morris AP, Hunt S, Bentley DR, Deloukas P, Cardon LR. Efficiency and consistency of haplotype tagging of dense SNP maps in multiple samples. Hum Mol Genet. 2004;13:2557-2565. [PubMed] |