修回日期: 2006-07-01
接受日期: 2006-07-11
在线出版日期: 2006-08-18
目的: 建立肝癌筛选血清蛋白质指纹图谱模型.
方法: 用表面加强激光解析电离飞行时间质谱技术(SELDI-TOF-MS)及WCX2蛋白芯片获得新发肝癌、肝硬化患者和正常人血清的蛋白质指纹图谱, 用计算机软件进行比较分析, 建立肝癌的筛选模型.
结果: 肝癌患者与健康对照组血清蛋白质指纹图谱之间有5个标志蛋白(4477, 8943, 5181, 8617, 13761 Da)在肝癌患者血清中高表达, 肝癌患者与肝硬化患者血清蛋白质指纹图谱之间2个标志蛋白(4477, 13761 Da)在肝癌患者血清中高表达, 1个标志蛋白(4097 Da)在肝癌患者血清中低表达. SELDI-TOF-MS技术的特异性(60/60, 100%); 敏感度(18/20, 90%). 分析系统筛选出4477, 8943, 13761, 4097 Da标志蛋白建立的肝癌诊断模型.
结论: 建立的血清蛋白质指纹图谱模型能够区分肝癌与非肝癌患者, SELDI-TOF-MS在肝癌的诊断及肿瘤特异性蛋白质生物标志分子的筛选等方面具有一定价值.
引文著录: 刘池波, 潘春琴, 孙灵芬. 应用SELDI-TOF-MS技术建立肝癌筛选血清蛋白质指纹图谱模型. 世界华人消化杂志 2006; 14(23): 2354-2357
Revised: July 1, 2006
Accepted: July 11, 2006
Published online: August 18, 2006
AIM: To establish a serum protein pattern model for screening hepatic cellular carcinoma.
METHODS: The proteomic spectra of patients with hepatic cellular carcinoma, hepatic cirrhosis, and healthy people were obtained by surface-enhanced laser desorption/ionization time-of-flight mass spectrometry (SELDI-TOF-MS) on WCX2 chips. The collected data were compared and analyzed by Biomarker Wizard software.
RESULTS: A group of proteomic peaks were detected. The expression of five protein molecules (4477, 8943, 5181, 8617, 13761 Da) in patients with hepatic cellular carcinoma was significantly higher than those in the controls, and the expression of 4477- and 13761-Da proteins were higher while the 4097-Da one was lower in HCC patients than cirrhosis ones. The specificity and sensitivity of SELDI-TOF-MS were 100% (60/60) and 90% (18/20), respectively. Four protein molecules (4477, 8943, 13761, 4097 Da) were screened as a proteomic model.
CONCLUSION: The discovered serum protein pattern model can efficiently identify patients with or without hepatocellular carcinoma. SELDI-TOF-MS plays a valuable role in the diagnosis of hepatocellular carcinoma and the discovery of new tumor-specific protein biomarkers.
- Citation: Liu CB, Pan CQ, Sun LF. Establishment of serum protein pattern model for screening hepatocellular carcinoma by surface-enhanced laser desorption/ionization time-of-flight mass spectrometry. Shijie Huaren Xiaohua Zazhi 2006; 14(23): 2354-2357
- URL: https://www.wjgnet.com/1009-3079/full/v14/i23/2354.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i23.2354
肝癌为我国常见的恶性肿瘤之一, 肝癌的早期诊断较为困难, 我们利用美国Ciphergen公司生产的表面增强激光解吸离子化时间飞行质谱(surface enhanced laser desorption/ionization)技术对肝癌患者和肝硬化、健康志愿者血清中的蛋白质进行对比分析, 采用生物信息学方法建立可作为肝癌诊断标志的蛋白质组合样式, 寻求可用于临床肝癌早期诊断的特异性生物标记物.
所有肝癌血清样品均为2003-08/2004-06台州市立医院的门诊、住院患者, 40例, 其中男30例, 年龄45-60岁; 女10例, 年龄55-64岁, 经病理证实的肝细胞性肝癌患者. 对照样品来自经健康查体正常的健康志愿者60例, 其中男40例, 年龄50-68岁; 女20例, 年龄50-65岁和经肝穿活检为肝硬化患者20例, 年龄35-45岁. 在10 mL血清分离管中全血采集, 4000 r/min条件下离心10 min. 取30 μL血清分装在0.5 mL离心管中, 于-70 ℃冰箱保存. 美国赛弗吉公司(Ciphergen Biosystems, Inc.)的表面增强激光解吸离子化飞行时间质谱仪(Surface Enhanced Laser Desorption Ionization-time OF Flight-Mass Spectrometry, SELDI-TOF-MS)及该公司配套WCX2(Weak Cationic Exchanger, 弱阳离子交换)芯片. 主要试剂: 尿素、乙腈、三氟乙酸等均购自Sigma公司, SPA(Sinapinic Acid)购自Ciphergen Biosystems公司. U9缓冲液(9 mol/L Urea, 20 g/L CHAPS, 10 g/L DTT, 50 mmol/L Tris-HCl, pH 9.0), U1缓冲液(用50 mmol/L Tris-HCl缓冲液9倍稀释U9缓冲液), WCX2缓冲液(100 mmol/L乙酸钠pH 4.0).
血清经冰浴融解后4 ℃ 20000 r/min离心10 min. 取20 μL血清, 加30 μL U9缓冲液, 4 ℃振摇20 min, 再加100 μL U1缓冲液, 4 ℃震荡30 min. 每个芯池中加150 μL WCX2缓冲液, 振荡孵育5 min 2次. 从上述蛋白变性后的血清样品中取50 μL, 加200 μL WCX2缓冲液稀释, 加至WCX2芯片的Bioprocessor(Ciphergen公司)中, 振荡60 min, 甩去血清标本, 每孔加150 μL WCX2缓冲液, 振荡孵育5 min 2次, 最后芯片用20 mmol/L HEPES(pH 7.4)淋洗, 晾干芯片. 芯片每孔分2次加SPA 0.5 μL, 2次之间自然晾干. 质谱仪参数设定为激光强度220, 灵敏度7, 优化范围800-20000质荷比(m/z). 每条芯片取1点用同一正常人血清作内参照, 芯片间CV≤10%. 检测前用ALL-IN-ONE多肽标准芯片校正, 系统质量偏差≤0.1%. 原始数据先以Proteinchip3.0软件校正.
统计学处理 采用Ciphergen ProteinChip软件和BioMarker Wizard软件对芯片检测得到的蛋白质相对含量及蛋白质质荷比数据按数据挖掘要求进行处理. 用Biomarker Wizard Software3.1软件和Biomarker Patterns Software4.0.1软件对数据进行分组及相关性分析. 比较2组之间蛋白质峰强度, P<0.05具有统计学意义.
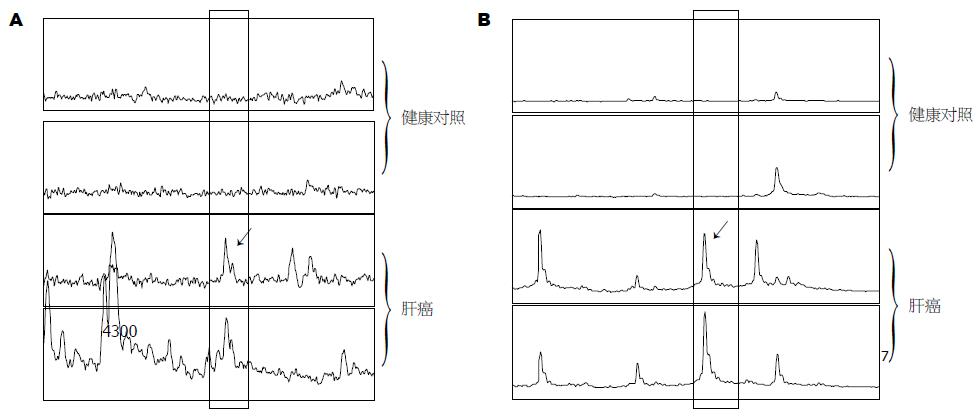
对100例样本检测到的质谱图进行分析比较, 共得到蛋白质峰为68个, 其中发现4477, 8943, 5181, 8617, 13761 Da这5个蛋白质峰具有显著性差异, 且这5个蛋白质均为肝癌患者高于健康志愿者(表1). 通过4477, 8943 Da这2个蛋白质峰建立肝癌蛋白质谱模型可用于区分肝癌与健康志愿者(图1). 此结果表明, 该方法的灵敏度为90%(18/20), 特异性为100%(60/60).
| m/z | 肝癌平均值 | 肝癌标准差 | 正常平均值 | 正常标准差 | P值 |
| 8943 | 9.29 | 7.19 | 1.05 | 0.39 | 3.1×10-7 |
| 8617 | 4.61 | 1.75 | 1.41 | 1.23 | 3.1×10-6 |
| 4477 | 1.20 | 0.65 | 0.30 | 0.10 | 5.4×10-5 |
| 13761 | 0.62 | 0.34 | 0.22 | 0.15 | 4.4×10-5 |
| 8151 | 3.78 | 3.01 | 1.06 | 0.44 | 3.6×10-4 |
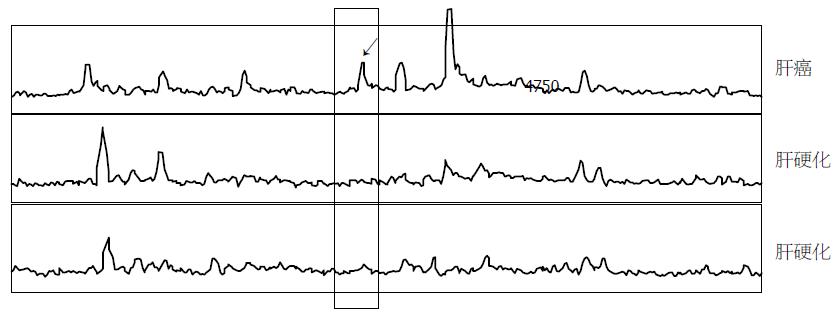
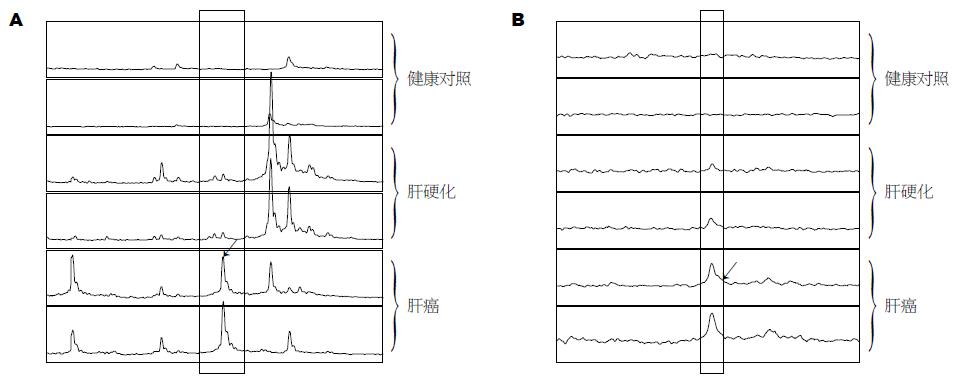
分别对实验中的60例样本检测到的质谱图进行分析比较, 共得到蛋白质峰为46个, 其中4477, 13761, 4097 Da这3个蛋白质峰具有显著性差异. 其中在4477, 13761 Da峰处肝癌的患者均比肝硬化高, 4097 Da峰处肝癌的患者均比肝硬化低, 通过4477 Da这个蛋白质峰即可区分肝癌与肝硬化患者(图2). 同时发现肝癌、肝硬化、健康志愿者3者之间有8943, 13761 Da这2个蛋白质随着病情的严重程度逐渐增加(图3).
目前临床上常用AFP作为肝癌的诊断指标, 但AFP对肝癌诊断阳性率一般为60%-70%, 且在原发性肝癌诊断中也存在假阳性, 因此缺乏可靠的早期诊断指标是其预后较差的主要原因.
蛋白质组学研究的主要技术是双向电泳和质谱等, 虽然双向电泳的技术很成熟, 但他复杂的实验过程和大量的样本需求不适应大规模的标本检测, 限制了双向电泳直接用于临床检测. SELDI蛋白芯片技术根据层析技术和质谱技术发展而来, 其表面可结合特定的蛋白质. 该技术不会破坏所测定的蛋白质, 检测简便, 特异性和敏感性高, 且可将传统方法检测不到的蛋白和多肽检出. 对照分析患者和健康志愿者的质谱图, 可发现和捕获新的特异性疾病相关蛋白. 目前, 用SELDI蛋白芯片技术获得有意义成果的数量已经日益升高[1-4]. 尤其在一些癌症的诊断上, 如前列腺癌[1]、卵巢癌[5]、乳腺癌[6]的诊断上. 从血清中寻求特异性生物标记物用于肝癌等诊断是目前临床急需解决的难题. 本研究采用SELDI-TOF-MS技术通过比较蛋白质组学研究发现一些敏感性和特异性均较高, 且易于临床检测的诊断方法. 我们采用WCX2蛋白质芯片对40例肝癌患者和60例健康志愿者及20例肝硬化患者的血清样本进行了蛋白质谱分析, 通过Biomarker Wizard分析显示, 在肝癌与健康志愿者两组之间以4477, 8943 Da这2个蛋白质建立模型可用于区分肝癌患者与健康志愿者. 所得结果敏感性为90%, 特异性为100%. 在肝癌组与肝硬化组中4477 Da只在肝癌组中高表达, 而在肝硬化组中没有出现. 同时发现肝癌、肝硬化、健康志愿者3者之间有8943, 13761 Da这2个蛋白随着病情的严重程度逐渐增加, 通过这几个峰的鉴别, 其结果准确性高, 有利于患者免于肝穿的痛苦.
本实验将SELDI技术应用于分析肝癌患者血清差异表达蛋白, 发现了几种新的肝癌相关蛋白质, 对肝癌的早期诊断有一定的价值. 对于SELDI技术来说, 每个m/z值对应的可能是很多分子量相近的多肽, 因此不能对体液中的蛋白质进行鉴定[7], 故该蛋白质的结构、功能及是否是已知蛋白等均不清楚, 需要在下一步的实验中予以解决: 通过质谱技术, 分析该蛋白质的多肽片段得到其肽指纹图, 在蛋白质数据库中搜寻与之匹配的蛋白质.
表面增强激光解析电离(SELDI-TOF)蛋白质芯片技术是2002年提出来的一项新兴的蛋白质组学研究技术, 已经引起了学术界的高度重视, 在生物学和临床医学等方面 具有广泛的应用前景. 利用蛋白质芯片具有高度并行性、高通量、微型化和自动化等其他方法无法比拟的优越性, 在病理学、药理学和毒理学等方面 初步取得了卓越的研究成果.
本文应用SELDI-TOF-MS技术分析肝癌、肝硬化、正常人之间所引起的蛋白质组学的变化, 为临床诊断、治疗提供依据, 同时探索SELDI技术在肿瘤诊断方面的应用, 以便深入分析肿瘤的发生与发展.
本文为肝癌临床诊断提供依据, 并可为肝癌预防、术后疗效观察等提供有效措施.
本文采用较先进的检测技术, 病例组、对照组设置合理, 有足够的例数, 结果有一定的可信度, 有新的发现, 能提出自己的观点, 有一定价值.
电编: 张敏 编辑:王晓瑜
| 1. | Adam BL, Qu Y, Davis JW, Ward MD, Clements MA, Cazares LH, Semmes OJ, Schellhammer PF, Yasui Y, Feng Z. Serum protein fingerprinting coupled with a pattern-matching algorithm distinguishes prostate cancer from benign prostate hyperplasia and healthy men. Cancer Res. 2002;62:3609-3614. [PubMed] |
| 2. | Weinberger SR, Boschetti E, Santambien P, Brenac V. Surface-enhanced laser desorption-ionization retentate chromatography mass spectrometry (SELDI-RC-MS): a new method for rapid development of process chromatography conditions. J Chromatogr B Analyt Technol Biomed Life Sci. 2002;782:307-316. [PubMed] |
| 3. | Zhang L, Yu W, He T, Yu J, Caffrey RE, Dalmasso EA, Fu S, Pham T, Mei J, Ho JJ. Contribution of human alpha-defensin 1, 2, and 3 to the anti-HIV-1 activity of CD8 antiviral factor. Science. 2002;298:995-1000. [PubMed] |
| 4. | Bane TK, LeBlanc JF, Lee TD, Riggs AD. DNA affinity capture and protein profiling by SELDI-TOF mass spectrometry: effect of DNA methylation. Nucleic Acids Res. 2002;30:e69. [PubMed] |
| 5. | Bandera CA, Ye B, Mok SC. New technologies for the identification of markers for early detection of ovarian cancer. Curr Opin Obstet Gynecol. 2003;15:51-55. [PubMed] |
| 6. | Paweletz CP, Trock B, Pennanen M, Tsangaris T, Magnant C, Liotta LA, Petricoin EF 3rd. Proteomic patterns of nipple aspirate fluids obtained by SELDI-TOF: potential for new biomarkers to aid in the diagnosis of breast cancer. Dis Markers. 2001;17:301-307. [PubMed] |