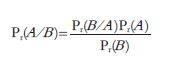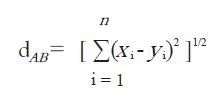修回日期: 2005-10-03
接受日期: 2005-12-02
在线出版日期: 2006-01-08
基因芯片数据分析的目的就是从看似杂乱无序的数据中找出它固有的规律, 本文根据数据分析的目的, 从差异基因表达分析、聚类分析、判别分析以及其它分析等角度对芯片数据分析进行综述, 并对每一种方法的优缺点进行评述, 为正确选用基因芯片数据分析方法提供参考.
引文著录: 吴斌, 沈自尹. 基因表达谱芯片的数据分析. 世界华人消化杂志 2006; 14(1): 68-74
Revised: October 3, 2005
Accepted: December 2, 2005
Published online: January 8, 2006
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2006; 14(1): 68-74
- URL: https://www.wjgnet.com/1009-3079/full/v14/i1/68.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i1.68
基因芯片数据分析就是对从基因芯片高密度杂交点阵图中提取的杂交点荧光强度信号进行的定量分析, 通过有效数据的筛选和相关基因表达谱的聚类, 最终整合杂交点的生物学信息, 发现基因的表达谱与功能可能存在的联系. 然而每次实验都产生海量数据, 如何解读芯片上成千上万个基因点的杂交信息, 将无机的信息数据与有机的生命活动联系起来, 阐释生命特征和规律以及基因的功能, 是生物信息学研究的重要课题[1]. 基因芯片的数据分析方法从机器学习的角度可分为监督分析和非监督分析, 假如分类还没有形成, 非监督分析和聚类方法是恰当的分析方法; 假如分类已经存在, 则监督分析和判别方法就比非监督分析和聚类方法更有效率。根据研究目的的不同[2,3], 我们对基因芯片数据分析方法分类如下: (1)差异基因表达分析: 基因芯片可用于监测基因在不同组织样品中的表达差异, 例如在正常细胞和肿瘤细胞中; (2)聚类分析: 分析基因或样本之间的相互关系, 使用的统计方法主要是聚类分析; (3)判别分析: 以某些在不同样品中表达差异显著的基因作为模版, 通过判别分析就可建立有效的疾病诊断方法.
对于使用参照实验设计进行的重复实验, 可以对2样本的基因表达数据进行差异基因表达分析, 具体方法包括倍数分析、t检验、方差分析等.
倍数分析是最早应用于基因芯片数据分析的方法[4], 该方法是通过对基因芯片的ratio值从大到小排序, ratio是cy3/cy5的比值, 又称R/G值. 一般0.5-2.0范围内的基因不存在显著表达差异, 该范围之外则认为基因的表达出现显著改变. 由于实验条件的不同, 此阈值范围会根据可信区间应有所调整[5,6]. 处理后得到的信息再根据不同要求以各种形式输出, 如柱形图、饼形图、点图等. 该方法的优点是需要的芯片少, 节约研究成本; 缺点是结论过于简单, 很难发现更高层次功能的线索; 除了有非常显著的倍数变化的基因外, 其它变化小的基因的可靠性就值得怀疑了; 这种方法对于预实验或实验初筛是可行的[7]. 此外倍数取值是任意的, 而且可能是不恰当的, 例如, 假如以2倍为标准筛选差异表达基因, 有可能没有1条入选, 结果敏感性为0, 同样也可能出现很多差异表达基因, 结果使人认为倍数筛选法是在盲目的推测[8,9].
差异基因表达分析的另一种方法是t检验[10], 当t超过根据可信度选择的标准时, 比较的两样本被认为存在着差异. 但是t检验常常受到样本量的限制, 由于基因芯片成本昂贵, 重复实验又很费时, 小样本的基因芯片实验是很常见的, 但是小样本导致了不可信的变异估计. 为了克服这种缺点, 研究者提出了调节性t检验(regularized t-test), 它是根据在基因表达水平和变异之间存在着相互关系, 相似的基因表达水平有着相似的变异这个经验, 应用贝叶斯条件概率(贝叶斯定理)统计方法, 通过检测同一张芯片临近的其它基因表达水平, 可以对任何基因的变异程度估计进行弥补. 这种方法对于基因表达的标准差估计优于简单的t-test和固定倍数分析法[11].
Math 1
方差分析(ANOVA)又称变异数分析或F检验, 其目的是推断两组或多组资料的总体均数是否相同, 检验两个或多个样本均数的差异是否有统计学意义, 方差分析可用于差异基因表达研究[12]. 方差分析需要参照实验设计, 参照样本常用多种细胞的mRNA混合而成, 由于所有的细胞同时表达的基因众多, 结果低表达基因在样本混合后就被稀释而减少了参照样本的代表性, 因此, 增加参照样本的细胞不会提高参照样本的代表性[13].
方差分析能计算出哪些基因有统计差异, 但它没有对那些组之间有统计差异进行区分, 比如用单因素方差分析对A、B、C、D 4组进行分析, 对于某一个基因, 方差分析能够分析出A组与B、C、D组之间有差异, 但是B、C、D之间无统计学意义. 这就需要使用均值间的两两比较(post-hoc comparisons)检验, 该检验是对经方差分析后的基因进行下一水平更细节的分析[14]. 即t-检验只能用于检验两样本中均值是否存在显著性差异, 而两两比较技术考虑了多于2样本间均数的比较.
上述所有的参数分析方法必须平衡假阳性、假阴性错误[15,16], 控制假阳性率有4种方法: (1)邦弗朗尼(Bonferroni)方法, 计算公式: Corrected P-value = P-value×n(number of genes in test), 如果纠正P值仍小于错误率(如0.05), 则该基因将属于有表达差异的基因. (2)Bonferroni Step-down(Holm)法, 这种校正方法与邦弗朗尼很相似, 但没有前者严格. 主要思想如下: 每个基因的P值从低到高排序, Corrected P-value=P-value×n(n-1/n-2......), 如果纠正P值仍小于错误率(如0.05), 则该基因将属于有表达差异的基因. (3)Westafall &Young参数法, 前面2种方法都是单独对P值进行纠正, 本方法通过同时对所有基因进行排序, 充分利用基因间的独立性进行P值纠正. 每个基因的P值是按原始资料的排序进行计算; 将资料划分为人工组和对照组而产生新的数据. 采用新数据计算所有基因的P值, 新P值再与以前的P值进行比较, 上述过程重复很多次, 最后计算出纠正P值. 如果纠正P值仍小于错误率(如0.05), 则该基因将属于有表达差异的基因. (4)Benjamini & Hochberg假阳性率法, 该方法是4种方法中最不严谨的方法, 因此可能产生很多的假阳性和假阴性, 其方法如下: 首先对每一个基因的P值由小到大排序, 最大的P值保持不变, 其它基因按下列公式计算P值, Corrected P-value = P value×(n/n-1)以此类推, 若P<0.05则为有差异基因.
上述前3种方法可概括为误差率判断族(family-wise error rate, FWER), 它的特点是允许很少的假阳性基因发生, 而假阳性率(false discovery rate, FDR)是允许一定率的假阳性基因发生. 总之, 假阳性率(FDR)在差异表达与控制假阳性率之间提供了一个好的平衡统计, Bonferroni是最严格的方法, 也是最保守的假阳性估计方法. Westfall & Young参数法是以基因的共同调节进行计算, 因此它的计算是很慢的, 对假阳性率的估计也是很保守的[17,18].
由于微阵列数据存在"噪声"干扰而且不满足正态分布假设, 因此使用t-检验和回归模型进行筛选的方法可能有风险. 非参数检验并不要求数据满足特殊分布的假设, 所以使用非参数方法对变量进行筛选虽然粗放, 但还是可行的[19]. 目前用于基因表达谱数据分析的非参数方法除了传统的非参数t-检验(nonparametric t-test)、Wilcoxon秩和检验(Wilcoxon rank sun test)等外[20], 一些新的非参数方法也应用于基因表达谱数据的分析中, 如经验贝叶斯法(empirical Bayes method)[21]、芯片显著性分析(significance analysis of microarray, SAM)[22]、混合模型法(the mixture model method, MMM)[23]等. 参数法的缺点是分析数据有假设检验, 比如改变样本中的变异可明显影响分析结果, 对同样数据的转换(如对数), 对其分析结果也有明显的影响. 非参数方法对于这种情况的发生更有效, 但是它对表达数据分析的敏感性不如参数方法.
组聚类分析的目的在于辨别在某些特性上相似的事物, 并按这些特性将样本划分成若干类(群), 使同类事物具有高度同质性, 而不同类事物则有高度异质性. 聚类分析是通过建立各种不同的数学模型, 它把基于相似数据特征的变量或样本组合在一起. 归为一个簇的基因在功能上可能相似或关联, 从而找到未知基因的功能信息或已知基因的未知功能. 但是由于使用数据转换、归一化等因素, 导致对聚类分析结果的影响较大; 此外, 聚类只是为了寻求类, 不管所聚的类别是否有意义[28].
分层聚类是将n个样品各作为一类, 计算n个样品两两之间的距离, 构成距离矩阵, 合并距离最近的两类为一新类, 计算新类与当前各类的距离[29]. 再合并、计算, 直至只有一类为止. 分层聚类是第一个被应用于基因表达谱数据分析的聚类方法[30], 由于结果的可视化和基因间关系的明确表现, 广泛地应用于基因表达谱的肿瘤亚型分类和幸存率研究中[31,32].
K-均值聚类是先选择初始凝聚点, 根据欧氏距离系数, 将每个样品归类, 各类的重心代替初始凝聚点, 根据欧氏距离将每个样品不断地归类, 直至分类达到稳定. K-均值算法是采用误差平方和为准则函数的动态聚类方法, 其计算快速, 适合于大规模的数据计算[33]. 如D'ambrosio et al[34]为了理解肥大细胞增生的分子机制和寻找其鉴定的分子标记, 选取肥大细胞增生症患者和正常人的骨髓的单核细胞进行基因芯片实验, 应用K-均值聚类和分层聚类得到同一类的10个基因, 进一步分析鉴定出3个基因属于该疾病的候选标记基因. 但是K-均值聚类也有不足之处, 它对初始凝聚点比较敏感, 如果初始凝聚点没有选择好就可能集合在标准功能值的局域极小值上. 而另一个问题在于它是完全无结构的方法, 聚类的结果是无组织的[35].
Math 2
神经网络中邻近的各个神经元通过侧向交互作用彼此相互竞争, 自适应地发展成检测不同信号的特殊检测器, 这就是自组织特征映射的含义. 其基本原理是将多维数据输入成几何学节点, 相似的数据模式聚成节点, 相隔较近的节点组成相邻的类, 从而使多维的数据模式聚成2维节点的自组织映射图. 自组织映射图允许对类进行调整, 属于监督类聚类[36]. 自组织映射图分类标准明确; 优化的次序好于其它聚类法, 在基因表达谱的数据分析中得到广泛的应用. 如Covell et al[37]认为多种肿瘤可能具有共同的基因表达谱, 他们选取了14种肿瘤和正常对照组织进行基因表达谱研究, 结果自组织图能完全将肿瘤和正常组织区分出来, 自组织图对各种肿瘤的分辨率达到80%的准确性, 其中对白血病、中枢神经系统肿瘤、黑色素瘤、子宫癌、淋巴瘤有很好的判别作用, 对直肠癌、乳腺癌、肺癌的判别差.
基因表达谱常采用单向聚类法(one-way clustering), 即要么以整个样本中特性相似的基因进聚类, 或者以基因表达相似的样本进行聚类. 对样本和基因同时进行聚类就是双向聚类法(two-way clustering)[38], 目前基因表达谱的数据分析常用的双向聚类有基因剃须(gene shaving, GS)和格子模型(plaid models). 基因剃须是通过基因的共同表达值或表达量来鉴定基因的亚类, 基因表达谱分析方法常用监督进行聚类, 没有考虑一个基因可能属于多个类. 基因剃须对基因或样本进行分类既可以是监督的, 也可以是非监督的. 基因剃须近年逐渐被应用于基因表达谱的分析中, 如Hastie et al[39]使用基因剃须方法分析了B细胞淋巴瘤患者的基因表达谱, 鉴定了一小类可用于生存率预判的基因. 作者认为基因剃须方法是一种潜在有用的基因表达谱数据分析方法. Jiang et al[40]使用了2种基因剃须方法筛选肺腺癌的标志基因, 通过和正常组织的基因表达谱比较, 分别筛选到13条和10条, 其中5条是共同的. 格子模型的目的是分析基因芯片数据可解释的生物结构, 即基因或样本的亚类. 各类之间可以进一步聚类, 从而获得稳定的、有意义的分层结构[41,42]. 目前应用格子模型进行基因表达数据分析的实例还不多.
判别分析能够依据样本的某些特性, 以判别样本所属类型. 与聚类分析不同的是, 判别分析是用某种方法将研究对象分成若干类的前提下, 建立判别函数, 用以判定未知对象属于已知分类中的哪一类. 基因判别分析(有监督学习)是在已有数据的基础上建立分类器, 并利用所建立的分类器对未知样品的功能或状态进行预测[45,46]. 目前使用的判别分析方法主要有: 支持向量机、决策树、贝叶斯分类、神经网络法等.
费希尔判别分析是以线形函数为准则进行判别[47], Cho et al[48]应用费希尔判别方法分析肿瘤患者的基因表达谱资料以判别肿瘤的分型. 如Dangond et al[49]将费希尔判别方法应用于计算肌萎缩侧索硬化病的基因表达谱研究中.
也被称为因果网络(causal networks), 是描述数据变量之间依赖关系的一种图形模式, 是一种用来进行推理的模型. 贝叶斯网络为人们提供了一种方便的框架结构来表示因果关系, 这使得不确定性推理在逻辑上更为清晰、更好理解[50]. 如Imoto et al[51]结合贝叶斯网络和生物学知识进行基因表达谱数据的基因网络分析, 并以酿酒酵母的基因表达谱数据为例进行了论证. Kim et al[52]将贝叶斯网络法应用于时间系列的基因表达谱数据的基因网络分析等.
支持向量机是数据挖掘中的一个新方法. 支持向量机能非常成功地处理回归问题(时间序列分析)和模式识别(分类问题、判别分析)等诸多问题, 它通过训练一种"分类器"来辨识与已知的共调控基因表达类型相似的新基因[53-55]. 例如Williams et al[56]为了鉴定出肾母细胞瘤复发的基因表达谱模型, 研究了27例肾母细胞瘤患者的肿瘤组织, 其中13例2 a内复发, 对复发和未复发的肿瘤组织进行基因芯片实验, 并应用支持向量机对基因表达谱数据进行分析, 结果发现了一小类可能用于肿瘤预诊的基因.
决策树是一种常用于预测模型的算法, 它通过将大量数据有目的的分类, 从中找到一些有价值的, 潜在的信息. 它的主要优点是描述简单, 分类速度快, 特别适合大规模的数据处理[57]. Dettling et al[58]比较了不同决策树算法对基因表达谱分析的影响. Middendorf et al[59]应用决策树方法研究了简单生物的基因调节机制.
ANN是一种应用类似于大脑神经突触联接的结构进行信息处理的数学模型. 在这一模型中, 大量的节点(或称"神经元", 或"单元")之间相互联接构成网络, 即"神经网络", 以达到处理信息的目的. 其优势是运行分析时无需在心目中有任何特定模型, 而且, 神经网络可以发现交互作用效果(如年龄和性别的组合效果)[60]. O'Neill et al[61]将神经网络法应用于淋巴瘤基因表达谱数据的分析, 该方法对淋巴瘤预后和诊断都具有较好的判别作用. Sawa et al[62]对酵母属基因表达谱数据进行了欧氏距离、相关系数、相互信息和基于神经网络的聚类分析, 发现基于神经网络的聚类结果较前3种方法更为合理.
在大规模基因表达数据的分析工作中, 由于组织样本例数远远小于所观察基因个数, 如果直接采用前述聚类分析可能产生较大误差, 故需要对聚类算法进行改进. 目前已经提出很多改进的聚类方法, 其中较为流行的方法是应用主成分分析方法对数据进行分析[63,64]. 主成分分析的目的是要对多变量数据矩阵进行最佳综合简化. 使用的方法是寻找这些变量的线性组合-称之为主成分, 使这些主成分间不相关. 为了能用尽量少的主成分个数去反映原始变量间提供的变异信息, 要求各主成分的方差从大到小排列, 第一主成分最能反映数据间的差异. 主成分分析通过合并原来的维数得到更少的维数来表示对象, 同时要求新的维数必须尽可能地反映原有维数所反映的信息, 它有较少的信息丢失. 主成分分析有助于简化分析和多维数据的可视化[65]. 如Crescenzi et al[66]应用主成分分析对60个肿瘤细胞株的基因表达谱数据进行分析, 结果发现肿瘤分型相关的基因有1 375个, 主成分分析得到细胞运动等5个独立的成份. 主成分分析是把原来多个变量化为少数几个综合指标的一种统计分析方法. 从数学角度来看, 这是一种降维处理技术. 而且使这些较少的综合指标既能尽量多地反映原来较多指标所反映的信息, 同时它们之间又是彼此独立的. 但是使用该方法可能导致一部分有用信息的丢失. 为此, Yeung et al[67]采用两个真实数据集和三个模拟数据集作为实验材料, 对采用主成分分析方法所得出的聚类结果作了评估. 他们发现, 进行主成分处理后的聚类质量没有明显提高, 甚至有所降低. 基于以上研究结果, 他们不主张使用PCA方法进行聚类分析.
基因表达分析包括3个层次[68], 首先是单基因水平, 即比较对照组与实验组的每个基因是否存在表达差异, 这主要指差异基因表达分析; 其次是多基因水平, 如按照基因的共同功能、相互作用、共同表达等进行的聚类分析; 最后是系统水平, 即以基因网络形式解释和理解生命现象. 在生物体系中, 基因从来不是单独起作用的, 它们相互作用呈网络状, 因此从网络的观点分析基因表达谱数据必然会导致对生物系统的更高层次的理解, 目前研究者们已经开始了这方面的研究. 正如前述的各种聚类方法, 假如几个基因被聚类在同一组, 它们有可能是共同表达的基因或者是有同样的信号通径, 深入分析这些基因的增强子可能发现它们共同的调节元件, 从而揭示生物系统更高层次的网络[69]. 另外应用目前已知全序列的模式生物(如酵母、结核分枝杆菌), 人们已研制出加载有他们全基因的芯片, 通过比较不同条件下(突变、基因撬出或设计时间系列)表达谱的变化, 再使用贝叶斯网络法等进行系统分析, 可揭示基因功能和调控网络[70]. 此外还可从代谢等角度研究, 比如从新陈代谢分析基因表达的网络关系等.
总之, 基因芯片数据分析的方法众多, 随着研究的进展不断地有新的数学方法应用于芯片的数据分析中步研究
基因芯片技术的出现为生命科学的研究带来了革命性的变化, 然而基因芯片技术产生的海量数据成为制约该技术应用的瓶颈. 了解基因芯片数据分析的研究进展对于正确应用该技术具有重要的意义. 本文对基因芯片数据分析方法综述全面、系统, 最重要的是对每一种方法进行了评述.
1 吴斌, 林乔, 王米渠, 王建. 试论影响基因芯片实验设计的因素. 世界华人消化杂志 2005; 13: 1206-1209
本文对基因表达芯片的数据分析方法进行了综述, 并对差异基因表达分析、聚类分析以及判别分析等分法的优缺点进行了评述, 文章选取的文献较新颖,内容较全面、表达较准确,描述清晰、层次分明,可读性较好, 反映了基因表达芯片数据分析方法的研究进展水平, 对于如何 选用数据分析方法具有较好的参考价值.
电编: 李琪 编辑:菅鑫妍 审读:张海宁
| 1. | Reimers M. Statistical analysis of microarray data. Addict Biol. 2005;10:23-35. [PubMed] |
| 2. | Hackl H, Cabo FS, Sturn A, Wolkenhauer O, Trajanoski Z. Analysis of DNA microarray data. Curr Top Med Chem. 2004;4:1357-1370. [PubMed] |
| 3. | Leung YF, Cavalieri D. Fundamentals of cDNA microarray data analysis. Trends Genet. 2003;19:649-659. [PubMed] |
| 4. | Gerhold D, Lu M, Xu J, Austin C, Caskey CT, Rushmore T. Monitoring expression of genes involved in drug metabolism and toxicology using DNA microarrays. Physiol Genomics. 2001;5:161-170. [PubMed] |
| 5. | Mutch DM, Berger A, Mansourian R, Rytz A, Roberts MA. The limit fold change model: a practical approach for selecting differentially expressed genes from microarray data. BMC Bioinformatics. 2002;3:17. [PubMed] |
| 6. | Yang IV, Chen E, Hasseman JP, Liang W, Frank BC, Wang S, Sharov V, Saeed AI, White J, Li J. Within the fold: assessing differential expression measures and reproducibility in microarray assays. Genome Biol. 2002;3:research0062. [PubMed] |
| 7. | Black MA, Doerge RW. Calculation of the minimum number of replicate spots required for detection of significant gene expression fold change in microarray experiments. Bioinformatics. 2002;18:1609-1616. [PubMed] |
| 8. | Cui X, Churchill GA. Statistical tests for differential expression in cDNA microarray experiments. Genome Biol. 2003;4:210. [PubMed] |
| 9. | Raraty MG, Murphy JA, Mcloughlin E, Smith D, Criddle D, Sutton R. Mechanisms of acinar cell injury in acute pancreatitis. Scand J Surg. 2005;94:89-96. [PubMed] |
| 10. | Baldi P, Long AD. A Bayesian framework for the analysis of microarray expression data: regularized t-test and statistical inferences of gene changes. Bioinformatics. 2001;17:509-519. [PubMed] |
| 11. | Long AD, Mangalam HJ, Chan BY, Tolleri L, Hatfield GW, Baldi P. Improved statistical inference from DNA microarray data using analysis of variance and a Bayesian statistical framework. Analysis of global gene expression in Escherichia coli K12. J Biol Chem. 2001;276:19937-19944. [PubMed] |
| 12. | Pavlidis P. Using ANOVA for gene selection from microarray studies of the nervous system. Methods. 2003;31:282-289. [PubMed] |
| 13. | Hatfield GW, Hung SP, Baldi P. Differential analysis of DNA microarray gene expression data. Mol Microbiol. 2003;47:871-877. [PubMed] |
| 14. | Pan KH, Lih CJ, Cohen SN. Analysis of DNA microarrays using algorithms that employ rule-based expert knowledge. Proc Natl Acad Sci USA. 2002;99:2118-2123. [PubMed] |
| 15. | Aubert J, Bar-Hen A, Daudin J, Robin S. Correction: Determination of the differentially expressed genes in microarray experiments using local FDR. BMC Bioinformatics. 2005;6:42. [PubMed] |
| 16. | Pawitan Y, Murthy KR, Michiels S, Ploner A. Bias in the estimation of false discovery rate in microarray studies. Bioinformatics. 2005;21:3865-3872. [PubMed] |
| 17. | Pawitan Y, Michiels S, Koscielny S, Gusnanto A, Ploner A. False discovery rate, sensitivity and sample size for microarray studies. Bioinformatics. 2005;21:3017-3024. [PubMed] |
| 18. | Grant GR, Liu J, Stoeckert CJ Jr. A practical false discovery rate approach to identifying patterns of differential expression in microarray data. Bioinformatics. 2005;21:2684-2690. [PubMed] |
| 19. | Zhao Y, Pan W. Modified nonparametric approaches to detecting differentially expressed genes in replicated microarray experiments. Bioinformatics. 2003;19:1046-1054. [PubMed] |
| 20. | Troyanskaya OG, Garber ME, Brown PO, Botstein D, Altman RB. Nonparametric methods for identifying differentially expressed genes in microarray data. Bioinformatics. 2002;18:1454-1461. [PubMed] |
| 21. | Efron B, Tibshirani R. Empirical bayes methods and false discovery rates for microarrays. Genet Epidemiol. 2002;23:70-86. [PubMed] |
| 22. | Tusher VG, Tibshirani R, Chu G. Significance analysis of microarrays applied to the ionizing radiation response. Proc Natl Acad Sci USA. 2001;98:5116-5121. [PubMed] |
| 23. | Pan W, Lin J, Le CT. A mixture model approach to detecting differentially expressed genes with microarray data. Funct Integr Genomics. 2003;3:117-124. [PubMed] |
| 24. | Strimmer K. Modeling gene expression measurement error: a quasi-likelihood approach. BMC Bioinformatics. 2003;4:10. [PubMed] |
| 25. | Segal MR, Dahlquist KD, Conklin BR. Regression approaches for microarray data analysis. J Comput Biol. 2003;10:961-980. [PubMed] |
| 26. | Li H, Gui J. Partial Cox regression analysis for high-dimensional microarray gene expression data. Bioinformatics. 2004;20:I208-I215. [PubMed] |
| 27. | Huang X, Pan W. Linear regression and two-class classification with gene expression data. Bioinformatics. 2003;19:2072-2078. [PubMed] |
| 28. | Azuaje F. Clustering-based approaches to discovering and visualising microarray data patterns. Brief Bioinform. 2003;4:31-42. [PubMed] |
| 29. | Guess MJ, Wilson SB. Introduction to hierarchical clustering. J Clin Neurophysiol. 2002;19:144-151. [PubMed] |
| 30. | Levenstien MA, Yang Y, Ott J. Statistical significance for hierarchical clustering in genetic association and microarray expression studies. BMC Bioinformatics. 2003;4:62. [PubMed] |
| 31. | Bertucci F, Salas S, Eysteries S, Nasser V, Finetti P, Ginestier C, Charafe-Jauffret E, Loriod B, Bachelart L, Montfort J. Gene expression profiling of colon cancer by DNA microarrays and correlation with histoclinical parameters. Oncogene. 2004;23:1377-1391. [PubMed] |
| 32. | Sorlie T, Perou CM, Tibshirani R, Aas T, Geisler S, Johnsen H, Hastie T, Eisen MB, van de Rijn M, Jeffrey SS. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications. Proc Natl Acad Sci USA. 2001;98:10869-10874. [PubMed] |
| 33. | Sherlock G. Analysis of large-scale gene expression data. Brief Bioinform. 2001;2:350-362. [PubMed] |
| 34. | D'ambrosio C, Akin C, Wu Y, Magnusson MK, Metcalfe DD. Gene expression analysis in mastocytosis reveals a highly consistent profile with candidate molecular markers. J Allergy Clin Immunol. 2003;112:1162-1170. [PubMed] |
| 35. | Steinley D. Local optima in K-means clustering: what you don't know may hurt you. Psychol Methods. 2003;8:294-304. [PubMed] |
| 36. | Toronen P, Kolehmainen M, Wong G, Castren E. Analysis of gene expression data using self-organizing maps. FEBS Lett. 1999;451:142-146. [PubMed] |
| 37. | Covell DG, Wallqvist A, Rabow AA, Thanki N. Molecular classification of cancer: unsupervised self-organizing map analysis of gene expression microarray data. Mol Cancer Ther. 2003;2:317-332. [PubMed] |
| 38. | Getz G, Levine E, Domany E. Coupled two-way clustering analysis of gene microarray data. Proc Natl Acad Sci USA. 2000;97:12079-12084. [PubMed] |
| 39. | Hastie T, Tibshirani R, Eisen MB, Alizadeh A, Levy R, Staudt L, Chan WC, Botstein D, Brown P. 'Gene shaving' as a method for identifying distinct sets of genes with similar expression patterns. Genome Biol. 2000;1:RESEARCH0003. [PubMed] |
| 40. | Jiang H, Deng Y, Chen HS, Tao L, Sha Q, Chen J, Tsai CJ, Zhang S. Joint analysis of two microarray gene-expression data sets to select lung adenocarcinoma marker genes. BMC Bioinformatics. 2004;5:81 rats. Dig Dis Sci 1995; 40: 2162-2169. [PubMed] |
| 41. | Lazzeroni L, Owen A. Plaid models for gene expression data. Statistica Sinica. 2002;12:61-86. |
| 42. | Available from: http://www-stat. stanford. edu/~owen/plaid. |
| 43. | Wang J, Delabie J, Aasheim H, Smeland E, Myklebost O. Clustering of the SOM easily reveals distinct gene expression patterns: results of a reanalysis of lymphoma study. BMC Bioinformatics. 2002;3:36. [PubMed] |
| 44. | Herrero J, Dopazo J. Combining hierarchical clustering and self-organizing maps for exploratory analysis of gene expression patterns. J Proteome Res. 2002;1:467-470. [PubMed] |
| 45. | Tsai CA, Lee TC, Ho IC, Yang UC, Chen CH, Chen JJ. Multi-class clustering and prediction in the analysis of microarray data. Math Biosci. 2005;193:79-100. [PubMed] |
| 46. | Brock A, Huang S, Ingber DE. Identification of a distinct class of cytoskeleton-associated mRNAs using microarray technology. BMC Cell Biol. 2003;4:6. [PubMed] |
| 47. | Billings SA, Lee KL. Nonlinear fisher discriminant analysis using a minimum squared error cost function and the orthogonal least squares algorithm. Neural Netw. 2002;15:263-270. [PubMed] |
| 48. | Cho JH, Lee D, Park JH, Lee IB. Gene selection and classification from microarray data using kernel machine. FEBS Lett. 2004;571:93-98. [PubMed] |
| 49. | Dangond F, Hwang D, Camelo S, Pasinelli P, Frosch MP, Stephanopoulos G,Stephanopoulos G, Brown RH Jr, Gullans SR. Molecular signature of late-stage human ALS revealed by expression profiling of postmortem spinal cord gray matter. Physiol Genomics. 2004;16:229-239. [PubMed] |
| 50. | Friedman N, Linial M, Nachman I, Pe'er D. Using Bayesian networks to analyze expression data. J Comput Biol. 2000;7:601-620. [PubMed] |
| 51. | Imoto S, Higuchi T, Goto T, Tashiro K, Kuhara S, Miyano S. Combining microarrays and biological knowledge for estimating gene networks via bayesian networks. J Bioinform Comput Biol. 2004;2:77-98. [PubMed] |
| 52. | Kim SY, Imoto S, Miyano S. Inferring gene networks from time series microarray data using dynamic Bayesian networks. Brief Bioinform. 2003;4:228-235. [PubMed] |
| 53. | Furey TS, Cristianini N, Duffy N, Bednarski DW, Schummer M, Haussler D. Support vector machine classification and validation of cancer tissue samples using microarray expression data. Bioinformatics. 2000;16:906-914. [PubMed] |
| 54. | Buerstatte CR, Behar KL, Novotny EJ, Lai JC. Brain regional development of the activity of alpha-ketoglutarate dehydrogenase complex in the rat. Brain Res Dev Brain Res. 2000;125:139-145. [PubMed] |
| 55. | Liu Y. Active learning with support vector machine applied to gene expression data for cancer classification. J Chem Inf Comput Sci. 2004;44:1936-1941. [PubMed] |
| 56. | Williams RD, Hing SN, Greer BT, Whiteford CC, Wei JS, Natrajan R, Kelsey A, Rogers S, Campbell C, Pritchard-Jones K. Prognostic classification of relapsing favorable histology Wilms tumor using cDNA microarray expression profiling and support vector machines. Genes Chromosomes Cancer. 2004;41:65-79. [PubMed] |
| 57. | Podgorelec V, Kokol P, Stiglic B, Rozman I. Decision trees: an overview and their use in medicine. J Med Syst. 2002;26:445-463. [PubMed] |
| 58. | Dettling M, Buhlmann P. Boosting for tumor classification with gene expression data. Bioinformatics. 2003;19:1061-1069. [PubMed] |
| 59. | Middendorf M, Kundaje A, Wiggins C, Freund Y, Leslie C. Predicting genetic regulatory response using classification. Bioinformatics. 2004;20 Suppl 1:I232-I240. [PubMed] |
| 60. | Agatonovic-Kustrin S, Beresford R. Basic concepts of artificial neural network (ANN) modeling and its application in pharmaceutical research. J Pharm Biomed Anal. 2000;22:717-727. [PubMed] |
| 61. | O'Neill MC, Song L. Neural network analysis of lymphoma microarray data: prognosis and diagnosis near-perfect. BMC Bioinformatics. 2003;4:13. [PubMed] |
| 62. | Sawa T, Ohno-Machado L. A neural network-based similarity index for clustering DNA microarray data. Comput Biol Med. 2003;33:1-15. [PubMed] |
| 63. | Wang A, Gehan EA. Gene selection for microarray data analysis using principal component analysis. Stat Med. 2005;24:2069-2087. [PubMed] |
| 64. | Sharov AA, Dudekula DB, Ko MS. A web-based tool for principal component and significance analysis of microarray data. Bioinformatics. 2005;21:2548-2549. [PubMed] |
| 65. | Liu A, Zhang Y, Gehan E, Clarke R. Block principal component analysis with application to gene microarray data classification. Stat Med. 2002;21:3465-3474. [PubMed] |
| 66. | Crescenzi M, Giuliani A. The main biological determinants of tumor line taxonomy elucidated by a principal component analysis of microarray data. FEBS Lett. 2001;507:114-118. [PubMed] |
| 67. | Yeung KY, Ruzzo WL. Principal component analysis for clustering gene expression data. Bioinformatics. 2001;17:763-774. [PubMed] |
| 68. | Slonim DK. From patterns to pathways: gene expression data analysis comes of age. Nat Genet. 2002;32 Suppl:502-508. [PubMed] |
| 69. | Hudson ME, Quail PH. Identification of promoter motifs involved in the network of phytochrome A-regulated gene expression by combined analysis of genomic sequence and microarray data. Plant Physiol. 2003;133:1605-1616. [PubMed] |
| 70. | Gutierrez-Rios RM, Rosenblueth DA, Loza JA, Huerta AM, Glasner JD, Blattner FR, Collado-Vides J. Regulatory network of Escherichia coli: consistency between literature knowledge and microarray profiles. Genome Res. 2003;13:2435-2443. [PubMed] |