修回日期: 2005-10-01
接受日期: 2005-11-04
在线出版日期: 2005-11-28
目的: 运用SELDI蛋白质芯片技术分析体外培养的肝癌细胞株(HepG2)与正常肝细胞株(L02)蛋白质表达差异.
方法: 在体外培养HepG2和L02细胞株, 收获细胞, 将细胞用细胞裂解液裂解后, 采用SELDI蛋白质芯片技术用IMAC3 及WCX2芯片检测HepG2、L02的蛋白质谱.
结果: 体外培养的肝癌细胞株与正常肝细胞株的蛋白质存在差异表达, IMAC3芯片共捕获61个蛋白, 发现差异峰7个, 与L02细胞相比, 其中3个差异蛋白在肝癌细胞中高表达, 4个差异蛋白在肝癌细胞中低表达. WCX2芯片共捕获91个蛋白, 发现差异峰14个, 其中3个差异蛋白在肝癌细胞中高表达, 11个差异蛋白在肝癌细胞中低表达.
结论: SELDI蛋白芯片技术检测肝癌细胞株与正常肝细胞株蛋白质的差异表达方法简便, 敏感性高, 重复性好. .
引文著录: 宋旭霞, 闫志勇, 王斌, 牟文凤, 钱冬萌, 丁守怡, 姚宗良. 体外培养的肝癌细胞株与正常肝细胞株蛋白质的差异表达. 世界华人消化杂志 2005; 13(22): 2689-2692
Revised: October 1, 2005
Accepted: November 4, 2005
Published online: November 28, 2005
AIM: To investigate the differentially expressed proteins between liver cancer cell line HepG2 and normal cell line L02 cultured in vitro using surface enhanced laser desorption ionization (SELDI) protein array technology.
METHODS: The in vitro cultured liver cancer cell line HepG2 and normal cell line L02 were harvested and then split by cell lysate solution. The protein mass spectra of the HepG2 and L02 cells were detected by IMAC3 and WCX2 based on SELDI protein array technology.
RESULTS: A total of 61 proteins were captured by IMAC3 array, and 7 different peaks were found. Comp-ared with those in the L02 cells, 3 differential proteins were highly expressed, while 4 were lowly expressed in the HepG2 cells. A total of 91 proteins were captured by WCX2 array, and 14 differential peaks were found. Among those differentially expressed proteins, 3 were highly expressed, while 11 were lowly expressed in the HepG2 cells.
CONCLUSION: SELDI protein array technology is con-venient, highly sensitive, and repeatable in the detecti-on of the differently expressed proteins between hepat-ocarcinoma cells and normal hepatocytes.
- Citation: Song XX, Yan ZY, Wang B, Mu WF, Qian DM, Ding SY, Yao ZL. Differential protein expression between in vitro cultured liver cancer cell line HepG2 and normal cell line L02. Shijie Huaren Xiaohua Zazhi 2005; 13(22): 2689-2692
- URL: https://www.wjgnet.com/1009-3079/full/v13/i22/2689.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i22.2689
肝细胞癌(hepatocellular carcinoma, HCC)是最常见的恶性肿瘤之一, 严重的威胁着人们的健康. 据国际癌症研究中心(IARC)估计, 2000年全球肝癌发病数为56.4万人[1], 其死亡率居世界第三位. 其中约55%发生在中国, 即中国肝癌发病30.6万人, 肝癌死亡30.0万人[2], 肝癌死亡率居我国肿瘤死因的第二位. 肝癌的早期诊断较为困难, 缺乏可靠的早期诊断指标是其预后较差的主要原因, 大部分肝癌患者发现时已3/4失去手术机会. 甲胎蛋白(AFP)是目前普遍应用的肝癌标志物, 但仍有30%的肝癌患者AFP是阴性的[3], 因此, 探索和建立一种简单、快速、敏感性高和特异性强的早期诊断技术已成为临床医学上的迫切需要. 目前对肝癌发生机制的研究大都是在基因水平, 但是mRNA的水平并不能真正代表所表达的蛋白质水平, 两者之间的相关性通常低于0.5%. 因此, 要求对生物功能的执行者--蛋白质进行研究. SELDI(surface enhanced laser desorption/ionization)蛋白质芯片技术是近年来新兴的一种蛋白质组学技术, 他具有简单, 快捷, 灵敏等特点, 可以检测分子质量在500-500 000 u之间的蛋白或多肽, 而且所需样本体积小(0.5-400 μL)[4-7], 是发现蛋白质标记物的理想方法[8]. 目前SELDI蛋白质芯片技术是蛋白质组学研究中的重要工具, 人们已3/4应用该项技术寻找肿瘤相关性的蛋白质[9-13], 并有报道用蛋白质飞行质谱技术检测体外培养的肝癌细胞株与转染HBV的肝癌细胞株蛋白质的差异表达[14]. 我们采用美国Ciphergen公司研制的SELDI蛋白质芯片技术分析了体外培养的肝癌细胞HepG2和正常肝细胞L02的蛋白质表达谱, 发现了一系列差异表达的蛋白. 为今后从血清中筛选标志蛋白及研究肝癌的发生、发展机制奠定了基础.
正常肝细胞株(L02)购自中科院上海细胞库, 肝母细胞瘤细胞株(HepG2)由第四军医大学吴力克主任医师赠送. HPLC水, 乙晴, 三氟乙酸, SPA(Sinapinic acid), TritonX-100, 尿素, HEPES, CHAPS购自美国Sigma公司, 蛋白质芯片时间质谱分析仪(PBSⅡ-C)及WCX-2(弱阳离子交换芯片)、IMAC3(固定金属亲和芯片)购自美国Ciphergen Biosystems公司. 蛋白质芯片WCX-2(弱阳离子交换芯片)芯片表面结合有弱型阴离子羧基, 可以被分析物表面的正电荷基团相互作用(如赖氨酸、精氨酸和组氨酸)而捕获蛋白, 可用于检测高等电点的蛋白质和生物标记分子. IMAC3(固定金属亲和芯片)其表面结合有亚硝基, 他可以鳌合金属离子(如Cu, Ni, Ga等), 加到芯片表面结合点上的蛋白质通过半胧氨酸和色氮酸位点上的磷酸化氨基酸等与金属离子结合, 主要用于检测磷酸化蛋白及生物标记分子.
1.2.1 细胞总蛋白质的提取: L02、HepG2细胞采用含100 mL/L胎牛血清的RPMI 1640培养基培养, 细胞长成单层后, 用无菌细胞刮刀刮取细胞, PBS洗3次, 细胞计数1010/L, 加入裂解液(8 mol/L Urea, 40 g/L CHAPS, 40 mmol/L Tris-HCl, pH 7.4)200 μL, 4℃剧烈震荡30 min, 14 000 g离心30 min. 采用日立的Gene Spec V蛋白核酸分析系统测定上清的蛋白浓度, 用裂解液调至样品浓度为2 g/L, 其余上清分装-86℃备用. 每种细胞收获3次, 以排除组间差别. 每份样品至少在两个以上的同种芯片上检测, 以排除不同芯片间的差异.
1.2.2 IMAC3蛋白芯片操作步骤: 每孔加入5 μL 50 mmol/L硫酸镍并在湿盒中孵育15 min, 重复操作一次. 用流动的去离子水清洗芯片10 s以除去过多的镍. 每孔加入5 μL结合缓冲液(0.5 mol/L NaCl, pH 7.0), 震荡孵育5 min, 将蛋白芯片放入蛋白工作平台(Bioprocessor), 每孔加入200 μL缓冲液, 震荡孵育5 min. 弃去缓冲液, 每孔立即加入50 μL 1∶2稀释于缓冲液中的样品(蛋白终浓度1 g/L),震荡孵育1 h. 弃去样品, 每孔使用200 μL缓冲液洗涤两次, 每次5 min, 然后用水进行清洗. 从蛋白工作平台中取出蛋白芯片, 空气中干燥, 每孔加入0.5 μL EAM, 重复一次. 干燥后用蛋白质芯片阅读机进行质谱分析.
1.2.3 WCX-2蛋白芯片操作步骤: 将芯片装入蛋白工作平台, 每孔加入200 μL结合缓冲液(50 mmol/L NaAc, pH 4.0)预处理芯片, 置于振荡器5 min, 重复上述操作一次. 在每孔中加入50 μL 1∶2稀释的样品, 剧烈震荡, 孵育1 h. 弃掉样品, 每孔用200 μL结合缓冲液洗涤2次, 每次5 min. 弃去孔中液体, 每孔加入200 μL HPLC水, 立刻甩出. 从蛋白工作平台中取出蛋白芯片, 空气中干燥, 每孔加入0. 5 μL EAM, 重复一次. 干燥后用蛋白质芯片阅读机进行质谱分析.
1.2.4 数据采集和结果分析: 蛋白质芯片采用PBSⅡ-C型读取数据, 用加有All-in-one标准蛋白的NP-20芯片校正质谱仪, 仪器参数设置如下: 激光强度220, 检测敏感度10, 优化Mr范围为2000-10000, 最高为Mr 50 000.
统计学处理 采用Ciphergen proteinchip 3.0版本的分析软件自动采集数据, 然后用Biomarker Wizard软件分析L02、HEPG2细胞的蛋白质谱差异.
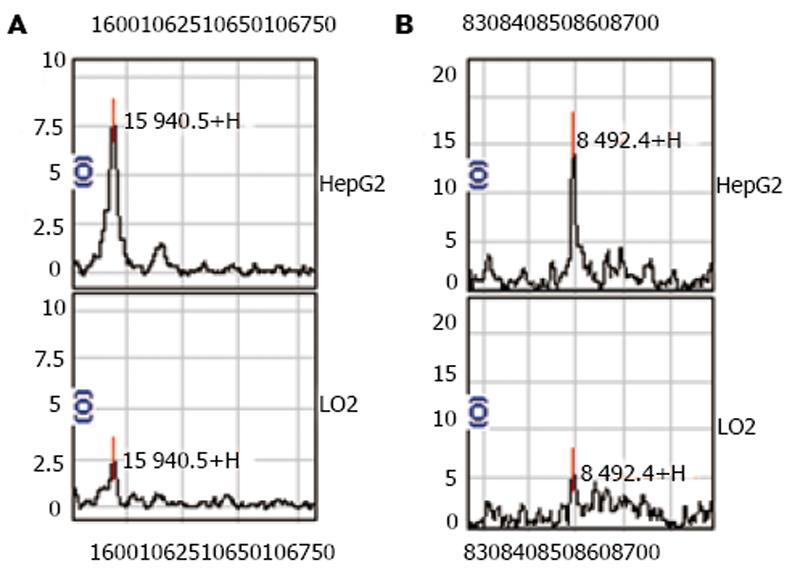
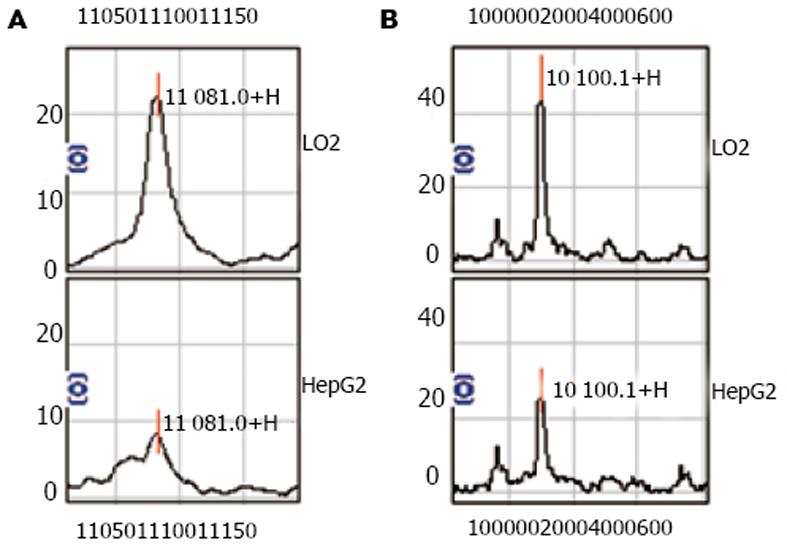
采用PBSⅡ-C型蛋白质芯片阅读机自动收集数据, 设有意义峰的信噪比(signal/noise)为2, 试验结果表明, IMAC3芯片共检测出61个蛋白峰, 发现HepG2、L02细胞的差异峰7个, 其中7 979, 13 428, 15 940 u蛋白分子在肝癌细胞中高表达, 而8 103, 8 492, 10 160, 11 304 u蛋白分子在肝癌细胞中低表达(图1).
采用PBSⅡ-C型蛋白质芯片阅读机自动收集数据, 设有意义峰的信噪比(signal/noise)为2, 试验结果表明, WCX2芯片共检测出91个峰, 发现HepG2、L02细胞差异峰共14个, 其中7 517, 7 945, 7 979 u蛋白分子在肝癌细胞中高表达, 5 061, 5 551, 5 818, 8 428, 10100, 10 312, 11 081, 11 621, 11 662, 11 830, 12 772 u蛋白分子在肝癌细胞中低表达(图2), 在上述差异蛋白中, 7 979 u蛋白峰可同时被IMAC3及WCX2两种芯片捕获.
蛋白质组是指一个基因组、一个细胞或组织或一种生物体所表达的全部蛋白质[15]. 蛋白质组学(proteomics)是蛋白质组概念的延伸, 是在整体水平上研究细胞内蛋白质组成及其活动规律的学科. 与传统的针对单一蛋白质进行的研究相比, 蛋白质组学所采用的高通量和大规模的研究手段, 在回答有关生命活动机制的基本问题方面达到空前的规模和速度. 蛋白质组学研究最终将构建出细胞的"功能图". SELDI-TOF-MS蛋白质芯片技术是近年发展起来的最先进的蛋白质组学技术, 他能对原始样品进行大量检测, 而其他的质谱分析在分析之前都需要有效的样品纯化和制备步骤. SELDI-TOF-MS技术具有高分辨力, 高重复性, 应用简便, 敏感性高等优点[16], 在蛋白质组学中得到越来越广泛的应用.
近来研究表明, 肿瘤性疾病从蛋白质组学的角度又可以被认为是一种蛋白质缺陷病, 其发生过程中有多种蛋白会发生异常变化. 从组织增生产生原位癌到产生癌变的过程中, 有不同的功能性蛋白参与; 转移也是多步骤复杂连续的过程, 与转移有关的特殊基因受到激活并有多种水解酶的参与. 总之, 肿瘤在发生、发展、转移的过程中, 在分子水平有不同的功能性蛋白参与, 并且功能性蛋白很可能在各个环节相互协调共同表达. 蛋白表达异常不仅包括蛋白表达量的增加或减少, 还包括蛋白翻译后加工上的改变, 从而导致肿瘤组织表达的蛋白质谱(protein profile)的改变. 虽然从mRNA水平能够快速研究相应转录子的表达谱, 但在翻译的过程中要进行磷酸化、巯基化、乙酰化、糖基化等修饰, 并不能了解肿瘤进展过程中蛋白表达质与量的变化. 另外有研究表明体内mRNA与相应蛋白的表达水平并没有直接的联系. 因此利用蛋白质组学方法检测蛋白质谱的变化可以更加准确地诊断肿瘤及了解肿瘤的发病机制.
体外培养的细胞株虽然不能完全反映体内细胞的生长状态和生物学活性, 但他具有成分单一, 均质性好, 实验条件容易控制等优点. 尤其在做对比性研究时可避免由组织细胞成分复杂, 细胞异质性高等缺点造成的结果不真实和不可靠[17]. 我们培养了正常肝细胞株L02和肝癌细胞株HepG2, 细胞裂解液裂解细胞, 蛋白定量后SELDI-TOF-MS分析蛋白表达的差异. 在实验中每种细胞收获三次, 以排除组间差别; 每份样品至少做两个点以排除由于芯片不同而引起的误差. 结果表明我们的实验重复性好, 敏感性高. 我们选用了肝癌细胞株HepG2与正常肝细胞株L02蛋白质谱进行比较, 结果发现与正常肝细胞质谱相比, 共有21个差异的蛋白峰, 其中6个标志蛋白在肝癌细胞中高表达, 15个标志蛋白在肝癌细胞中低表达, 在上述差异蛋白中, 7 979 u蛋白峰可同时被IMAC3及WCX2两种芯片捕获. 另外我们在WCX2芯片上发现的7 945 u蛋白峰与Melle et al[18]在肝癌组织中发现的7 935 u的蛋白峰很有可能为一个峰.
任何疾病在出现病理变化之前, 细胞内的蛋白质在成分和数量上都会有相应改变, 有的可能通过血清中的蛋白质反映出来. 我们还研究了HBV感染携带者与HCC患者及正常对照血清蛋白质的差异性表达, 共发现了18个差异的蛋白峰. 我们比较了培养的细胞株与血清的蛋白质谱, 发现二者有一个共同的蛋白峰7 979 u在肝癌中高表达, 这可能是肝癌组织特异性的分泌蛋白, 由肝癌组织产生分泌入血.
我们发现的这些组织特异性蛋白生物标记对肝癌的早期诊断有潜在的应用价值, 更主要的是为研究肝细胞癌变机制提供了一个基础, 而且这些蛋白有可能为肝癌治疗提供新的靶位, 可以进行RNA干扰来阻断其高表达或通过基因导入来促进低表达蛋白的表达. 下一步我们打算应用串联质谱分析鉴定特定的蛋白, 从而进一步研究肝癌的发病机制及肝癌的治疗.
电编: 张敏 编辑: 菅鑫妍 审读: 张海宁
| 1. | Parkin DM. Global cancer statistics in the year 2000. Lancet Oncol. 2001;2:533-543. [PubMed] [DOI] |
| 2. | Wang HT, Wang J, Ou QJ, Liu XP, Chen S. Expression of grow-th hormone receptor mRNA in hepatocellular carcinoma and matched para-cancer cirrhotic liver tissue. Aizheng. 2002;21:146-148. [PubMed] |
| 4. | Robinson JC, Kerjan P, Mirande M. Macromolecular assem-blage of aminoacyl-tRNA synthetases: quantitative analysis of protein-protein interactions and mechanism of complex asse-mbly. J Mol Biol. 2000;304:983-994. [PubMed] [DOI] |
| 5. | Stoop AA, Jespers L, Lasters I, Eldering E, Pannekoek H. High-density mutagenesis by combined DNA shuffling and phage display to assign essential amino acid residues in protein-pro-tein interactions: application to study structure-function of pla-sminogen activation inhibitor 1 (PAI-I). J Mol Biol. 2000;301:1135-1147. [PubMed] [DOI] |
| 6. | Merchant M, Weinberger SR. Recent advancements in surface-enhanced laser desorption/ionization-time of flight-mass spectrometry. Electrophoresis. 2000;21:1164-1177. [PubMed] [DOI] |
| 7. | Srinivas PR, Srivastava S, Hanash S, Wright GL Jr. Proteomics in early detection of cancer. Clin Chem. 2001;47:1901-1911. [PubMed] |
| 8. | Chen YD, Zheng S, Yu JK, Hu X. Application of serum protein pattern model in diagnosis of colorectal cancer. Zhonghua Zhongliu Zazhi. 2004;26:417-420. [PubMed] |
| 9. | Vlahou A, Schellhammer PF, Mendrinos S, Patel K, Kondylis FI, Gong L, Nasim S, Wright Jr GL Jr. Development of a no-vel proteomic approach for the detection of transitional cell carcinoma of the bladder in urine. Am J Pathol. 2001;158:1491-1502. [PubMed] [DOI] |
| 10. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] [DOI] |
| 11. | Petricoin EF 3rd, Ornstein DK, Paweletz CP, Ardekani A, Hackett PS, Hitt BA, Velassco A, Trucco C, Wiegand L, Wood K, Simone CB, Levine PJ, Linehan WM, Emmert-Buck MR, Steinberg SM, Kohn EC, Liotta LA. Serum proteomic patterns for detection of prostate cancer. J Natl Cancer Inst. 2002;94:1576-1578. [PubMed] [DOI] |
| 12. | Li J, Zhang Z, Rosenzweig J, Wang YY, Chan DW. Proteomics and bioinformatics approaches for identification of serum bio-markers to detect breast cancer. Clin Chem. 2002;48:1296-1304. [PubMed] |
| 13. | Poon TC, Yip TT, Chan AT, Yip C, Yip V, Mok TS, Lee CC, Leung TW, Ho SK, Johnson PJ. Comprehensive proteomic pro-filing identifies serum proteomic signatures for detection of hepatocellular carcinoma and its subtypes. Clin Chem. 2003;49:752-760. [PubMed] [DOI] |
| 14. | 丁 守怡, 钱 冬萌, 闫 志勇, 宋 旭霞, 牟 文凤, 王 斌. 蛋白质芯片飞行质谱技术检测体外培养的肝癌细胞株与转染HBV的肝癌细胞株蛋白质的差异表达. 世界华人消化杂志. 2005;13:1684-1687. [DOI] |
| 15. | Peng J, Gygi SP. Proteomics: the move to mixtures. J Mass Spectrom. 2001;36:1083-1091. [PubMed] [DOI] |
| 16. | Persidsky Y, Gendelman HE. Mononuclear phagocyte imm-unity and the neuropathogenesis of HIV-1 infection. J Leukoc Biol. 2003;74:691-701. [PubMed] [DOI] |
| 18. | Melle C, Kaufmann R, Hommann M, Bleul A, Driesch D, Ernst G, von Eggeling F. Proteomic profiling in microdissected hepatocellular carcinoma tissue using ProteinChip technology. Int J Oncol. 2004;24:885-891. [PubMed] [DOI] |