修回日期: 2005-10-01
接受日期: 2005-10-25
在线出版日期: 2005-11-28
目的: 探讨TG-相互作用因子(TGIF)对胃癌SGC-7901细胞株中维甲酸信号通路的影响.
方法: 在TGIF表达质粒稳定转染SGC-7901细胞后, 用Western blot鉴定高表达TGIF的阳性克隆. 在TGIF反义寡核苷酸瞬时转染SGC-7901细胞株后, 用RT-PCR检测转染效率. 再用1 μmol/L ATRA分别处理稳定转染组或瞬时转染组及其对照组细胞, MTT观察细胞增殖速度的变化, 流式细胞术观察细胞凋亡率的变化.
结果: 稳定转染TGIF表达质粒和瞬时转染TGIF反义寡核苷酸对SGC-7901细胞的增殖和凋亡没有明显影响. 但在ATRA作用后, TGIF表达质粒转染组细胞的增殖速度比未转染组和空白质粒转染组细胞快(0.434±0.035 vs 0.386±0.020, 0.360±0.014, P<0.05), 而细胞的凋亡率的变化比未转染组和空白质粒转染组细胞小, 仅由1.14%增加至1.39%. TGIF反义寡核苷酸转染组细胞的增殖速度比未转染组和突变寡核苷酸转染组细胞慢(0.320±0.044 vs 0.388±0.024, 0.409±0.041, P<0.05), 而细胞凋亡率的变化比后两组大, 由3.09%上升至10.2%.
结论: TGIF能拮抗ATRA抑制SGC-7901细胞增殖和诱导其凋亡的作用, TGIF可能参与了对SGC-7901细胞的维甲酸信号通路的抑制.
引文著录: 丁莉利, 胡忠良, 郑长黎. TGIF对胃癌细胞株SGC-7901的维甲酸信号通路的影响. 世界华人消化杂志 2005; 13(22): 2654-2657
Revised: October 1, 2005
Accepted: October 25, 2005
Published online: November 28, 2005
AIM: To investigate the influence of TG-interacting factor (TGIF) on the retinoid signaling pathway in gastr-ic cancer cell line SGC-7901 in vitro.
METHODS: Gastric cell line SGC-7901 were stably tra-nsfected with plasmid PcDNA3.1-TGIF and the highly expressing clone of TGIF was selected by Western blot. Then SGC-7901 cells were transiently transfected with TGIF oliganuleotides and the transfection efficien-cy were analyzed by reverse transcription polymerase chain reaction (PT-PCR). After treatment with 1 ?mol/L all trans-retinoic acid (ATRA), the proliferation of the cells was detected by MTT, and the apoptosis was measured by flow cytometry.
RESULTS: Before treatment with ATRA, the proliferat-ion and apoptosis of SGC-7901 cells were not significa-ntly different between PcDNA3.1-TGIF plasmids and TGIF oliganuleotides group. After treatment, the growth of TGIF transfected cells was more rapid than that of the cells transfected with empty vector and the parental cells (0.434±0.035 vs 0.360±0.014, 0.386±0.020, P <0.05), while the apoptotic rate was just changed from 1.14% to 1.39%, which was less than that of the two latter cells. However, the growth of the cells transfected TGIF oliganuleotides was markedly slower than that of the cells transfected with mutant oliganuleotides and the parental cells (0.320±0.044 vs 0.409±0.041, 0.388±0.024, P <0.05), while the apoptotic rate was increased from 3.09% to 10.2%, which was greater than that of the two latter cells.
CONCLUSION: TGIF can resist the proliferation-inhibition and apoptosis-induction effect of ATRA in SGC-7901 cells, and it may play a role in inhibiting the retinoic signaling pathway in the cells.
- Citation: Ding LL, Hu ZL, Zheng CL. Effects of TG-interacting factor on retinoic signaling pathway in gastric cancer cell line SGC-7901. Shijie Huaren Xiaohua Zazhi 2005; 13(22): 2654-2657
- URL: https://www.wjgnet.com/1009-3079/full/v13/i22/2654.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i22.2654
胃癌是一种严重危害人类生命健康的恶性肿瘤, 目前对胃癌的发病机制尚不十分清楚. TG-相互作用因子(TGIF)是一种广泛表达的核内转录抑制因子, TGIF除参与多种生物的正常发育外, 还可能与多种肿瘤的发生、发展有关[1-3]. TGIF参与了对多条细胞信号传导通路的调节, 尤其是TGF-b信号通路和维甲酸信号通路[4,5]. 研究发现维甲酸信号通路的异常也与多种肿瘤的发生、发展有关[6-10]. 但TGIF能否通过维甲酸信号通路来影响胃癌的发生、发展尚未见文献报道. 我们选用对全反式维甲酸(ATRA)敏感的胃癌细胞株SGC-7901[11], 在用TGIF表达质粒稳定转染或TGIF反义寡核苷酸瞬时转染SGC-7901细胞后, 用ATRA处理细胞, 观察转染前后和ATRA处理前后细胞增殖速度和凋亡的变化, 以探讨TGIF对胃癌SGC-7901细胞株中维甲酸信号通路的影响.
胃癌SGC-7901细胞株购自中科院上海细胞库. 空白质粒PcDNA3.1和TGIF表达质粒PcDNA3.1-TGIF由美国国立卫生研究院遗传药理所Mouradian教授惠赠. 羊源性多克隆抗TGIF抗体购自Santa Cruz公司. TGIF反义寡核苷酸(TGIF ASDON)和突变寡核苷酸参照文献[12], 由上海生物工程技术服务有限公司合成, 均用硫代磷酸化法修饰. TGIF和b-actin扩增引物(表1)由软件Primer Premier 5.0设计, 由上海生物工程技术服务有限公司合成. 阳离子脂质体Lipofectin购自Invitrogen公司, RT-PCR试剂盒购自Promega公司, RPMI 1640培养基和超级小牛血清分别购自Invitrogen公司和杭州四季青公司, ATRA购自Sigma公司.
| 引物 | 序列 | 长度 (bp) |
| TGIF | 上游 5'-GAGAAGGAGAAGGGGCAACCTA-3' | 420 |
| 下游 5' -TGGCAGATCACTGATGGACG-3' | ||
| β-actin | 上游 5'-CCTTCCTGGGCATGGAATCCT-3' | 204 |
| 下游 5'-GGAGCAATGATCTTGATCTT-3' |
1.2.1 细胞培养: 细胞接种于含100 mL/L小牛血清的RPMI 1640培养液中, 在50 mL/L CO2、95% 湿度、37℃培养箱连续培养, 细胞长满瓶底70-80%时传代, 实验用细胞均处于对数生长期. ATRA用无水乙醇配成浓度为10 mmol/L的贮存液, -20℃避光保存. 用药组将ATRA贮存液加入到培养液至终浓度分别为1 mmol/L, 无水乙醇终浓度低于1 mL/L时对细胞的生长和分化无影响. ATRA避光操作.
1.2.2 TGIF基因转染和Western blot检测: 参照Life Technologies公司的试剂说明书进行, 用脂质体lipofe-ctin将PcDNA3.1和PcDNA3.1-TGIF质粒分别转染SGC-7901细胞株, 以未转染细胞作对照. 转染48 h后, 800 mg/L G418筛选至未转染组细胞大量死亡后, 逐渐降低G418的含量, 最后以200 mg/L G418维持筛选, 直至克隆形成. 用细胞裂解液裂解细胞后, 提取细胞总蛋白, 根据BCA试剂盒说明书测细胞总蛋白浓度, 各取80 mg总蛋白行SDS-聚丙烯酰胺凝胶电泳, 电转膜及免疫印迹. 50 g/L脱脂牛奶(PBS配制)封闭1 h, 羊抗人TGIF一抗(1∶300)4℃过夜, 辣根过氧化物酶标记的兔抗羊二抗(1∶1 000), 于水平摇床37℃孵育2 h, DAB显色.
1.2.3 TGIF ASDON转染和RT-PCR检测: 转染前一天, 接种SGC-7901细胞至50 mL培养瓶中(5×105个/瓶)贴壁培养18 h, 无血清培养基洗涤细胞两次, 转染按Lipofectin说明书进行. 实验共分三组, 即TGIF ASDON转染组、突变寡核苷酸转染组和未转染组. 转染6 h后, 弃转染液. 无血清培养基洗涤细胞二次, 各组细胞换用正常培养液或含ATRA的培养液继续培养. 按Trizol说明书提取细胞总蛋白, 琼脂糖凝胶电泳鉴定RNA的提取质量, 核酸分析仪检测RNA含量, 2 mg总RNA按逆转录试剂盒说明书合成cDNA第一链. 再取2 mg cDNA建立PCR反应体系, 进行PCR扩增. 紫外灯下观察结果并摄像.
1.2.4 MTT分析: 消化对数生长期SGC-7901细胞、质粒PcDNA3.1和质粒PcDNA3.1-TGIF稳定转染的SGC-7901细胞并计数, 接种于96孔板中(3×103个/孔). 加盖, 50 mL/L CO2、95%湿度、37℃培养箱中培养24 h后, 吸出培养基, 换用正常培养液或含ATRA的培养液200 mL(4孔/组), 继续培养72 h. 分别加5 g/L的MTT 20 mL于各孔中, 继续培养4 h, 倒尽板中的培养液, 各孔加150 mL DMSO, 轻轻振荡后于酶标仪上(波长490 nm)测定各孔的A值. 消化对数生长期SGC-7901细胞并计数、接种于96孔板中(3×103个/孔), 贴壁培养18 h, 无血清培基洗涤细胞两次, 按Lipofectin说明书分别转染TGIF ASDON、TGIF突变寡核苷酸, 并以未转染细胞做对照. 转染6 h后, 弃转染液, 无血清培养基洗涤细胞二次, 换用正常培养液或含ATRA的培养液200 mL(4孔/组), 继续培养72 h, 用同上方法测定各孔A值.
1.2.5 流式细胞术: 消化对数生长期SGC-7901细胞、质粒PcDNA3.1和质粒PcDNA3.1-TGIF稳定转染的SGC-7901细胞分别接种于50 mL培养瓶培养24 h后, 各组细胞换用正常培养液或含ATRA的培养液继续培养48 h(3瓶/组), 0.2 g/L EDTA和2.5 g/L胰酶消化并收集细胞, PBS洗涤2次, 750 mL/L乙醇4℃固定, 流式细胞仪测定各瓶细胞的DNA含量, Cell Quest软件分析细胞群体的凋亡百分率. 消化对数生长期SGC-7901细胞接种于50 mL培养瓶培养18 h后, 按1.2.4.2方法处理细胞(3瓶/组), 并用同上方法分析细胞群体的凋亡百分率.
统计学处理 数值用mean±SD表示, 采用SPSS11.0统计包进行统计分析, 两组间均数比较使用t检验, 多组间均数比较使用方差分析, 组间均数两两比较使用LSD(least significant difference)法, 以P<0.05为差异有统计学意义.
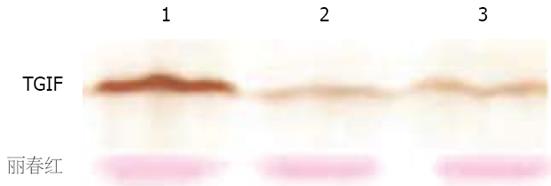
TGIF蛋白的阳性克隆的鉴定Western blot的结果显示: PcDNA3.1-TGIF转染组细胞的TGIF蛋白表达水平明显高于未转染组细胞和PcDNA3.1转染组细胞(图1), 表明我们已建立了稳定表达TGIF的细胞系, 并将用其进行实验.
VPNV能否通过细胞核膜是基因表达的关键环节. 以r = 2.0时制备疫苗, 质粒DNA被阳离子肽充分包裹, 能有效抵抗DNA酶的降解, 这时才具备基因转染的能力, 但电镜下直径较大, 致使其转染效率较低, 因此相应细胞上清中的GM-CSF水平也较低; 当r = 4.0时, 形成的病毒样颗粒呈大小均一的近圆形颗粒, 且直径较小, 约10-40 nm, 绝大多数颗粒直径小于25 nm, 如图5显示. 当r = 8时, 因过多的多肽包裹DNA, 使颗粒的直径更小而致密.
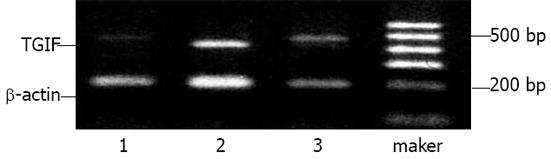
经RT-PCR分析发现SGC-7901细胞转染TGIF ASDON 24 h后, TGIF mRNA表达已明显减少, 低于未转染组和突变寡核苷酸转染组细胞(图2).
在无ATRA作用时, 未转染组、突变寡核苷酸转染组和TGIF ASDON转染组细胞的增殖速度无显著性差异(P>0.05); 经1 mmol/L ATRA作用后, 三组细胞的增殖速度均下降 (P<0.05), 但TGIF ASDON转染组细胞的增殖速度比未转染组和突变寡核苷酸转染组细胞慢(P<0.05). 在无ATRA作用时, 未转染组、突变寡核苷酸转染组和TGIF ASDON转染组细胞的凋亡率分别为2.29%, 3.22%和3.09%; 在ATRA作用后, 三组细胞的凋亡率均上升, 分别为6.35%, 6.63%和10.2%, TGIF ASDON转染组细胞的凋亡率变化比未转染组和突变寡核苷酸转染组细胞大. 瞬时转染TGIF反义寡核苷酸能促进ATRA抑制SGC-7901细胞增殖和诱导其凋亡的作用.
TGIF基因定位于18p11, 由8 162碱基组成, 其cDNA全长1 562碱基, 共有3个外显子, 编码一种由272个氨基酸残基组成的分子质量约为30 ku的核蛋白, 该基因编码的蛋白能识别并结合于细胞核DNA启动子中的一段5'TGTCA3'序列, 故被命名为TG相互作用因子基因. TGIF与DNA结合的核心核苷酸序列5'TGTCA3'也存在于多种维甲酸类受体RAR和RXR识别的启动子序列上, 并与这些RAR和RXR的识别位点比邻或重叠. 研究证实TGIF的同源结构域能与RXR竞争性结合于含有RXRE的启动子上, TGIF和RXRα在CRBPⅡ-RXRE上的结合位点存在部分重叠, 瞬时转染TGIF能抑制RXRα介导的转录反应, 提示TGIF可能通过与RAR和/或RXR竞争性结合于靶基因启动子序列来抑制维甲酸信号通路的作用[5].
但我们的实验结果显示稳定转染TGIF表达质粒的SGC-7901细胞的增殖速度与空白质粒转染组和未转染组细胞比较没有显著性差异, 三组细胞间的凋亡率也相差不大, 这表明高表达TGIF对SGC-7901细胞的增殖和凋亡无明显影响, 不能促进胃癌SGC-7901细胞的恶性生长, 并与文献报道一致[13]. 转染TGIF ASDON的SGC-7901细胞的增殖速度与突变寡核苷酸转染组与未转染组细胞比较没有显著性差异, 三组细胞的凋亡率也相差不大, 说明反义TGIF不能逆转胃癌SGC-7901细胞的恶性生长特点, 这进一步证实TGIF对SGC-7901细胞的增殖和凋亡无明显影响, 因此TGIF可能不影响胃癌细胞的增殖和凋亡. 但这也有可能是由于体外培养细胞在缺乏维甲酸类配体的条件下, 维甲酸信号通路未激活, TGIF无法发挥其抑制维甲酸信号通路的作用.
我们的实验结果还显示ATRA能抑制SGC-7901细胞的增殖和诱导其凋亡. ATRA及其代谢产物9-cRA是维甲酸信号通路中维甲酸类受体RAR和RXR的配体, 因此ATRA能通过维甲酸信号通路抑制SGC-7901细胞的增殖和诱导其凋亡. 稳定转染PcDNA3.1-TGIF质粒能一定程度地拮抗ATRA抑制细胞增殖和诱导细胞凋亡的作用. 我们通过反义寡核苷酸转染实验还发现, TGIF ASDON能下调TGIF的表达, 促进ATRA抑制细胞增殖和诱导细胞凋亡的作用, 提高SGC-7901细胞对ATRA作用的敏感性, 这从相反的角度进一步证实TGIF能拮抗ATRA抑制细胞增殖和诱导细胞凋亡的作用. TGIF可能通过与RAR和/或RXR竞争性结合于启动子上的RARE和/或RXRE位点, 抑制SGC-7901细胞的维甲酸信号通路.
有文献报道在膀胱移行细胞癌中存在TGIF基因所在位点18p11的扩增[1], 在多株食管癌细胞系中有TGIF基因的扩增[2]. 体液中存在生理浓度的维甲酸, 他在生长、分化、发育、代谢、器官形成等生命过程中起重要作用, TGIF能阻断维甲酸信号通路, 因此TGIF有可能通过抑制维甲酸信号通路, 促进肿瘤的发生、发展. TGIF也有可能通过抑制维甲酸信号通路拮抗维甲酸类药物的抑增殖和促凋亡作用, 因此TGIF基因的扩增和高表达可能是肿瘤细胞对维甲酸类药物产生耐药性的原因之一. 而耐药性的产生是维甲酸类药物治疗肿瘤失败的重要原因, 严重影响病人的预后. 因此研究TGIF在维甲酸信号通路中的作用将有助于进一步了解肿瘤细胞对维甲酸类药物产生耐药性的原因
电编: 张敏 编辑: 菅鑫妍 审读: 张海宁
| 1. | Voorter C, Joos S, Bringuier PP, Vallinga M, Poddighe P, Schalken J, du Manoir S, Ramaekers F, Lichter P, Hopman A. Detection of chromosomal imbalances in transitional cell carcinoma of the bladder by comparative genomic hybridization. Am J Pathol. 1995;146:1341-1354. [PubMed] |
| 2. | Nakakuki K, Imoto I, Pimkhaokham A, Fukuda Y, Shimada Y, Imamura M, Amagasa T, Inazawa J. Novel targets for the 18p11.3 amplification frequently observed in esophageal squamous cell carcinomas. Carcinogenesis. 2002;23:19-24. [PubMed] [DOI] |
| 3. | Lou LY, Herrera I, Soosaipillai A, Diamandis EP. Identification of heat shock protein 90 and other proteins as tumour antigens by serological screening of an ovarian carcinoma expression library. Br J Cancer. 2002;87:339-343. [PubMed] [DOI] |
| 4. | Wotton D, Knoepfler PS, Laherty CD, Eisenman RN, Massague J. The Smad transcriptional corepressor TGIF recruits mSin3. Cell Growth Differ. 2001;12:457-463. [PubMed] |
| 5. | Bertolino E, Reimund B, Wildt-Perinic D, Clerc RG. A novel homeobox protein which recognizes a TGT core and func-tionally interferes with a retinoid-responsive motif. J Biol Chem. 1995;270:31178-31188. [PubMed] [DOI] |
| 6. | Melnick A, Licht JD. Deconstructing a disease: RARalpha, its fusion partners, and their roles in the pathogenesis of acute promyelocytic leukemia. Blood. 1999;93:3167-3215. [PubMed] |
| 7. | Xu XC, Ro JY, Lee JS, Shin DM, Hong WK, Lotan R. Differ-ential expression of nuclear retinoid receptors in normal, pre-malignant, and malignant head and neck tissues. Cancer Res. 1994;54:3580-3587. [PubMed] |
| 8. | Xu XC, Sozzi G, Lee JS, Lee JJ, Pastorino U, Pilotti S, Kurie JM, Hong WK, Lotan R. Suppression of retinoic acid receptor beta in non-small-cell lung cancer in vivo: implications for lung cancer development. J Natl Cancer Inst. 1997;89:624-629. [PubMed] [DOI] |
| 9. | Widschwendter M, Berger J, Daxenbichler G, Muller-Holzner E, Widschwendter A, Mayr A, Marth C, Zeimet AG. Loss of retinoic acid receptor beta expression in breast cancer and mo-rphologically normal adjacent tissue but not in the normal breast tissue distant from the cancer. Cancer Res. 1997;57:4158-4161. [PubMed] |
| 10. | Xu XC, Mitchell MF, Silva E, Jetten A, Lotan R. Decreased expression of retinoic acid receptors, transforming growth factor beta, involucrin, and cornifin in cervical intraepithelial neoplasia. Clin Cancer Res. 1999;5:1503-1508. [PubMed] |
| 11. | Wu Q, Chen Z, Su W. Growth inhibition of gastric cancer cells by all-trans retinoic acid through arresting cell cycle progression. Chin Med J. 2001;114:958-961. [PubMed] |
| 12. | Wotton D, Lo RS, Lee S, Massague J. A Smad transcriptional corepressor. Cell. 1999;97:29-39. [PubMed] [DOI] |
| 13. | Hu ZL, Wen JF, Xiao DS, Zhen H, Fu CY. Effects of transformi-ng growth interacting factor on biological behaviors of gastric carcinoma cells. World J Gastroenterol. 2005;11:84-88. [PubMed] [DOI] |