修回日期: 2005-09-01
接受日期: 2005-09-06
在线出版日期: 2005-10-28
目的: 建立RNA多聚酶Ⅱ-染色质免疫沉淀-引物延伸-飞行时间质谱技术(PolⅡ-ChIP-PE-MS), 并利用该方法检测SNP等位特异磷酸化PolⅡ结合量, 为活体状态下研究疾病关联基因SNP的功能研究提供一种高通量的研究手段.
方法: 采用PCR-RFLP方法对HepG2细胞印迹基因SNRPN(仅父源等位基因表达)基因组DNA和cDNA进行基因分型, 利用PolⅡ CTD末端Ser5特异性抗体进行染色质免疫沉淀, 针对SNP位点两侧序列设计PCR引物和延伸引物, 以外侧引物进行PCR扩增, 采用延伸引物进行VLET引物延伸, 产物纯化后经MALDI-TOF质谱鉴定.
结果: 对SNRPN杂合子细胞的SNRPN C1654312T SNP进行PolⅡ-ChIP-PE-MS分析发现, 基因组DNA样本和染色质免疫沉淀前样本在C、T等位点都包含相同PolⅡ结合量, 但免疫沉淀后标本(被磷酸化RNA多聚酶Ⅱ结合的染色质)就仅在T等位点有结合, 与产生mRNA转录的等位点相对应.
结论: 成功建立起PolⅡ-ChIP-PE-MS方法, 利用该方法检测SNP等位特异磷酸化PolⅡ结合量是可靠的, 可以利用PolⅡ-ChIP-PE-MS策略鉴定疾病关联基因SNP的功能.
引文著录: 晏泽辉, 邓国宏, 王宇明. PolⅡ-ChIP-PE-MS方法的建立及其在SNP功能研究中的应用. 世界华人消化杂志 2005; 13(20): 2431-2436
Revised: September 1, 2005
Accepted: September 6, 2005
Published online: October 28, 2005
AIM: To construct a new method named PolII-ChIP-PE-MS (RNA polymerase II-Chromatin immunoprecipitation-primer extension-mass spectrum), and to validate the reliability of this method in the study of single nucleotide polymorphism (SNP) allele-specific quantification of RNA polymerase loading.
METHODS: The C/T SNP of SNRPN at nt 1654312 (numbered according to NT_026446) was genotyped in a genomic DNA sample and a cDNA sample of HepG2 cell line by polymerase chain reaction restriction fragment length polymorphism (PCR-RFLP). After the ChIP assay was performed for phosphorylated Pol II using antibodies highly specific for certain phosphorylated serine residues of the CTD, the extension of short primer was carried out and then the samples were analyzed using matrix-assisted laser desorption ionization time-of-flight (MALDI-TOF).
RESULTS: Comparison of genomic DNA and cDNA of HepG2 cells conformed that, in cell lines heterozygous with respect to this SNP, only one of the two alleles present in the genomic DNA produced an mRNA transcript. When chromatin from heterozygous cell lines was analyzed for the SNP at nt1654312 in SNRPN, the pro-immunoprecipitated starting material and the genomic DNA contained similar amounts of each allele, whereas the material (chromatin to which phosphorylat-ed Pol II was bound) contained predominantly one alle-le, corresponding to the single allele that produced an mRNA transcript.
CONCLUSION: Pol II-ChIP-PE-MS is successfully co-nstructed and it is an applicable and reliable method to detect the SNP allele-specific quantification of RNA polymerase loading. It can be used in detecting the function of disease-related gene SNP.
- Citation: Yan ZH, Deng GH, Wang YM. Construction of RNA polymerase II-Chromatin immunoprecipitation-primer extension-mass spectrum and its application in function research of single nucleotide polymorphism. Shijie Huaren Xiaohua Zazhi 2005; 13(20): 2431-2436
- URL: https://www.wjgnet.com/1009-3079/full/v13/i20/2431.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i20.2431
目前对于常见疾病的遗传易感基因单核苷酸多态性(SNP)的功能研究特别是非编码区(启动子或内含子区域)SNP功能研究相对薄弱[1]. 传统的SNP功能研究手段通常是通过报告基因试验研究不同等位对该序列启动子活性的影响, 或通过电泳移动漂移实验(EMSA)分析不同等位的寡核苷酸探针结合转录因子能力的差异[2]. 但这两类方法均为体外功能研究方法, 缺乏染色质的天然结构及转录的三维调控过程, 且只能对单个SNP位点逐个进行分离的研究, 存在着明显的的局限性, 说服力有限. 我们前期研究所确定的肝病关联SNP位点位于基因转录区[3], 迫切需要发展一种快速、高通量、多位点协同的SNP功能研究方法. 我们利用染色质免疫沉淀(chromatin immunoprecipitation, ChIP)和飞行时间质谱测定等策略建立RNA多聚酶Ⅱ-ChIP-引物延伸-飞行时间质谱技术(RNA polymeraseⅡ-chromatin immunoprecipitation-primer extension-mass spectrum, PolⅡ-ChIP-PE-MS), 以活体状态下检测影响基因调空的SNP的敏感方法, 并利用该方法验证检测SNP等位特异磷酸化RNA多聚酶Ⅱ结合量的可靠性, 为活体状态下研究疾病关联基因SNP的功能研究提供一种高通量研究手段.
日本TOMY公司UR-200P超声波破碎仪, Dynal MPC®-S磁力架. 羊抗鼠IgG包被的Dynabeads®M-280免疫磁珠购自Dynal公司; RNA多聚酶ⅡH14 mAb购自Covance公司; Anti-T Antigen pAb101购自Santa公司; 甲醛、甘氨酸、LiCl、脱氧胆酸钠(DOC)、NP-40、十二烷基硫酸钠(SDS)、鲑鱼精DNA、焦磷酸钠购自Sigma公司; Tris、HEPES、完全蛋白酶抑制剂、糖原、牛血清白蛋白(BSA)、Triton X-100、蛋白酶K、RNA酶A、TriPure、RT-PCR试剂盒等购自Roche公司; RPMI 1640、优级胎牛血清购自Hycolone公司; Taq聚合酶、Hotstar DNA聚合酶、ddNTP、SAP购自MBI公司; 内切酶BstUⅠ、PstⅠ购自BioLab公司; 其他试剂为国内产品, 购自北京鼎国生物技术公司. 细胞裂解缓冲液: 50 mmol/L HEPES(pH7.5), 140 mmol/L NaCl, 1 mmol/L EDTA, 100 mL/L甘油, 5 g/L NP-40, 2.5 g/L Triton X-100, 1×完全蛋白酶抑制剂, 5 g/L胃酶抑素, 10 mmol/L焦磷酸钠. 核裂解缓冲液: 10 mmol/L Tris(pH8), 200 mmol/L NaCl, 1 mmol/L EDTA, 0.5 mmol/L EGTA, 1×完全蛋白酶抑制剂, 5 g/L胃酶抑素, 10 mmol/L焦磷酸钠. 超声降解缓冲液: 10 mmol/L Tris(pH8), 1 mmol/L EDTA, 0.5 mmol/L EGTA, 100 mL/L甘油, 1×完全蛋白酶抑制剂, 5 g/L胃酶抑素, 10 mmol/L焦磷酸钠. ChIP洗涤液1(1×RIPA缓冲液): 50 mmol/L HEPES(pH7.5), 0.5 mol/L LiCl, 1 mmol/L EDTA, 10 g/L NP-40, 7 g/L DOC, 1×完全蛋白酶抑制剂, 5 g/L胃酶抑素, 10 mmol/L焦磷酸钠. ChIP洗涤液2: 含100 mg/L的鲑鱼精子DNA的1×RIPA缓冲液. ChIP洗涤液3: 含300 mmol/L NaCl和终浓度为100 mg/L鲑鱼精子DNA的1×RIPA缓冲液. ChIP洗涤液4: 含250 mmol/L LiCl的1×RIPA缓冲液. PCR引物由北京奥科生物公司合成, 实验中所用引物见表1.
| 引物目的 | 引物名称 | 引物序列 (5'→3') |
| SNRPN 基因鉴定 | 正向引物 | CTACTCTTTGAAGCTTCTGCC |
| 反向引物 | TGAAGATTCGGCCATCTTGC | |
| 第一轮扩增正向引物 | ACGTTGGATGTGCCCAGCTTGCATTGTTTC | |
| SNRPN PE分析 | 第一轮扩增反向引物 | ACGTTGGATGGATGCTTCTGAAGACCTAGG |
| SNRPN 延伸引物 | TGCATTGTTTCTAGGAGAACC |
在含有100 mL/L的胎牛血清RPMI 1640培养基中37℃, 50 mL/L CO2条件下按常规方法培养HepG2细胞数天, 维持胞生长量为107-108个. 按照说明书的方法利用TriPure试剂提取HepG2细胞DNA和总mRNA. 按照RT-PCR试剂盒操作程序进行逆转录以及PCR扩增. 我们按照Sakatani et al[4]所述的方法利用BstUⅠ和PstⅠ对SNRPN的nt 1654312位点SNP进行PCR-RFLP分型.
1.2.1 染色质免疫沉淀: 对HepG2细胞参照参考文献[5]进行的ChIP测定. 终浓度为10 g/L的甲醛固定处于对数生长期的HepG2细胞, 然后用理想配比过剩的甘氨酸(终浓度为0.125 mol/L)终止固定. 刮脱、收集、洗涤细胞后加入适量体积的细胞裂解缓冲液充分溶解, 冰上孵育10 min后, 5 000 g, 4℃离心10 min; 去上清; 然后用细胞核裂解缓冲液重悬沉淀, 冰上孵育10 min后, 5 000 g, 4℃再次离心10 min后去上清. 用超声降解缓冲液再次重悬沉淀, 用超声破碎仪断裂染色质20次, 把染色质片段化到500-1 000 bp左右(冰上操作, 超声探头在标本中的浸没深度至少1 cm; 功率200 W; 脉冲时间20 s; 每次脉冲后将标本在冰水中放置1 min, 每次超声降解过程中避免气泡出现). 将超声降解后得到的样本4℃高速(16 000 g)离心10 min后转移上清到新的试管中, 然后依照实验要求把标本分装成3份(另取10 µL上清用蛋白酶K消化1 h用电泳检测超声片段化效果). 用100 µL含5 g/L BSA冷的PBS预处理包被有羊抗鼠IgG的免疫磁珠12 h, 以减少非特异性吸附. 然后4℃在一个旋转平台上将2 µg RNA多聚酶ⅡH14 mAb和Anti-T Antigen pAb101对照抗体分别与预处理的免疫磁珠轻微摇动孵育过夜. 用DynaMPC磁架收集磁珠, 用含有5 g/L BSA冷的PBS洗涤磁珠3次, 然后分别加入分装好的两份超声降解的标本, 充分混合, 4℃在一个旋转平台上轻微摇动孵育1 h, 最后用DynaMPC磁架收集磁珠(去上清)即得磁珠-染色质免疫沉淀复合物. 分别用ChIP洗涤液1, 2, 3, 4洗涤磁珠-染色质免疫沉淀复合物各2次, 然后用TE溶液洗涤1次. 每次洗涤分别用不同洗涤液充分混合轻摇5 min. 然后用100 µL TE重悬磁珠, 加入1 µg RNA酶A和1 µg蛋白酶K, 在55℃条件下孵育3 h, 然后在65℃条件下孵育12 h以逆转交联; 最后用酚-氯仿-异戊醇(25∶24∶1)抽提2次, 以糖原为载体, 用冰无水乙醇沉淀DNA, 自然凉干后, 用100 µL的TE溶解管底的DNA沉淀(有可能肉眼看不到), 然后标本编号, -20℃保存备用. 然后进行SNRPN基因的PCR扩增, 并利用BstUⅠ和PstⅠ对染色质免疫沉淀标本的SNRPN nt1654312位点进行PCR-RFLP分型.
1.2.2 引物延伸和质谱测定: 我们采用Sun et al[6]建立的VSET(very short extension)方法来进行引物延伸. 首先利用2 µL染色质免疫沉淀标本为模板进行第一轮PCR扩增, 扩增条件是: 96℃预变性1 min, 然后94℃45 s, 56℃ 45 s, 72℃ 30 s, 循环扩增6次; 94℃ 45 s, 65℃ 45 s, 72℃ 30 s, 循环扩增30次; 最后72℃延伸10 min. 然后向PCR产物中加入2 µL SAP, 37℃孵育20 min以去掉反应体系中未结合的残留dNTP, 随后85℃孵育10 min以灭活SAP. 然后以5 µL该PCR产物为模板, 在含有100 µmol/L延伸引物、1 U Hotstar DNA聚合酶和2.5 mmol/L dTTP、ddATP、ddGTP、ddCTP的25 µL反应体系中进行VSET引物延伸, 延伸反应条件是: 94℃预变性5 min, 然后94℃ 5 s, 52℃ 5 s, 72℃ 5 s, 循环延伸40次. 利用醋酸铵-酒精沉淀法纯化延伸产物后溶解在10 µL ddH2O中送上海基康生物技术有限公司进行MALDI-TOF测定.
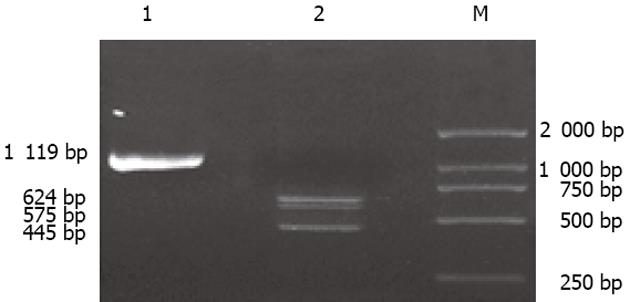
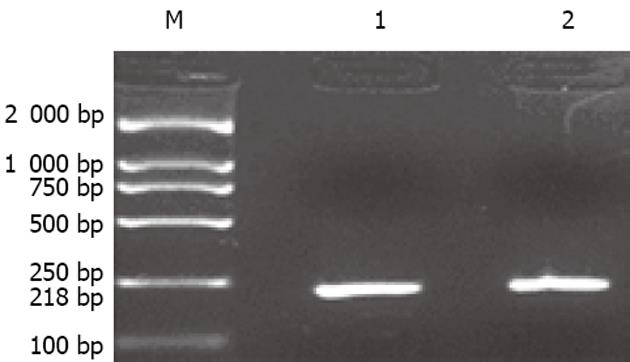
HepG2细胞基因组DNA PCR扩增及BstUⅠ和PstⅠ双酶切后20 g/L琼脂糖电泳分析结果(图1)证明HepG2细胞株SNRPN基因nt1654312位点SNP为C/T杂合子基因型. HepG2细胞cDNA扩增及BstUⅠ酶切后30 g/L琼脂糖电泳分析结果(图2)证明HepG2细胞株SNRPN基因nt1654312保持印迹状态, 仅出现一种等位即T等位表达.
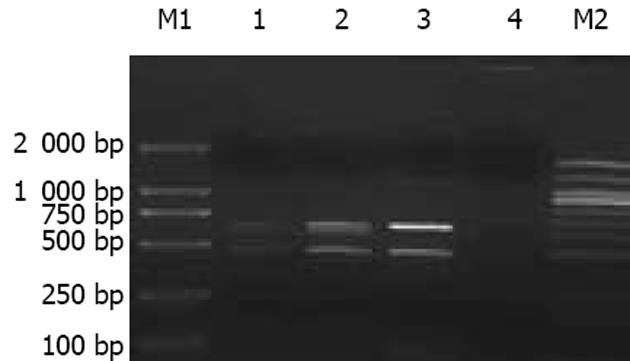
C1654312T杂合子细胞ChIP产物利用BstUⅠ和PstⅠ双酶切后20 g/L琼脂糖电泳分析结果表明免疫沉淀前样本与基因组DNA一样, 依然包含C/T杂合的双等位状态, 但免疫沉淀后标本中就只有T等位的DNA片段被沉淀出来(图3).
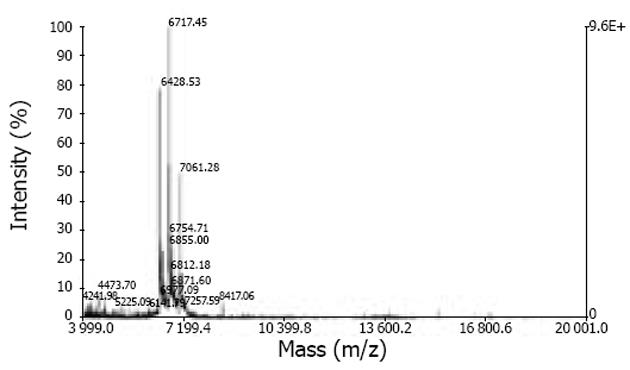
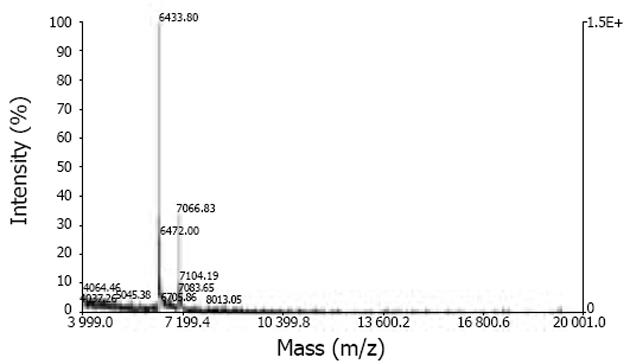
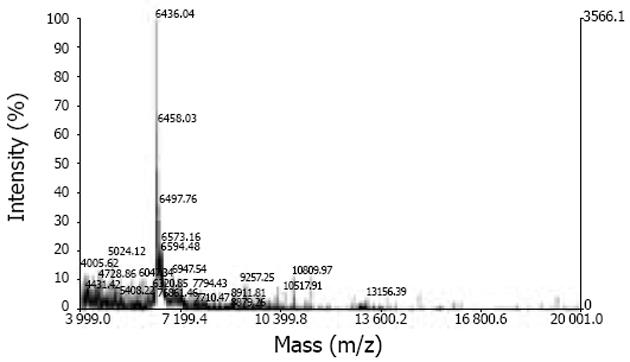
SNRPN C1654312T杂合子HepG2细胞经PolⅡ-ChIP-PE-MS分析结果表明: 染色质免疫沉淀前样本在C、T等位点包含相同PolⅡ结合量(图4); 免疫沉淀后标本(被磷酸化RNA多聚酶Ⅱ结合的染色质)就仅在T等位点有结合(图5), 与产生mRNA转录的等位点相对应; 而对照标本无任何等位表达存在(图6), 说明PolⅡ-ChIP-PE-MS特异性强, 结果可信.
由于SNP为双等位多态, 两种等位多态之间仅有1个碱基的差异, 其功能研究必须在表达水平或蛋白水平揭示单碱基等位特异性的差异. 编码区的SNP通常更容易预测他们的序列和编码蛋白的结构和功能, 但更多的SNP位于非编码区. 对于非编码区(启动子或内含子区域)的关联位点, SNP的等位特异性差异无法在mRNA转录本得到反映, 通常以该SNP位点附近序列为对象, 通过报告基因试验研究不同等位对该序列启动子活性的影响, 或通过电泳移动漂移实验(EMSA)分析不同等位的寡核苷酸探针结合转录因子能力的差异[2]. 这两类方法都存在着明显的局限性: (1)必须逐个位点、逐个等位构建报告基因或合成寡核苷酸探针, 工作量繁重, 费时费力; (2)目标DNA片段很短, 为体外功能研究, 缺乏染色质的天然结构及转录的三维调控过程, 其生物学中肯性受到质疑; (3)只能对SNP位点逐个进行分离的研究, 无法揭示多个位点SNP单倍型的功能; (4)阴性结果只能说明该位点SNP不是致病位点, 不能排除相邻的其他位点. 另外疾病关联rSNP位点通常位于基因转录调控区, 紧密连锁不平衡的区域内可能存在众多的SNP, 逐点进行功能研究存在困难, 要找出其中与疾病直接相关的调节性SNP, 用上述两种方法存在困难, 迫切需要发展一种快速、高通量、多位点协同的SNP功能研究方法, 能够用于评价活体状态下也就是在一个正常功能的细胞核中的自然染色体中SNP对染色质的转录调节作用. 研究已经证实, 与染色质结合的RNA聚合酶Ⅱ是真核细胞mRNA转录所必需的转录因子, RNA聚合酶Ⅱ激活后被磷酸化, 结合到转录起始位点, 启动真核细胞所有mRNA的转录. mRNA转录过程与RNA多聚酶ⅡCTD末端特异性丝氨酸残基磷酸化有关, Ser5的磷酸化与转录的启动、延伸和mRNA的带帽全过程相关, 磷酸化的RNA多聚酶Ⅱ与相关染色质片段的结合量与相对应基因的转录活性是有关系的[7,8]. 我们设想, 位于调控区的SNP要表现功能意义, 应该具备2个条件: (1)位于转录起始点附近; (2)对转录的影响具有等位特异性. 因此我们有理由推测: 活体状态下, 在感兴趣一个或者多个SNP是杂合子的细胞中, 这些SNP也能够影响蛋白质-DNA的相互作用, 表现出等位特异性差异, 因此检测蛋白质-DNA的相互作用的等位特异性差异就可以反映出现在这些SNP表达过程中的不同影响; 杂合子细胞同源染色体上SNP位点两个等位区域转录活性在宏观上可以表现为磷酸化RNA多聚酶Ⅱ结合量, 因此检测磷酸化RNA多聚酶Ⅱ结合量差异即可精巧地反映等位特异性转录活性的差异.
一般调节性多态性的作用是微小的, 这就需要一种高通量、敏感的分析方法来测定同一份免疫沉淀样本中两个不同等位点的相对结合量才能反应出这种微小差异. 而染色质免疫沉淀(ChIP)技术是近几年来发展的一种高通量DNA-蛋白相互作用研究技术, 能实现维持染色质天然结构状态的转录活性观察, 且易于高通量进行[9,10]. 为达到此目的, 我们通过PCR来扩增免疫沉淀样本中特异性DNA序列, 然后利用引物延伸和基于质谱吸光的原理建立的飞行时间-质谱技术(MALDI-TOF)来进行分析. 在本研究中, 我们试图证明: 是否可以用RNA多聚酶Ⅱ-ChIP-引物延伸及飞行时间质谱技术(PolⅡ-ChIP-PE-MS)来检测磷酸化的RNA多聚酶Ⅱ对细胞中一个基因的两种不同等位点的结合量差异; 同时也试图研究这种差异是否与体内基因表达等位差异性有关系. 我们首先通过磷酸化RNA多聚酶Ⅱ-ChIP技术将杂合子细胞目标基因SNP附近的DNA片段沉淀、分离, 然后利用该位点特异的引物扩增, 采用引物延伸-质谱(PE-MS)技术[10,11]鉴定沉淀的DNA片段中目标SNP两种等位的比率, 该比率可以反映两种等位染色体之间该位点RNA多聚酶Ⅱ结合量的差异. 如果有差异, 表明该SNP能影响所在基因的转录活性; 如果没有差异, 则该SNP不能影响基因的转录量. 为此, 我们利用该方法研究了SNRPN(编码小核糖核蛋白多肽N)的表达. SNRPN是一个印迹基因, 它的两个等位基因根据父系来源的不同有显著的不同表达水平(仅父源等位基因表达). 为了区分不同表达量的等位基因, 我们选择SNRPN外显子4上C1654312T(数字依据NT_026446来确定)为杂合子的HepG2细胞作为研究对象, 以研究该C/T SNP(dbSNP rs705)的等位特异性表达情况, 比较染色体DNA并以cDNA验证该结果. 结果表明, C/T杂合子HepG2细胞系中, SNRPN基因依然维持其印迹状态, 逆转录PCR产物酶切实验证实SNRPN等位基因仅仅只有一个等位产生mRNA的转录, 仅仅父源的T等位出现转录. 同时我们利用一种对RNA多聚酶ⅡCTD的磷酸化丝氨酸残基高度特异性的抗体进行ChIP测定两种等位对磷酸化RNA多聚酶Ⅱ的结合量. 当对SNRPN杂合子细胞进行PolⅡ-ChIP-PE-MS分析发现, 基因组DNA以及免疫沉淀前标本在每个等位基因都有相同的磷酸化RNA多聚酶Ⅱ结合量, 但免疫沉淀后的物质(被磷酸化RNA多聚酶Ⅱ结合的染色质)则仅在一个等位基因即T等位有磷酸化RNA多聚酶Ⅱ结合, 这表明在RNA多聚酶Ⅱ被TFⅡH磷酸化之前(也就是mRNA合成开始之前)SNRPN的C等位基因是被转录抑制的, 这一结果与实验中产生mRNA转录的等位点相对应.
本研究说明, PolⅡ-ChIP-PE-MS检测出的磷酸化RNA多聚酶Ⅱ结合量可以反应出等位基因特异性的表达量, 该方法可以精确的反应出双等位的SNP两种等位之间的精确表达量的差异, 可以用于筛选影响基因表达的调节性SNP及其功能研究. 利用PolⅡ-ChIP-VSET-MS策略进行SNP的功能研究, 能够实现ChIP的优点, 在活体状态下用甲醛固定生长期的细胞, 能够自然出现单倍型的活体染色质的环境, 转录调节过程能得到活体状态下一样的控制, 具有在活体状态下进行分析的技术优势; 其次该方法是通过检测RNA多聚酶Ⅱ等位特异性结合量的差异来精确反映体内的调节性多态性的特征, 在RNA转录本中无合适特定遗传标志出现的条件下也能敏感的检测出影响体内基因调节的SNP, 特别适合那些位于非编码区(启动子或内含子区域)调节性SNP的功能研究, 而后者正是当前研究的难点和热点; 再次, 本研究虽然只分析了印迹基因SNRPN的等位表达状态, 但是这种带有一般性的方法能够应用于任何自然状态下发生的DNA-蛋白质相互作用, 这就能够使我们可以利用该方法体内状态下来研究被特殊的候选因子结合的等位特异性调节转录过程状态; 另外, 该方法可以同时免疫沉淀出所有的被RNA多聚酶Ⅱ结合的(有或影响mRNA表达)DNA片段, 因此设计多种位点的延伸引物, 同时利用质谱分析技术的高通量特性, 可以进行多重引物延伸-质谱测定, 从而可以进行快速、高通量、多位点协同的SNP功能研究.
电编: 张敏 编辑: 潘伯荣 审读: 张海宁
| 2. | Pitarque M, von Richter O, Rodriguez-Antona C, Wang J, Oscarson M, Ingelman-Sundberg M. A nicotine C-oxidase gene (CYP2A6) polymorphism important for promoter activity. Hum Mutat. 2004;23:258-266. [PubMed] [DOI] |
| 3. | Deng G, Zhou G, Zhai Y, Li S, Li X, Li Y, Zhang R, Yao Z, Shen Y, Qiang B. Association of estrogen receptor alpha polymorphisms with susceptibility to chronic hepatitis B virus infection. Hepatology. 2004;40:318-326. [PubMed] [DOI] |
| 4. | Sakatani T, Wei M, Katoh M, Okita C, Wada D, Mitsuya K, Meguro M, Ikeguchi M, Ito H, Tycko B. Epigenetic heterogeneity at imprinted loci in normal populations. Biochem Biophys Res Commun. 2001;283:1124-1130. [PubMed] [DOI] |
| 5. | Takahashi Y, Rayman JB, Dynlacht BD. Analysis of promoter binding by the E2F and pRB families in vivo: distinct E2F proteins mediate activation and repression. Genes Dev. 2000;14:804-816. [PubMed] |
| 6. | Sun X, Ding H, Hung K, Guo B. A new MALDI-TOF based mini-sequencing assay for genotyping of SNPS. Nucleic Acids Res. 2000;28:E68. [PubMed] [DOI] |
| 7. | O'Brien T, Hardin S, Greenleaf A, Lis JT. Phosphorylation of RNA polymerase II C-terminal domain and transcriptional elongation. Nature. 1994;370:75-77. [PubMed] [DOI] |
| 8. | Komarnitsky P, Cho EJ, Buratowski S. Different phosphorylated forms of RNA polymerase II and associated mRNA processing factors during transcription. Genes Dev. 2000;14:2452-2460. [PubMed] [DOI] |
| 9. | Odom DT, Zizlsperger N, Gordon DB, Bell GW, Rinaldi NJ, Murray HL, Volkert TL, Schreiber J, Rolfe PA, Gifford DK. Control of pancreas and liver gene expression by HNF transcription factors. Science. 2004;303:1378-1381. [PubMed] [DOI] |
| 10. | Buck MJ, Lieb JD. ChIP-chip: considerations for the design, analysis, and application of genome-wide chromatin immunoprecipitation experiments. Genomics. 2004;83:349-360. [PubMed] [DOI] |
| 11. | Storm N, Darnhofer-Patel B, van den Boom D, Rodi CP. MALDI-TOF mass spectrometry-based SNP genotyping. Methods Mol Biol. 2003;212:241-262. [PubMed] |