修回日期: 2005-08-14
接受日期: 2005-08-26
在线出版日期: 2005-10-15
目的: 建立直肠癌筛选血清蛋白质指纹图谱模型并初步验证.
方法: 用表面加强激光解析电离飞行时间质谱技术(SELDI-TOF-MS)及WCX2蛋白芯片获得新发直肠癌、直肠息肉患者和正常人血清的蛋白质指纹图谱, 用计算机软件进行比较分析, 建立直肠癌的筛选模型, 并对其进行了盲法验证.
结果: 直肠癌组与对照组共有26个蛋白质有显著性差异(P<0.05); 以其中4个蛋白质生物标志物(质/荷比9 295, 3 730, 3 938和4 095)组建的筛选模型检测正确率为96.8%(93/96), 经盲法验证, 其灵敏度为95.0%(38/40), 特异性为93.4%(45/48)
结论: 建立的血清蛋白质指纹图谱模型能够区分直肠癌与非直肠癌患者, SELDI-TOF-MS在直肠癌的诊断及肿瘤特异性蛋白质生物标志分子的筛选等方面具有一定价值.
引文著录: 闫志勇, 钱冬萌, 丁守怡, 宋旭霞, 王斌. 应用 SELDI-TOF-MS 技术建立直肠癌筛选血清蛋白质指纹图谱模型. 世界华人消化杂志 2005; 13(19): 2395-2398
Revised: August 14, 2005
Accepted: August 26, 2005
Published online: October 15, 2005
AIM: To establish a serum protein pattern model for screening rectal carcinoma.
METHODS: The proteomic spectra of patients with rectal carcinoma, rectal polypus, and healthy people were obtained by surface-enhanced laser desorption/ionization time-of-flight mass spectrometry (SELD-TOF-MS) on WCX-2 chips. The collected data were compared and analyzed by Biomaker Wizard software (BPS) to set up the primary serum protein pattern model for screening rectal carcinoma. Then the pattern was evaluated by masked test.
RESULTS: A total of 26 protein was significantly different between the rectal carcinoma and normal controls (P < 0.05), among which 4 (m/z 9 295, 3 730, 3 938, and 4 095) were selected to set up an optimal serum protein biomarker pattern model. And the correct rate of this model was 96.8% (93/96). Its sensitivity and specificity was 95.0% (38/40) and 93.4% (45/48), respectively, when tested by masked samples.
CONCLUSION: The discovered serum protein pattern model can efficiently identify patients with and without rectal carcinoma. SELDI-TOF-MS plays a valuable role in the diagnosis of rectal carcinoma and the discovery of new tumor-specific protein biomarkers.
- Citation: Yan ZY, Qian DM, Ding SY, Song XX, Wang B. Establishment of serum protein pattern model for screening rectal carcinoma by SELDI-TOF-MS. Shijie Huaren Xiaohua Zazhi 2005; 13(19): 2395-2398
- URL: https://www.wjgnet.com/1009-3079/full/v13/i19/2395.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i19.2395
直肠癌是一种常见恶性肿瘤, 在消化道恶性肿瘤中仅次于胃癌居第2位, 治疗后5年存活率仅为50-60%, 居癌症死因第3位[1]. 造成其高死亡率的主要原因是目前尚缺乏特异性高的早期诊断方法[2]. 最近几年出现的表面加强激光解析电离飞行时间质谱技术(surface-enhanced laser desorption/ionization time-of-flight mass spectrometry, SELDI-TOF-MS)是蛋白质组学研究中重 要的工具, 已开始应用于肿瘤的早期诊断生物标志物的筛选[3-8]. 我们利用该技术检测了直肠癌、直肠息肉以及健康人血清的蛋白质表达谱, 分析并建立了用于直肠癌筛选血清蛋白质指纹图谱模型, 现报告如下
直肠癌患者96例, 为2003-02/2005-05期间青岛大学医学院第一、二附属医院、青岛市立医院、青岛市肿瘤医院经临床病理确诊且未经治疗的新发病例, 其中男61例, 女35例, 中位年龄58岁(37-72岁). 另选性别、年龄匹配的直肠息肉患者30例和正常健康者58例作为非直肠癌对照. 上述标本中, 随机筛选56例直肠癌和40例非直肠癌对照用于建立血清蛋白质指纹图谱筛选模型, 其余用于模型的验证分析. NaAc、尿素, 水(HPLC级), 乙氰, 三氟乙酸, SPA(Sinapinic acid), Triton, Tris-Cl均购自美国Sigma公司; 蛋白质芯片时间质谱分析仪(PBSⅡC)、WCX-2弱阳离子交换蛋白质芯片(weak cation exchange)和All-in-one标准蛋白质NP20校正芯片为美国Ciphergen Biosystems公司产品.
1.2.1 血清的采集和处理: 采集各样本全血4-5 mL于洁净试管中, 立即放入4 ℃静置4 h, 4 ℃ 3 000 r/min离心20 min, 剔除溶血标本, 其余分离血清, 4 ℃ 4 000 r/min再次离心5 min以去除残留的细胞碎片, 将血清每管100 mL分装至新的离心管中, 置-86 ℃冰箱保存备用. 实验前取出血清样品置冰上融化后, 4 ℃ 12 000 g离心10 min去除血清中的不溶物. 然后取10 mL血清加入20 mL U9缓冲液(9 mol/L尿素, 20 g/L CHAPS, 50 mmol/L Tris-HCl, 10 g/L DTT, pH9.0)混匀, 冰浴 300 r/min震荡30 min, 然后加入360 mL WCX-2 缓冲液(50 mmol/L NaAc, pH4.0)冰上迅速混均备用.
1.2.2 芯片的预处理和样品的检测: 将芯片装入生物芯片处理器中, 每孔加入200 mL WCX-2 缓冲液, 置于振荡器(MS1 Minishaker)300 r/min 5 min, 弃去缓冲液重复操作一次. 每孔中加入100 mL上述处理好的血清样品300 r/min振荡1 h, 甩出样品, 加入200 mL WCX-2 缓冲液, 室温300 r/min振荡洗涤芯片5 min, 共洗涤2次后, 加入200 mL HEPES(1 mmol/L, pH4.0)后立刻甩出. 拆开芯片处理器取出芯片, 干燥后立刻在每点上加SPA 0.5 mL, 干燥后重复加SPA一次.
1.2.3 数据采集: 采用PBSⅡC型质谱分析仪和Ciphergen proteinchip 3.1软件读取数据, 数据采集前用All-in-one标准蛋白质的NP20芯片校正仪器, 至误差在0.1%范围内, 采用自动采集和处理数据, 设置激光强度为210, 检测敏感度9, 优化分子质量范围为2-10 ku, 最高分子量为50 ku, 每点在不同位置共采集数据130次.
1.2.4 直肠癌血清蛋白质指纹图谱筛选模型的建立: 随机筛选56例直肠癌和40例非肿瘤对照(直肠息肉18例, 健康人22例)用于建立诊断模型. 将上述96例标本的蛋白质图谱去除背景噪音(baseline), 再分别用总离子流、在人群血清中稳定存在的6 638 u蛋白峰对所有图谱进行标准化, 以减少实验误差. 然后用Biomarker Wizard软件计算直肠癌组和非肿瘤组各蛋白峰值的差异(P值), 将此组数据进行转化, 用BPS 5.1(Biomarker Patterns systems)软件筛选并建立最佳的诊断模型.
1.2.5 诊断模型的验证: 用其余样本(直肠癌40例、直肠息肉12例, 健康人36例)的蛋白质谱对上述建立的诊断模型进行盲法验证(masked analysis), 分析该模型的特异性和灵敏度.
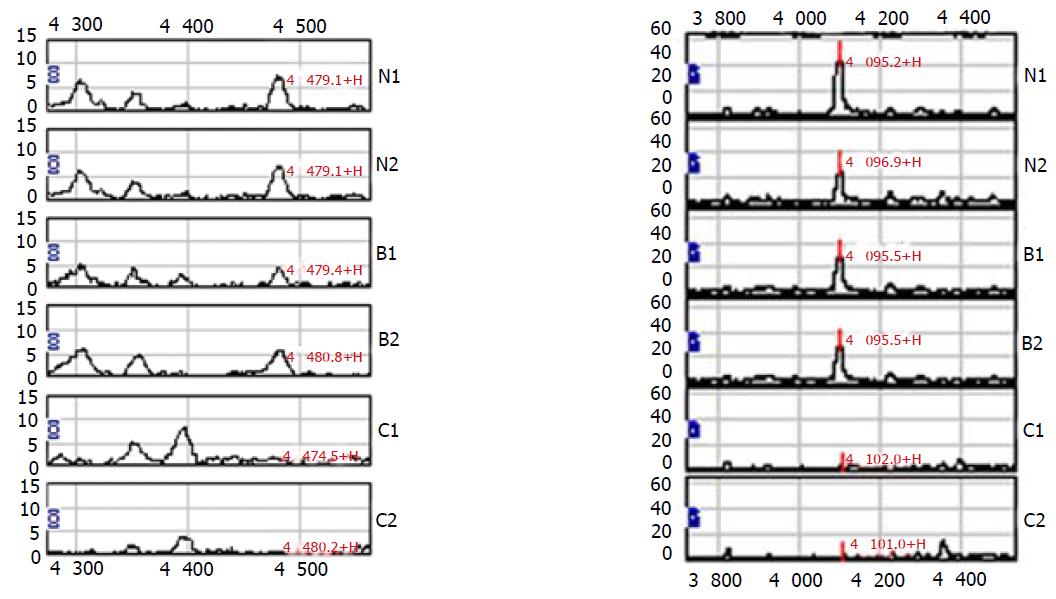
利用BPS软件对56例直肠癌和40例非直肠癌标本获得的蛋白质谱进行比较, 共发现有26个蛋白峰有显著差异(P<0.05), 部分图谱见图1.
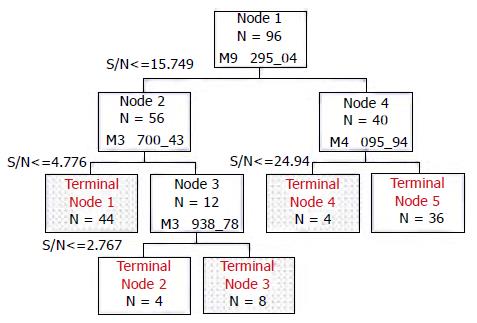
利用BPS 5.1软件对上述数据进行分析, 建立了可通过分子量为9 295, 3 730, 3 938和4 095的4个蛋白质生物标志物来区分直肠癌和非直肠癌的筛选模型(图2).
如图所示, 节点(Node)1为根节点, 如果样本中分子量为9 295 u的蛋白质相对含量≤15.749则被划分到左侧的枝节点2中, 反之则被划分到右侧的枝节点4中. 依次类推, 最终获得5个终节点(terminal node), 其中终节点1, 3, 4为直肠癌患者, 而终节点2, 5为非直肠癌.
该分类树模型在建立时, 56例直肠癌中55例区分正确, 40例非直肠癌标本中有38例被错划为癌症, 正确率为96.8%(表1).
| 组别 | n | 正确划分例数 | 正确率(%) |
| 直肠癌组 | 56 | 55 | 98.2 |
| 对照组 | 40 | 38 | 95.1 |
| 合计 | 96 | 93 | 96.8 |
用上述建立的模型对其余40例直肠癌、48例对照(直肠息肉12例, 健康人36例)的蛋白质谱进行盲法验证, 40例直肠癌中38例诊断正确, 48例对照中45例诊断正确, 此结果表明, 该方法的灵敏度为95.0%, 特异性为93.4%.
直肠癌是发生在直肠内的一种常见恶性肿瘤, 在消化道恶性肿瘤中仅次于胃癌而居第2位, 全球每年结、直肠癌新发病例数达94万, 其中近50万人死亡, 居癌症死因第3位. 近年来由于生活方式和饮食结构的改变, 直肠癌的发病率呈明显上升趋势, 发病高峰年龄提前了10岁左右[1]. 早期直肠癌治疗预后较好, 进行根治性切除后5年存活率约为50-60%, 其中Ⅰ期直肠癌可达到90%以上, 因此早期发现对于提高患者的生存率非常关键[2,9,10]. 目前早期诊断结、直肠癌的方法主要是纤维内镜检查, 该法不易为患者接受, 尤其不适合对健康人群的筛查; 检测血清中CEA、TPA和CA199等肿瘤标记物的方法敏感性与特异性还比较低, 因此, 在直肠癌病人确诊时约30-40%已发生远处转移[10-12].
SELDI-TOF-MS是近年来发展起来的有效的差异蛋白质组学研究技术, 主要结合了质谱、层析等技术原理, 能对血清、尿液等原始样品进行大规模检测, 具有高分辨力, 高重复性, 操作简便等优点, 而且所需样品量少, 在蛋白质组学中得到越来越广泛的应用, 尤其是在筛选肿瘤标志物中, 用该技术将患者与正常人或其他某种疾病患者的图谱进行比较分析, 能够发现新的、特异性肿瘤等疾病的相关蛋白, 并快速获得其蛋白质组学信息[3,13]. 目前报道用该技术已经发现了膀胱癌、卵巢癌、前列腺癌、乳腺癌、肺癌、肝癌以及肾脏肿瘤等多种肿瘤的新的肿瘤特异性标志分子[4-8,14,15]. 我们用SELDI蛋白芯片技术对新发直肠癌患者组和对照组进行蛋白质谱比较, 共发现有26个蛋白分子的相对含量显著差异; 建立的直肠癌筛选血清蛋白质指纹图谱模型利用其中分子质量为9 295, 3 730, 3 938和4 095的4个蛋白质生物标志物即可进行直肠癌的筛选, 其灵敏度为95.0%, 特异性为93.4%.
SELDI技术进行差异蛋白的比较, 其应用的成功关键是严格的平行操作和对照设计. 我们为避免不同病人因治疗方法、药物使用等因素引起血清蛋白改变的干扰, 所有直肠癌患者均选择新发、未经任何治疗的病例. 在对照设置时, 除实验组和对照组的年龄、性别等分布尽量匹配外, 非直肠癌组包含了直肠的良性病变病例, 可以避免因局部组织受损而造成的非肿瘤特异性蛋白的影响.
对于建立的直肠癌筛选血清蛋白质指纹图谱模型的验证, 我们选用了未用于建立模型的其他样本, 因此通过盲筛后获得的评价数据更科学、可靠. 尤其值得一提的是, 在盲筛时, 实验室1名健康志愿者的血清被筛选模型划为直肠癌组, 经过调查发现该志愿者3代以内的亲属中有3人患直肠癌, 因此已建议其高度重视.
为了完善所建立的模型, 我们正在将SELDI技术扩大到对治疗前后的直肠癌患者血清进行差异蛋白的研究, 此外还将增加其他肿瘤作为对照, 以使该直肠癌筛选蛋白指纹图谱模型的特异性进一步提高, 并能作为患者疗效和预后的辅助判断方法.
电编: 李琪 编辑:张海宁
| 1. | Zheng S. [Challenge and opportunity in prevention and treatment of colorectal cancer in China]. Zhejiang Daxue Xuebao Yixueban. 2004;33:375-378. [PubMed] |
| 2. | Mladen DM, Dragoslav MP, Sanja Z, Bozidar B, Snezana D. Problems in screening colorectal cancer in the elderly. World J Gastroenterol. 2003;9:2335-2337. [PubMed] [DOI] |
| 3. | Merchant M, Weinberger SR. Recent advancements in surface-enhanced laser desorption/ionization-time of flight-mass spectrometry. Electrophoresis. 2000;21:1164-1177. [PubMed] [DOI] |
| 4. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] [DOI] |
| 5. | Li J, Zhang Z, Rosenzweig J, Wang YY, Chan DW. Proteomics and bioinformatics approaches for identification of serum biomarkers to detect breast cancer. Clin Chem. 2002;48:1296-1304. [PubMed] |
| 6. | Bañez LL, Prasanna P, Sun L, Ali A, Zou Z, Adam BL, McLeod DG, Moul JW, Srivastava S. Diagnostic potential of serum proteomic patterns in prostate cancer. J Urol. 2003;170:442-446. [PubMed] [DOI] |
| 7. | Zhukov TA, Johanson RA, Cantor AB, Clark RA, Tockman MS. Discovery of distinct protein profiles specific for lung tumors and pre-malignant lung lesions by SELDI mass spectrometry. Lung Cancer. 2003;40:267-279. [PubMed] [DOI] |
| 8. | Suhr J, Eisenhardt A, Schmid KW, vom Dorp F, Otto T. [Biochips in bladder cancer research]. Urologe A. 2003;42:927-932. [PubMed] [DOI] |
| 9. | Engel J, Kerr J, Eckel R, Günther B, Heiss M, Heitland W, Jauch KW, Siewert JR, Hölzel D. Quality of treatment in routine care in a population sample of rectal cancer patients. Acta Oncol. 2005;44:65-74. [PubMed] [DOI] |
| 10. | Boyle KM, Sagar PM, Chalmers AG, Sebag-Montefiore D, Cairns A, Eardley I. Surgery for locally recurrent rectal cancer. Dis Colon Rectum. 2005;48:929-937. [PubMed] [DOI] |
| 11. | Nicolini A, Caciagli M, Zampieri F, Ciampalini G, Carpi A, Spisni R, Colizzi C. Usefulness of CEA, TPA, GICA, CA 72.4, and CA 195 in the Diagnosis of primary colorectal cancer and at its relapse. Cancer Detect Prev. 1995;19:183-195. [PubMed] |
| 12. | Yamamoto S, Akasu T, Fujita S, Moriya Y. Postsurgical surveillance for recurrence of UICC stage I colorectal carcinoma: is follow-up by CEA justified? Hepatogastroenterology. 2005;52:444-449. [PubMed] |
| 13. | Seibert V, Ebert MP, Buschmann T. Advances in clinical cancer proteomics: SELDI-ToF-mass spectrometry and biomarker discovery. Brief Funct Genomic Proteomic. 2005;4:16-26. [PubMed] [DOI] |
| 14. | Paradis V, Degos F, Dargère D, Pham N, Belghiti J, Degott C, Janeau JL, Bezeaud A, Delforge D, Cubizolles M. Identification of a new marker of hepatocellular carcinoma by serum protein profiling of patients with chronic liver diseases. Hepatology. 2005;41:40-47. [PubMed] [DOI] |
| 15. | Junker K, Gneist J, Melle C, Driesch D, Schubert J, Claussen U, Von Eggeling F. Identification of protein pattern in kidney cancer using ProteinChip arrays and bioinformatics. Int J Mol Med. 2005;15:285-290. [PubMed] [DOI] |