目的: 建立一种基于聚合酶链反应(PCR) 的简便快速、具有较高敏感性和特异性的检测乙型肝炎病毒(HBV)共价闭合环状DNA(cccDNA)的方法.
方法: 分别提取HepG2.2.15细胞内的cccDNA及培养上清中的松驰环DNA(rcDNA)样品, 试剂盒纯化; 设计2对特异性引物, 其扩增区域跨越rcDNA单链区; 设计2对非特异性引物, 扩增区域位于rcDNA双链区. 经单链特异性绿豆芽核酸酶(MBN)分别消化cccDNA及rcDNA样品; 以特异性引物和非特异性引物对消化前后的两种样品分别进行PCR扩增, 并改变PCR扩增参数如底物数量、循环次数等, 观察特异性引物能否顺利扩增消化后的cccDNA, 同时又不扩增消化后的rcDNA. HBV基因组质粒样品作为对照. 此外还采用实际乙型肝炎患者体内病毒样本检验此策略的实用性.
结果: 分别以非特异性引物和特异性引物扩增不同模板数的HBV rcDNA样品, 2对非特异性引物可扩增出模板数在102以上的HBV rcDNA样品, 2对cccDNA特异性引物也可以扩增出模板数在104以上的样品. 特异性引物在PCR反应模板数较多时将不能区分消化前的rcDNA和cccDNA. 不同数量HBV cccDNA和rcDNA模板在MBN消化前后, 分别应用非特异性引物和特异性引物进行PCR扩增, 发现不同数量的cccDNA模板分子经过MBN消化后, 仍可用特异性引物和非特异性引物扩增出相应条带; rcDNA样品经过MBN消化后, 非特异性引物可扩增出产物条带, 而特异性引物无法扩增出条带. 采用此种策略, 我们发现慢性乙肝患者血清HBV核酸样品主要成份为rcDNA, 并带有少量cccDNA, 而肝细胞内HBV核酸样品富含cccDNA, 与实际情况一致.
结论: 联合应用MBN选择性消化和cccDNA特异性引物的PCR检测法简便快速, 敏感性和特异性均较满意.
关键词: 肝炎病毒, 乙型; 共价闭合环状DNA; 聚合酶链反应; 绿豆芽核酸酶
引文著录: 汤勃, 王宇明, 刘俊, 张瑞. 改良聚合酶链反应检测HBV共价闭合环状DNA. 世界华人消化杂志 2005; 13(18): 2188-2192
Detection of hepatitis B virus cccDNA with modified polymerase chain reaction
Bo Tang, Yu-Ming Wang, Jun Liu, Rui Zhang
Bo Tang, Yu-Ming Wang, Jun Liu, Rui Zhang, Department of Infectious Diseases, Southwest Hospital of the Third Military Medical University, Chongqing 400038, China
Supported by: National Natural Science Foundation of China, No. 30230320.
Correspondence to: Bo Tang, Department of Infectious Diseases, Southwest Hospital of the Third Military Medical University, Chongqing 400038, China. tobei@163.com
Received: July 19, 2005
Revised: July 24, 2005
Accepted: July 28, 2005
Published online: September 28, 2005
AIM: To establish a simple and fast hepatitis B virus covalently closed circular DNA(cccDNA) detecting method based on polymerase chain reaction(PCR) with satisfactory sensitivity and specificity.
METHODS: The cccDNA and the relaxed circular DNA (rcDNA) were extracted from HepG2.2.15 cells and supernatant, respectively, and then purified. Two pairs of specific PCR primes were designed to cover the single strand area of rcDNA. And two pairs of non-specific PCR primes were designed to cover the double strand area of rcDNA. Before and after digested by single-strand-specific mung bean nuclease(MBN), cccDNA and rcDNA samples were amplified by specific primes and non-specific primes. Whether the digested cccDNA can be amplified by specific primes, without amplifying the digested rcDNA, was observed. The PCR parameters such as substrate amount and circulation times were changed during amplification. The HBV genome plasmid was used as control; and the HBV samples from patient with hepatitis B was used for practical test.
RESULTS: Different amounts of rcDNA template were amplified by specific and non-specific primes. More than 104 and 102 rcDNA template molecules were amplified by two pairs of specific and non-specific primes, respectively. The specific primes could not discriminate between rcDNA and cccDNA when the template molecules were overabundant. Before and after the digestion by MBN, different amounts of cccDNA were amplified by specific and non-specific primes; and after the digestion, rcDNA templates were amplified by non-specific primes, but not by specific primes. With this strategy, we found the virus samples from the serum of the patient with chronic hepatitis B contained mainly rcDNA and a small quantity of cccDNA, while the samples from hepatocytes contained mainly cccDNA.
CONCLUSION: The combination of MBN selective digestion and specific PCR amplification of the cccDNA is a simple, fast, sensitive and specific method for the detection of HBV cccDNA.
Key Words: Hepatitis B virus; Covalently closed circular DNA; Polymerase chain reaction; Mung bean nuclease
0 引言
HBV感染肝细胞的标志是HBV复制中间体, 包括共价闭合环状DNA(covalently closed circular DNA, cccDNA)、单链DNA(single-stranded DNA, ssDNA)以及HBV mRNA[1]. 通常cccDNA检测方法为Southern印迹杂交, 步骤繁琐, 检测周期长达3-4 d; 放射性标记探针可能造成环境污染和健康损伤, 而非放射标记探针敏感性又较差. 因此1996年Kock et al[2]设计特异性引物, 以PCR方法检测cccDNA取得成功, 并被广泛应用[3-5]. 然而, 正如作者所提到的那样, 这种方法在PCR反应模板较多的情况下, 其特异性难以保证. 绿豆芽核酸酶(mung bean nuclease, MBN; 也称曲霉核酸酶S1)以内切酶形式非特异性地降解核酸为核苷酸[6], 其中对于单链核酸的水解活性较双链核酸高30 000倍. 因此在合适的浓度和时间下, MBN可以切断HBV rcDNA而同时保持cccDNA的完整性. 根据上述原理, 我们改良了Kock et al的方法, 加入了MBN消化步骤, 提高了PCR反应的特异性.
1 材料和方法
1.1 材料
PCR试剂盒、绿豆芽核酸酶为美国Promega公司产品; 荧光定量PCR仪为德国Roche LightCycler, HBV核酸定量检测试剂盒为深圳匹基公司产品; 病毒核酸提取试剂盒、PCR产物纯化试剂盒为美国Clontech公司产品; 肝细胞分离系列试剂为美国Gibco公司产品. HBV全基因组质粒为全军感染病研究所自行构建. HepG2.2.15细胞为第三军医大学免疫教研室提供, 乙型肝炎患者肝组织来源于肝移植受体肝脏, 由第三军医大学第一附属医院肝胆外科提供.
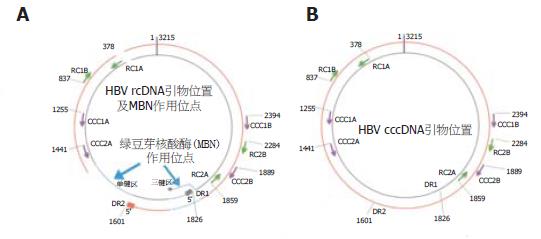
以胰酶消化培养HepG2.2.15细胞10 min, 收集细胞, 200 r/min离心3 min, 弃上清. PBS重悬, 彻底洗涤6次. Hirt法提取染色体外DNA: 加入细胞裂解液[5 g/L SDS, 10 mmol/L Tris.Cl (pH8.0), 10 mmol/L NaCl, 10 mmol/L EDTA]200 μL, 振荡混合30 s, 37 ℃水浴过夜, 加入5 mol/L NaCl(终浓度1 mol/L)50 μL, 4 ℃静置12 h, 28 000 g、4 ℃离心50 min, 弃沉淀(含细胞染色体), 以PCR产物纯化试剂盒从上清中浓缩HBV cccDNA, 按试剂盒说明书进行, 核酸样品溶解于去离子水100 μL. 以荧光定量PCR法检测样品5 μL, 计算出HBV核酸样品总拷贝数. 在50 mL高速离心管中加入300 g/L蔗糖溶液25 mL; 收集HepG2.2.15细胞培养上清, 0.22 μm过滤去除死亡细胞及残片, 取过滤液15 mL缓慢加在蔗糖溶液垫上, 230 000 g、4 ℃离心18 h, 弃上清, DNA酶Ⅰ(DNAseⅠ)反应缓冲液200 μL溶解沉淀, 加入DNAseⅠ10U消化60 min(完全降解死亡细胞释出的cccDNA), 以病毒核酸提取试剂盒提取HBV颗粒内部未被消化的rcDNA, 按试剂盒说明书进行, 核酸样品溶解于去离子水100 μL中. 以荧光定量PCR法检测样品5 μL, 计算出HBV核酸样品总拷贝数. 收集1位慢性乙型肝炎患者血清, 荧光定量PCR法确定含有高拷贝数的HBV DNA后, 使用病毒核酸提取试剂盒提取HBV核酸, 按试剂盒说明书进行, 核酸样品溶解于去离子水100 μL中; 肝细胞内HBV核酸样品: 以两步灌流法从肝移植后受体肝组织(约1 g)中分离肝细胞, PBS重悬, 彻底洗涤6次, 此后步骤同HBV cccDNA样品制备. HBV基因组从第1821核苷酸位截断后插入质粒, 常规提取纯化, 溶解于去离子水100 μL中. 以荧光定量PCR法检测样品5 μL, 计算出HBV核酸样品总拷贝数. 取10×缓冲液1 μL, 分别加入数量不等的HBV核酸模板及0.5U MBN, 去离子水补足体积至10 μL, 置于PCR仪内37 ℃孵育5 min, 立即置于冰水浴中, 加入1 mol/L Tris(pH8.0)2 μL中止反应. 所有引物均由上海生工生物工程有限公司合成, PAGE纯化. cccDNA特异性引物包括2对引物: CCC1A(正链引物)5'-CCTCTGCCGATCCAT CTGCGGAAC-3'(1255-1279, 25 bp), CCC1B(负链引物)5'-CTGCGAGGCGAGGGAGTTCTTCTTC-3'(2376-2400, 25 bp); 反应条件为95 ℃ 40 s, 72 ℃ 3 min, 35个循环, 72 ℃延伸5 min. CCC2A(正链引物)5'-CTGAATCCCGCGGACGACCC-3'(1441-1460, 21 bp), CCC2B(负链引物)5'-ACCCAAGGCACAGCTTGGAGG-3'(1867-1889, 23 bp); 反应条件为95 ℃ 40 s, 67 ℃ 40 s, 72 ℃ 1 min, 35个循环, 72 ℃延伸5 min. HBV DNA非特异性引物包括2对引物: RC1A(正链引物)5'-TGTGTCTGCGGCGTTTTATC-3'(378-397, 20 bp), RC1B(负链引物)5'-GTTTAAATGTATACCCAGAGAC-3'(816-837, 22 bp); 反应条件为95 ℃ 45 s, 54 ℃ 40 s, 72 ℃ 1 min, 35个循环, 72 ℃延伸5 min; RC2A(正链引物)5'-ACTGTTCAAGCCTCCAAGCT-3'(1859-1878, 20 bp), RC2B(负链引物)5'-AGTGCGAATCCACACTC-3'(2268-2284, 17 bp), 反应条件为95 ℃ 45 s, 51 ℃ 40 s, 72 ℃ 1 min, 35个循环, 72 ℃延伸5 min.2对cccDNA特异性引物跨越HBV基因组(rcDNA)单链区和3链区(这两区均对MBN的消化作用敏感), 如果被切开, 则特异性引物将不能扩增此种线性模板; 2对非特异性引物则分别处于P区和前C/C区, 无论完整的cccDNA、rcDNA或是被切开的rcDNA均可顺利扩增. 各引物位置及MBN作用位点见图1 A, B.
图1 HBV rcDNA、cccDNA、引物位置及MBN作用位点.
A: HBV rcDNA正链为不完整外环, 负链为完整内环 B: HBV cccDNA内外环均完整, 无MBN作用位点. MBN切割位点单链区和三链区以粗箭头和虚线表示; cccDNA特异性引物为单箭头, CCC1A/CCC1B和CCC2A/CCC2B分别为2对引物; 非特异性引物为双箭头, RC1A/RC1B和RC2A/RC2B分别为2对引物. DR1: 直接重复序列1; DR2: 直接重复序列2.
2 结果
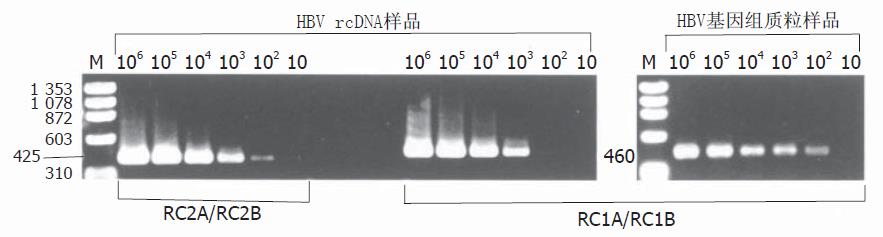
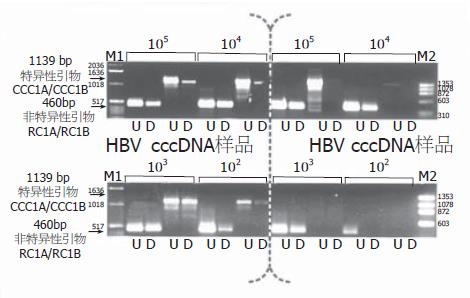
对非特异性引物可扩增出模板数在102以上的HBV rcDNA样品(图2); 而对于同样的rcDNA模板, 2对cccDNA特异性引物也可以扩增出模板数在104以上的样品(图3). 由此可知, 特异性引物的"特异性"是相对的, 在PCR反应模板数较多时将不能区分rcDNA和cccDNA. 此结果和Kock et al的一致. HBV全基因组质粒是在第1821核苷酸位切开后插入质粒, 因此P区引物RC1A/RC1B可以扩增出模板数在102以上的样品, 而2对特异性引物在任何模板数下均不能扩增出目标条带(图2, 3). 不同数量HBV cccDNA和rcDNA模板在MBN消化前后, 分别应用非特异性引物RC1A/RC1B和特异性引物CCC1A/CCC1B进行PCR扩增, 从105~102不同数量的cccDNA模板分子经过MBN消化后, 仍可用特异性引物CCC1A/CCC1B和非特异性引物RC1A/RC1B扩增出相应条带, 惟亮度较未经消化的模板有不同程度的降低, 表明过量的MBN降解了部分cccDNA分子. 见图4垂直虚线左半部分. 而rcDNA样品经过MBN消化后, 如果模板数>103, 则非特异性引物RC1A/RC1B可扩增出产物条带, 亮度较未经消化的模板为低; 对于cccDNA特异性引物CCC1A/CCC1B, 模板数即使为105, 经MBN消化后亦无法扩增任何相应条带. 见图4垂直虚线右半部分.
图2 非特异性引物RC2A/RC2B和RC1A/RC1B分别扩增不同数量的HBV rcDNA及HBV基因组质粒模板电泳图.
M: ΦX174 DNA/Hae Ⅲ相对分子质量标准; 106-10: PCR体系中分别加入106-10个模板分子.
图3 特异性引物CCC1A/CCC1B和CCC2A/CCC2B分别扩增不同数量的HBV rcDNA及HBV基因组质粒模板电泳图.
M1: 1kb DNA ladder; M2: ΦX174 DNA/HAE Ⅲ相对分子质量标准; 106-10: PCR体系中分别加入106-10个模板分子板分子.
图4 不同数量HBV cccDNA和rcDNA模板经MBN消化后以非特异性引物RC1A/RC1B和特异性引物CCC1A/CCC1B进行PCR扩增后电泳图.
垂直黑色虚线左半部分为HBV cccDNA模板样品, 数量为105-102; 垂直黑色虚线右半部分为HBV rcDNA模板样品, 数量为105-102. M1: 1kb DNA ladder; M2: ΦX174 DNA/HAE Ⅲ相对分子质量标准; U: 未经MBN消化的样品; D: 经过MBN消化的样品.
将血清和肝细胞内HBV核酸样品分别按1, 1:10, 1:100去离子水稀释, 各取5μL进行MBN消化和PCR扩增. 血清HBV核酸样品不稀释时, MBN消化前后均可扩增出cccDNA特异性条带; 10倍稀释样品消化前可见条带, 消化后无特异性条带; 100倍稀释样品无论是否消化, 均不能扩增出相应条带. 提示血清HBV核酸样品主要成份为rcDNA, 并带有少量cccDNA. 而富含cccDNA的肝细胞内HBV核酸样品即使在100倍稀释后, MBN消化也未能完全阻止PCR阳性条带的出现.
3 讨论
HBV入侵靶细胞后, 其病毒复制周期的第一步即为松驰环型基因组(rcDNA)转变为共价闭合环状形式(cccDNA), 然后以此为模板转录出数种HBV mRNA[10]. 同时cccDNA继续在细胞核内累积, 形成一个稳定的cccDNA池. cccDNA的存在和数量不仅是HBV感染靶细胞的特征性标志, 也是抗HBV药物疗效的重要指标[11-16]. 近年来国内外研究者试图以PCR法代替传统Southern杂交检测cccDNA, 主要原理均为利用rcDNA为不完整双环, 设计跨越单链区和三链区的引物而达到只扩增完整cccDNA的目的[17-20](图1). 然而, 正如Kock et al和我们的实验结果表明的那样, 单纯使用此种引物, 在PCR模板数量超过104时, 其扩增的选择性即不存在(图2, 3), 主要原因可能是负链引物引导的新生链有时能够克服三链区及末端蛋白的阻碍而继续延伸[2]. 故若能切断rcDNA的单链区而保持cccDNA的完整, 则可以保证特异性引物选择性扩增的可靠性[21,22].
MBN是单链核酸水解酶, 可利用MBN切断HBV rcDNA单链区和(或)三链区, 使rcDNA线性化, 从而不能被跨越此区域的特异性引物所扩增. 相反, cccDNA则可以抵抗MBN的消化而保持完整双环, 继续作为PCR模板而被选择性扩增. 实验中我们发现, 105个rcDNA分子经MBN处理后, 特异性引物不能扩增任何产物. 本科室构建的HBV基因组质粒是在HBV基因组第1821位核苷酸切开后单拷贝插入质粒, 因此理论上同样不能被cccDNA特异性引物扩增出相应条带, 图3显示的实验结果表明的确如此. 而另一方面, 低至100个cccDNA分子则因抵抗了MBN消化, 得以通过特异性引物扩增(图4).
一般认为, 慢性乙型肝炎患者血清内HBV核酸主要为Dane颗粒中的rcDNA, 同时可能包含少量由于肝细胞坏死释放的cccDNA; 而乙型肝炎患者肝细胞内的HBV核酸主要为cccDNA[23-25], 我们提取这两种来源的核酸, 系列稀释后进行MBN消化前后的特异性引物PCR扩增, 验证本方法的临床实用性[26,27]. 结果表明, 不加稀释的血清HBV核酸样品经消化、PCR扩增后可见cccDNA条带, 同样处理的10倍或者100倍稀释样品后则不可见cccDNA条带. 相反, 肝细胞内来源的HBV核酸即使稀释100倍, 仍可以抵抗MBN消化而出现cccDNA特异性条带. 这一结果符合理论预测和临床实际[28]. 实验所用的HBV rcDNA和cccDNA样品分别提取自HepG2.2.15细胞株培养上清和细胞内. rcDNA样品提取过程中, 先过滤、纯化HBV Dane颗粒, 再以过量的DNaseⅠ完全降解Dane颗粒外可能存在的DNA, 最后提取Dane颗粒内的HBV基因组核酸应为较纯净的rcDNA[27]. cccDNA样品的纯净则得益于如下措施: 胰酶化促使黏附于细胞表面的HBV颗粒脱离; PBS洗涤6次; 裂解细胞时没有按常规加入蛋白酶K, 使rcDNA保持在核衣壳内而没有随细胞染色体外HBV cccDNA一道被提取. MBN消化反应条件的优化极为重要. 虽然MBN对于单链核酸的活性较双链核酸高约30 000倍, 若酶的浓度>14 MU/g模板DNA, MBN同样可以在双链DNA上造成缺口并降解. 用于MBN消化的HBV核酸分子均为pg数量级(1 pg HBV核酸≈105个HBV基因组分子)[17,29,30], 因此即使每次消化反应使用0.5 U的MBN也是绝对过量的, 正如实验中所看到的那样, 各种数量的样品经MBN消化后, 所有引物扩增的PCR条带亮度均有不同程度的降低, 提示模板数量减少. 我们经过多次实验, 确定10 μL消化体系内加入0.5 U酶、孵育5 min后立即中止反应可以避免cccDNA分子的完全降解. 总之, 我们改良的这种HBV cccDNA PCR检测法简便易行, 从样品提取到获得电泳照片只需6 h, 同时联合了PCR法的敏感性和MBN消化的选择性, 敏感性和特异性均较好. 主要缺点在于各公司出品的MBN之间不完全一致, MBN消化不当所致的假阴性率和PCR反应的假阳性率较高, 需要预先优化反应条件. 若用我们已反复优化的方法, 可望获得较满意效果.