修回日期: 2004-04-12
接受日期: 2004-04-15
在线出版日期: 2004-07-15
Sp1/Krüppel样转录因子(Sp1-like and Krüppel-like transcription factors, Sp1/ KLFs)是参与真核细胞转录的高度相关锌指蛋白, 以细胞和启动子特异性方式通过调控富含GC启动子的基因表达, 参与调节细胞功能如细胞增生、凋亡、分化和肿瘤形成.
引文著录: 张安平, 张连阳. Sp1/Krüppel样转录因子家族研究进展. 世界华人消化杂志 2004; 12(7): 1641-1645
Revised: April 12, 2004
Accepted: April 15, 2004
Published online: July 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(7): 1641-1645
- URL: https://www.wjgnet.com/1009-3079/full/v12/i7/1641.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i7.1641
启动基因转录是非常复杂的过程, 与Sp1/Krüppel样转录因子家族相关的序列特异性DNA连接蛋白在调控基因转录中发挥作用. 在Sp1/Krüppel样转录因子家族中Sp1首先被克隆和纯化, 在SV40启动子中Sp1通过3个Cys2His2锌指与富含GC的位点特异性结合. 在果蝇属胚胎调节因子Krüppel中也存在类似的DNA结合区, 随后具有与Sp1高度相似锌指结构的其他转录因子相继发现, 形成一个新的Sp1/Krüppel样转录因子(KLFs)家族[1-7]. 他们存在于从线虫属到人类的种属中, 参与基因转录复制[8-10]. 在人类存在21个Sp1/KLFs基因, 大鼠中有17个Sp1/KLFs蛋白与人同源, 而小鼠则有11个同源(表1). 目前对于人类和其他物种的Sp1/KLFs蛋白的功能和结构尚无系统比较, Sp1/KLFs家族与各种富含GC的DNA元件结合而调节转录、细胞生长和控制生物形态发育, 除了KLF2 (LKLF)和KLF1 (EKLF)外, 编码Sp1样蛋白的基因是随意分布在整个基因组中, 其产物功能独立, 这与其他重要的进化调节因子如Hox转录因子、KRAB因子截然不同[11-13]. 根据序列和结构相似性将Sp1/KLFs家族分为三个亚家族(图1A). 与Sp1高度相关的命名为Sp1-6形成亚家族I, 其他的组成另外两个亚家族II和III, 依次命名为KLF1-16.
| 蛋白 | KLF 成员 | 种类 | 染色体 定位 | 转录活性 (功能区域) | 表达方式 | 相互作用的共激活子和/或共抑制因子 | 细胞功能 |
| Sp1 | - | 人、小鼠、 大鼠、果蝇属 | 12q13 | 激活因子(富含Q区) | 普遍表达 | CRSP,p300/CBP, TAFII130 | 胚胎发育 |
| Sp2 | - | 17q21 | 未知(富含Q区) | 未知 | 未知 | ||
| Sp3 | - | 2q31 | 激活因子或抑制因子(富含Q区) | 普遍表达 | 未知 | 未知 | |
| Sp4 | - | 人、小鼠、 大鼠 | 7p15 | 激活因子(富含Q区) | 脑 | 未知 | 出生后存活、 雄性生殖 |
| mSp5 | - | 小鼠 | 未知 | 未知 | 普遍表达 | 未知 | 未知 |
| Sp6 | KLF14 | 人、小鼠 | 17q21 | 激活因子 | 普遍表达 | 未知 | 未知 |
| EKLF | KLF1 | 人、小鼠 | 19p13 | 激活因子(酸性区) | 红细胞、 肥大细胞 | p300/CB、PCAF、SWI/SNF、mSin3A | 红细胞形成 |
| LKLF KLF2 | LLF2 | 人、小鼠 | 19p13 | 激活因子(酸性区) | 肺、血管、 淋巴细胞 | 未知 | 血管和肺的发育、T细胞存活 |
| BKLF | KLF3 | 人、小鼠、 大鼠 | 4p14 | 激活因子/抑制因 子(PVALS/T基序) | 形成红细胞的 组织、脑组织 | CtBP2 | 未知 |
| GKLF | KLF4 | 人、小鼠、 大鼠、斑马鱼 | 9q31 | 激活因子和/或抑制因子(酸性区) | 消化道 | p300/CBP | 抑制增生、存活 |
| IKLF | KLF5 | 人、小鼠、 大鼠 | 13q21 | 激活因子 | 消化道、 上皮组织 | 未知 | 细胞生长 |
| CPBP | KLF6 | 人、小鼠 | 10p15 | 激活因子 | 谱表表达 | 未知 | 肿瘤因子 |
| UKLF | KLF7 | 人、小鼠 | 2q32 | 激活因子(酸性区) | 普遍表达 | 未知 | 细胞周期停滞 |
| BKLF3 | KLF8 | 人 | Xp11 | 抑制因子 | 普遍表达 | CtBP2 | 未知 |
| BTEB1 | KLF9 | 人、小鼠 | 9q13 | 激活因子/抑制因子(SID) | 普遍表达 | 神经轴突生长 | |
| TIEG1 | KLF10 | 人 | 8q22 | 抑制因子 | 普遍表达 | mSin3A | 凋亡、抑制增生 |
| TIEG2/ FKLF | KLF11 | 人 | 2p25 | 激活因子和/或抑制因子(SID,R2, R3) | 普遍表达 | mSin3A | 抑制增生 |
| AP-2rep | KLF12 | 13q21 | 抑制因子 | 抑制因子 | 脑. 肾、肝、肺 | CtBP1 | 未知 |
| BTEB3/RFLAT-1/FKLF-2 | KLF13 | 人、小鼠、 大鼠 | 15q12 | 激活因子/抑制因子 (SID, R2、R3) | 普遍表达 | mSin3、PCAF、 p300/CBP | 抑制增生 |
| KKLF | KLF15 | 人、小鼠、 大鼠 | 3q13 | 抑制因子 | 普遍表达 | 未知 | 未知 |
| BTEB4 mDRRF | KLF16 | 人、小鼠 | 19q13 | 抑制因子(SID) | 普遍表达 | mSin3A | 致癌物的 新陈代谢 |
| BTEB5 | - | 人 | 7 | 未知 | 未知 | 未知 | 未知 |
作为具有功能的位点特异性转录因子至少有DNA结合区、核定位信号和转录调控区. Sp1/KLFs家族蛋白的主要特征是含有高度保守的DNA结合区(同源性超过65%), 在羧基端存在3个Cys2His2锌指(图1B), 锌指可能参与蛋白之间的相互作用[14]. Sp1/KLFs蛋白家族的氨基端变化较大, 含有转录激活区或转录抑制区, 在锌指内或与其相邻区存在核定位序列[15-16].
与Sp1/KLFs家族成员C末端区相邻的是具有81个氨基酸的DNA结合区, 由3个锌指组成, 锌指均含有Cys2His2的共同序列C-X2-5-C-X3-(F/Y)-X5-ψ- X2-H-X3-5-H (X代表任何氨基酸, ψ是疏水残基)[17]. Sp1与该家族的其他成员的氨基酸相似性至少在66.7% (图1B). 在前两个锌指中各含有23个氨基酸而第3个锌指则含有21个氨基酸. 锌指之间有7个氨基酸的高度保守序列TGE(R/K)(P/k/r)(F/y)X连接.
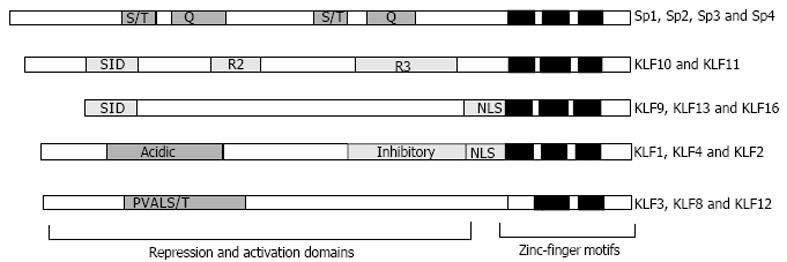
尽管Sp1/KLFs家族成员均具有高度相似的DNA结合活性, 但功能各异, 如Sp1是最为有效的转录激活因子之一[7,18], KLF11(TIEG2)则是转录抑制因子[19-20]. 该家族成员是激活因子或是抑制因子, 主要依赖与他们结合的启动子以及相互作用的共调节因子的功能. 通过氨基端的特异激活区和抑制区而与共激活或共抑制因子作用发挥调节转录的功能(图2). Sp1和Sp3氨基端的富含谷氨酰胺区与转录因子TAFII130相互作用而使转录激活[21-26]. KLF3 (BKLF), KLF8 (BKLF3)和KLF12 (AP2-rep)氨基端的PVALS/T基序与属于C末端结合蛋白(CtBP)家族的抑制因子结合而抑制转录[4,27]. 同属亚家族III的TIEG蛋白(TIEG1/TIEG2, KLF10/KLF11)和 BTEB蛋白 (BTEB1, BTEB3/ BTEB4, KLF9, KLF13/KLF16)均有保守的抑制性序列, 他们共有一个保守基序即Sin3相关的-螺旋, 他与转录抑制因子Mad1的SID区相互作用, 这些SID区通过与组蛋白脱乙酰转移酶共抑制因子复合物mSin3A相互作用而介导转录抑制[20]. 而某些细胞信号事件可能改变这种功能, 如在KLF11 SID区相邻的四个残基磷酸化参与细胞外的调节激酶2(ERK2)信号, 干扰与mSin3A的相互作用而导致抑制功能丧失[28].
该家族大部分蛋白与调节生长或发育相关(表1). 如敲除大鼠Sp1基因则导致发育迟缓并在出生后早期死亡. KLF1和 KLF2分别在红细胞和T淋巴细胞中特异性表达, 敲除 KLF1则导致在红细胞形成出现选择性缺陷, 而敲除KLF2则与T细胞的静止和存活相关[29]. Nakamura et al[30]认为Sp1/KLFs家族成员可能参与毛囊、肢芽等细胞的分化. Sp1在恶性肿瘤中的表达和活性比在良性肿瘤中要高, 认为Sp1可能参与肿瘤的形成[31]. KLF6与人脑垂体肿瘤的发生相关, 新近研究表明他还可能参与直肠癌、脑星型胶质瘤和鼻咽癌的发生[32]. 与之相似的是KLF4可促进一些肿瘤细胞如乳腺癌的生长[33-34], 其作用与一些肿瘤抑制因子被下调相似[35], 这些基因在不同细胞的肿瘤形成转化中发挥不同的作用. 最近有报道KLF6可能作为前列腺癌肿瘤抑制因子而存在[36], 当KLF6的转录调控区发生突变时其抑制细胞生长的作用降低.
Sp1/Krüppel样转录因子家族成员与共抑制/激活因子相互作用参与转录调节, 研究表明组蛋白乙酰化和去乙酰化可能是Sp1/KLFs蛋白是激活/抑制因子的功能转换开关. 组蛋白去乙酰化酶特异性抑制因子HDAC1的过度表达通过TSA可明显抑制KLF4启动子的转录活性[37]. 在大肠癌细胞系中KLF4的5'不翻译区的超甲基化可能是KLF4作为肿瘤抑制因子的原因之一[38], KLF4也可通过抑制组氨酸脱乙酰化酶的启动子发挥抑制因子的功能[39]. KLF13可激活如SV40, 趋化因子 RANTES, β-球蛋白的启动子[22,40-41], 却抑制细胞色素P450 CYP1A1[34], 这表明KLF13部分反式调节活性是启动子依赖性. Song et al[16]研究表明CREB结合蛋白(CBP)共激活因子和他的同源物 p300, CBP/p300相关因子(PCAF)结合, KLF13锌指区乙酰化, 可使KLF13 DNA结合活性活化, 尽管在KLF13氨基区没有相关的激活因子存在, 但该区域仍可能具有激活区的功能. 最近发现的三个独特抑制区SID, R2和R3[20], 均存在于KLF13的氨基区, 可与mSin3A组氨酸脱乙酰化共抑制因子复合物相互作用, 使KLF13抑制CYP1A1的表达. 而KLF13的SID残基与具有激活功能的氨基区重叠, 则KLF13以激活因子存在[16]. 运用转基因技术可确定与Sp1/KLFs氨基区相互作用的新结构[42].
总之, 近年对Sp1/Krüppel样转录因子的研究已取得很大进展, 但是他们的精确结构、生化功能, 以及氨基区的转录调节功能是目前研究的热点. 而整个家族成员形成一个功能网络相互重叠, 因此该家族成员在转录调节中相互关系, 以及在人类疾病如肿瘤发生中的作用尚待进一步研究.
编辑: N/A
| 1. | Turner J, Crossley M. Mammalian Krüppel-like transcription factors: more than just a pretty finger. Trends Biochem Sci. 1999;24:236-240. [PubMed] [DOI] |
| 2. | Black AR, Black JD, Azizkhan-Clifford J. Sp1 and krüppel-like factor family of transcription factors in cell growth regulation and cancer. J Cell Physiol. 2001;188:143-160. [PubMed] [DOI] |
| 3. | Dang DT, Pevsner J, Yang VW. The biology of the mammalian Krüppel-like family of transcription factors. Int J Biochem Cell Biol. 2000;32:1103-1121. [PubMed] [DOI] |
| 4. | Bieker JJ. Krüppel-like factors: three fingers in many pies. J Biol Chem. 2001;276:34355-34358. [PubMed] [DOI] |
| 5. | Cook T, Gebelein B, Urrutia R. Sp1 and its likes: biochemical and functional predictions for a growing family of zinc finger transcription factors. Ann N Y Acad Sci. 1999;880:94-102. [PubMed] [DOI] |
| 6. | Philipsen S, Suske G. A tale of three fingers: the family of mammalian Sp/XKLF transcription factors. Nucleic Acids Res. 1999;27:2991-3000. [PubMed] [DOI] |
| 8. | Ossipova O, Stick R, Pieler T. XSPR-1 and XSPR-2, novel Sp1 related zinc finger containing genes, are dynamically expressed during Xenopus embryogenesis. Mech Dev. 2002;115:117-122. [PubMed] [DOI] |
| 9. | Saeed SA, Kendall PA, McDonald-Gibson WJ, Collier HO. Haptoglobin concentration and the activity of an endogenous inhibitor of prostaglandin synthetase in blood plasma or serum. Biochem Soc Trans. 1978;6:1166-1169. [PubMed] [DOI] |
| 10. | Oates AC, Pratt SJ, Vail B, Yan Yl, Ho RK, Johnson SL, Postlethwait JH, Zon LI. The zebrafish klf gene family. Blood. 2001;98:1792-1801. [PubMed] [DOI] |
| 11. | Patel NH, Prince VE. Beyond the Hox complex. Genome Biol. 2000;1:REVIEWS1027. [PubMed] [DOI] |
| 12. | Ferrier DE, Holland PW. Ancient origin of the Hox gene cluster. Nat Rev Genet. 2001;2:33-38. [PubMed] [DOI] |
| 13. | Eichler EE, Hoffman SM, Adamson AA, Gordon LA, McCready P, Lamerdin JE, Mohrenweiser HW. Complex beta-satellite repeat structures and the expansion of the zinc finger gene cluster in 19p12. Genome Res. 1998;8:791-808. [PubMed] [DOI] |
| 14. | Zhang W, Kadam S, Emerson BM, Bieker JJ. Site-specific acetylation by p300 or CREB binding protein regulates erythroid Krüppel-like factor transcriptional activity via its interaction with the SWI-SNF complex. Mol Cell Biol. 2001;21:2413-2422. [PubMed] [DOI] |
| 15. | Shields JM, Yang VW. Two potent nuclear localization signals in the gut-enriched Krüppel-like factor define a subfamily of closely related Krüppel proteins. J Biol Chem. 1997;272:18504-18507. [PubMed] [DOI] |
| 16. | Song A, Patel A, Thamatrakoln K, Liu C, Feng D, Clayberger C, Krensky AM. Functional domains and DNA-binding sequences of RFLAT-1/KLF13, a Krüppel-like transcription factor of activated T lymphocytes. J Biol Chem. 2002;277:30055-30065. [PubMed] [DOI] |
| 17. | Wolfe SA, Nekludova L, Pabo CO. DNA recognition by Cys2His2 zinc finger proteins. Annu Rev Biophys Biomol Struct. 2000;29:183-212. [PubMed] [DOI] |
| 18. | Ryu S, Zhou S, Ladurner AG, Tjian R. The transcriptional cofactor complex CRSP is required for activity of the enhancer-binding protein Sp1. Nature. 1999;397:446-450. [PubMed] [DOI] |
| 19. | Cook T, Gebelein B, Belal M, Mesa K, Urrutia R. Three conserved transcriptional repressor domains are a defining feature of the TIEG subfamily of Sp1-like zinc finger proteins. J Biol Chem. 1999;274:29500-29504. [PubMed] [DOI] |
| 20. | Zhang JS, Moncrieffe MC, Kaczynski J, Ellenrieder V, Prendergast FG, Urrutia R. A conserved alpha-helical motif mediates the interaction of Sp1-like transcriptional repressors with the corepressor mSin3A. Mol Cell Biol. 2001;21:5041-5049. [PubMed] [DOI] |
| 21. | Asano H, Li XS, Stamatoyannopoulos G. FKLF, a novel Krüppel-like factor that activates human embryonic and fetal beta-like globin genes. Mol Cell Biol. 1999;19:3571-3579. [PubMed] [DOI] |
| 22. | Asano H, Li XS, Stamatoyannopoulos G. FKLF-2: a novel Krüppel-like transcriptional factor that activates globin and other erythroid lineage genes. Blood. 2000;95:3578-3584. [PubMed] |
| 23. | Imataka H, Sogawa K, Yasumoto K, Kikuchi Y, Sasano K, Kobayashi A, Hayami M, Fujii-Kuriyama Y. Two regulatory proteins that bind to the basic transcription element (BTE), a GC box sequence in the promoter region of the rat P-4501A1 gene. EMBO J. 1992;11:3663-3671. [PubMed] |
| 24. | Kobayashi A, Sogawa K, Imataka H, Fujii-Kuriyama Y. Analysis of functional domains of a GC box-binding protein, BTEB. J Biochem. 1995;117:91-95. [PubMed] [DOI] |
| 25. | Majello B, De Luca P, Lania L. Sp3 is a bifunctional transcription regulator with modular independent activation and repression domains. J Biol Chem. 1997;272:4021-4026. [PubMed] [DOI] |
| 26. | Gill G, Pascal E, Tseng ZH, Tjian R. A glutamine-rich hydrophobic patch in transcription factor Sp1 contacts the dTAFII110 component of the Drosophila TFIID complex and mediates transcriptional activation. Proc Natl Acad Sci U S A. 1994;91:192-196. [PubMed] [DOI] |
| 27. | van Vliet J, Turner J, Crossley M. Human Krüppel-like factor 8: a CACCC-box binding protein that associates with CtBP and represses transcription. Nucleic Acids Res. 2000;28:1955-1962. [PubMed] [DOI] |
| 28. | Ellenrieder V, Zhang JS, Kaczynski J, Urrutia R. Signaling disrupts mSin3A binding to the Mad1-like Sin3-interacting domain of TIEG2, an Sp1-like repressor. EMBO J. 2002;21:2451-2460. [PubMed] [DOI] |
| 29. | Kuo CT, Veselits ML, Leiden JM. LKLF: A transcriptional regulator of single-positive T cell quiescence and survival. Science. 1997;277:1986-1990. [PubMed] [DOI] |
| 30. | Nakamura T, Unda F, de-Vega S, Vilaxa A, Fukumoto S, Yamada KM, Yamada Y. The Krüppel-like factor epiprofin is expressed by epithelium of developing teeth, hair follicles, and limb buds and promotes cell proliferation. J Biol Chem. 2004;279:626-634. [PubMed] [DOI] |
| 31. | Kumar AP, Butler AP. Enhanced Sp1 DNA-binding activity in murine keratinocyte cell lines and epidermal tumors. Cancer Lett. 1999;137:159-165. [DOI] |
| 32. | Vax VV, Gueorguiev M, Dedov II, Grossman AB, Korbonits M. The Krüppel-like transcription factor 6 gene in sporadic pituitary tumours. Endocr Relat Cancer. 2003;10:397-402. [PubMed] [DOI] |
| 33. | Foster KW, Frost AR, McKie-Bell P, Lin CY, Engler JA, Grizzle WE, Ruppert JM. Increase of GKLF messenger RNA and protein expression during progression of breast cancer. Cancer Res. 2000;60:6488-6495. [PubMed] |
| 34. | Foster KW, Ren S, Louro ID, Lobo-Ruppert SM, McKie-Bell P, Grizzle W, Hayes MR, Broker TR, Chow LT, Ruppert JM. Oncogene expression cloning by retroviral transduction of adenovirus E1A-immortalized rat kidney RK3E cells: transformation of a host with epithelial features by c-MYC and the zinc finger protein GKLF. Cell Growth Differ. 1999;10:423-434. [PubMed] |
| 35. | Adam PJ, Regan CP, Hautmann MB, Owens GK. Positive- and negative-acting Kruppel-like transcription factors bind a transforming growth factor beta control element required for expression of the smooth muscle cell differentiation marker SM22alpha in vivo. J Biol Chem. 2000;275:37798-37806. [PubMed] [DOI] |
| 36. | Narla G, Heath KE, Reeves HL, Li D, Giono LE, Kimmelman AC, Glucksman MJ, Narla J, Eng FJ, Chan AM. KLF6, a candidate tumor suppressor gene mutated in prostate cancer. Science. 2001;294:2563-2566. [PubMed] [DOI] |
| 37. | Chen ZY, Rex S, Tseng CC. Krüppel-like factor 4 is transactivated by butyrate in colon cancer cells. J Nutr. 2004;134:792-798. [PubMed] |
| 38. | Zhao W, Hisamuddin IM, Nandan MO, Babbin BA, Lamb NE, Yang VW. Identification of Krüppel-like factor 4 as a potential tumor suppressor gene in colorectal cancer. Oncogene. 2004;23:395-402. [PubMed] [DOI] |
| 39. | Ai W, Liu Y, Langlois M, Wang TC. Kruppel-like factor 4 (KLF4) represses histidine decarboxylase gene expression through an upstream Sp1 site and downstream gastrin responsive elements. J Biol Chem. 2004;279:8684-8693. [PubMed] [DOI] |
| 40. | Song A, Chen YF, Thamatrakoln K, Storm TA, Krensky AM. RFLAT-1: a new zinc finger transcription factor that activates RANTES gene expression in T lymphocytes. Immunity. 1999;10:93-103. [PubMed] [DOI] |
| 41. | Martin KM, Cooper WN, Metcalfe JC, Kemp PR. Mouse BTEB3, a new member of the basic transcription element binding protein (BTEB) family, activates expression from GC-rich minimal promoter regions. Biochem J. 2000;345 Pt 3:529-533. [PubMed] [DOI] |
| 42. | Li JZ, Chen X, Yang H, Wang SL, Gong XL, Feng H, Guo BY, Yu L, Wang ZG, Fu JL. Establishment of transgenic mice carrying gene encoding human zinc finger protein 191. World J Gastroenterol. 2004;10:264-267. [PubMed] |