修回日期: 2004-04-10
接受日期: 2004-04-27
在线出版日期: 2004-07-15
目的: 探讨转入靶向survivin的siRNA对肝癌细胞生物学行为的影响.
方法: 设计、合成一对survivin编码基因的反向重复序列, 中间间隔9个核苷酸序列, 通过定向克隆至载体PSilencer2.1, 构建siRNA真核表达载体, 经稳定转染HepG2细胞后对转基因后肿瘤细胞的生物学行为进行观察.
结果: 测序证实siRNA真核表达载体构建成功. 通过RT-PCR及流式细胞术检测, siRNA在mRNA及蛋白质水平抑制survivin基因表达分别达73%和75%. 转染细胞生长速度明显减慢, 凋亡率增加15倍. 裸鼠体内成瘤率下降75%, 9-30 d肿瘤体积分别为0.10±0.01 cm3, 0.30±0.03 cm3, 0.39±0.11 cm3, 0.45±0.13 cm3, 0.49±0.07 cm3, 0.58±0.01 cm3, 0.60±0.10 cm3, 0.65±0.07 cm3, 与对照组相比差异显著(P<0.01).
结论: 靶向survivin的siRNA能有效降低目的基因的表达并能在体内体外抑制肝癌细胞株HepG2的生长. 为进一步逆转肿瘤细胞的耐药性提供了理论指导.
引文著录: 卢昕, 胡安斌, 张勇, 陈立波, 郑启昌. 靶向survivin的siRNA对肝癌细胞生物学行为的影响. 世界华人消化杂志 2004; 12(7): 1534-1538
Revised: April 10, 2004
Accepted: April 27, 2004
Published online: July 15, 2004
AIM: To study the influence of siRNA targeting survivin on the biological behavior of hepatocellular carcinoma (HCC).
METHODS: One pair of 21 bp reverse repeated motifs of survivin target sequence with 9 spacer were synthesized and inserted into plasmid psilencer 2.1 to generate siRNA eukaryotic expression vector. After stable transfection into HepG2 cells, the biological behaviors of the survivin siRNA transfected HCC cells were observed.
RESULTS: The recombinant plasmid Psilence(+)-survivin was successfully constructed. survivin mRNA and protein expression inhibitory ratio reached 73% and 75% respectively. It was demonstrated that transfected cells with survivin siRNA were inhibited on growth and increased on apoptosis. Subsequent study in nude mouse model demonstrated lower succeeding rate in cells transfected with survivin siRNA and the tumor size from 9-30 day was 0.10±0.01 cm3, 0.30±0.03 cm3, 0.39±0.11 cm3, 0.45±0.13 cm3, 0.49±0.07 cm3, 0.58±0.01 cm3 0.60±0.10 cm3, and 0.65±0.07 cm3 respectively. The difference was obvious (P < 0.01).
CONCLUSION: siRNA targeting survivin gene can specifically suppress survivin expression in HepG2 cells and inhibit tumor cells growth both in vivo and in vitro. This provides a theory basis to reverse the drug resistance in tumor cells.
- Citation: Lu X, Hu AB, Zhang Y, Chen LB, Zheng QC. Effect of siRNA targeting survivin gene on biological behavior of hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2004; 12(7): 1534-1538
- URL: https://www.wjgnet.com/1009-3079/full/v12/i7/1534.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i7.1534
肝癌是我国常见的恶性肿瘤之一. 深入了解肝癌发病的分子机制以寻求新的治疗方法是提高肝癌整体疗效的关键. 目前研究已表明细胞凋亡障碍是肿瘤发生、发展过程中的重要事件[1]. 肿瘤细胞对凋亡的抵抗性是他们在缺氧及营养缺乏等异常生长环境下得以生存的重要原因[2]. 抑制肿瘤细胞内与凋亡抵抗相关基因的表达不仅可诱导肿瘤细胞的凋亡而且还可选择性增加肿瘤细胞对以凋亡为机制的治疗方法的敏感性, 从而提高肿瘤病人的总体生存率[3]. survivin基因是迄今为止克隆出的最小凋亡抑制蛋白家族成员, 可通过抑制caspase级链反应下游的caspase3, caspase7发挥其抗凋亡作用[4-5]. 现在研究已证实survivin基因参与了肝癌的发生与发展, 并且还与化疗的耐药性相关[6]. 降低survivin基因在肝癌细胞中的表达不仅可诱导肿瘤细胞的凋亡、抑制肿瘤细胞的生长, 还可增强其对化疗的敏感性, 从而提高肝癌治疗的整体疗效. 因此将survivin基因作为治疗肝癌的候选靶基因在理论上是可行的.
RNA干扰(RNA interference, RNAi)技术是近年来发展起来的新技术, 具有抑制作用强、稳定性高、细胞摄取相对容易等优点[7-8]. 目前RNAi在基因功能、抗肿瘤和抗病毒基因治疗等方面的研究已取得广泛应用[9-10]. 在此我们利用转录载体Psilencer2.1构建靶向survivin的siRNA真核表达载体以研究其对肝癌细胞生物学的影响, 从而为肝癌的基因治疗提供新的方法.
肝癌细胞株HepG2由同济医院肝脏外科中心朱虹博士惠赠, 用含100 mL/L胎牛血清(购自Hyclone公司)、100 kU/L青霉素和100 mg/L链霉素的RPMI1640培养基, 在37 ℃, 饱和湿度及50 mL/L CO2的细胞培养箱内传代培养. siRNA真核表达载体的构建. 模板DNA合成. 具有互补序列的能够编码短发卡RNA(shRNA)的双链寡核苷酸由武汉晶赛公司合成. 此互补序列为两条反向重复序列, 中间9个插入序列分别为TTCAAGAGA、AAGAACTCT, 两端分别带有BamH I和Hind III的酶切位点(下划线处)便于与Psilencer2.1载体(购自Ambion公司)连接. 其转录产物所形成的siRNA的作用靶点为人survivin mRNA 94-112位核苷酸(GeneBank no.NM-001168), 此片段已利用Blast进行查询, 确定其为特异性序列. 双链寡核苷酸序列5' -GATCCGGACCACCGCATCTCTAC ATTCAAGAGATGTAGAGATGCGGTGGTCCTTTTTTGGAAA-3', 3'-GCCTGGTGGCGTAGAGATGTAAG TTCTCTACATCTCTAC GCCACCAGGAAAAAACCTTTTCGA-5' 阴性对照双链寡核苷酸转录产物所形成的siRNA的作用序列为: 5' -GCTTCATAAGGCGC ATAGC-3' . 此序列不与任何人类基因序列同源. siRNA真核表达载体的构建及鉴定. 用T4DNA连接酶(购自Ambion公司)将双链寡核苷酸与Psilencer2.1线性载体定向连接后转化大肠杆菌DH5, 挑选氨苄青霉素抗性菌落并扩增培养, 然后快速小量制备质粒并行核酸测序鉴定(由上海博亚公司完成).
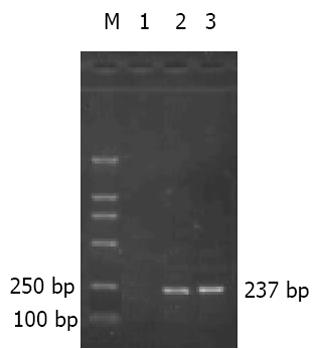
细胞转染及鉴定1×105个HepG2细胞接种于6孔板, 其融合达90%时分别用Psilencer2.1阳性重组子和对照重组子进行转染, 具体操作按脂质体Dosper(购自Roche公司)说明书进行. 转染48 h后按1: 10稀释传代并换用选择培养基(600 mg/L, G418)继续培养14 d, 然后将出现的细胞克隆在培养瓶中扩增培养并传代建系, 分别命名为HepG2/Silence(+)和HepG2/Silence(-)细胞. 阳性细胞克隆neo基因表达的检测. 首先从筛选得到的阳性细胞克隆中提取总RNA, 利用RT-PCR扩增HepG2/Silence(+)和HepG2/Silence(-)细胞中的neo基因片段, 以证实目的基因转染是否成功. 扩增neo基因的引物序列为: 上游引物: 5'-AGAGGCTATTCTGCTATGAC-3'下游引物: 5' -GCTTCAGTGACAACGTCGAG-3'. 扩增片段大小为237 bp.
1.2.1 转染效应检测: (1)survivin基因 mRNA表达检测 以RT-PCR法检测在siRNA干预下survivin基因 mRNA的表达改变. 实验共分三组: HepG2细胞、HepG2/Silence(-)细胞和HepG2/Silence(+)细胞, 每组细胞均设三个复瓶. 首先分别收集各组HepG2细胞1×106个, 然后提取总RNA逆转录成cDNA, 最后进行PCR反应. 引物序列为: 上游引物: 5'-CATGGGTGCCCCGACGTT-3' , 下游引物: 5' -TCAATCCATGGCAGCCAGCT-3' . 扩增片段大小为430 bp. 结果以其和内参照-actin吸光光度值的比值来反映其相对含量. (2)survivin蛋白表达检测 以流式细胞仪检测survivin基因蛋白的表达改变. 分别收集各类HepG2细胞1×106个, 经多聚甲醛固定、PBS洗涤后用0.25%Tritonx-100室温破膜, 然后分别加入小鼠抗人survivin一抗及FITC标记的羊抗小鼠IgG(购自晶美公司). 利用流式细胞仪(BD公司)分析结果, 用无标记和阴性对照样品调节前散射和侧散射, 对不同细胞设置GATE, 计算survivin阳性细胞百分比.
1.2.2 肿瘤细胞的生长分析: 将HepG2细胞、HepG2/Silence(-)细胞和HepG2/Silence(+)细胞分别接种于培养瓶中, 每瓶1×104个细胞, 每种细胞接种三个复瓶, 台盼蓝染色后计数活细胞, 每瓶数3次, 连续测定5 d, 绘制生长曲线.
1.2.3 细胞凋亡观察: 离心收集细胞于1.5 mL离心管内, 采用细胞凋亡-Hoechst染色试剂盒(购自江苏碧云天公司)并按照说明书操作对各组细胞进行处理, 荧光显微镜下观察并拍照. 离心收集细胞于5 mL培养管内, 采用Annexin V 检测试剂盒(购自BD公司)对各组细胞进行凋亡率的检测.
1.2.4 裸鼠异体肿瘤接种实验: 收集处于对数生长期的HepG2、HepG2/silence(-)和HepG2/Silence(+)细胞, 调整细胞密度为4×109/L; 将裸鼠随机分为3组, 每组四只分别接种上述各种细胞株, 每只裸鼠背部皮下注射0.1 mL细胞悬液, 连续观察4 wk, 测量肿瘤长短径, 根据公式V=长径×短径2×0.52来计算肿瘤大小, 绘制肿瘤生长曲线并计算成瘤率.
统计学处理 采用SPSS统计软件对数据进行方差分析, t检验, P<0.05为具有统计学差异.
将重组子Psilencer2.1(+)survivin进行序列分析, 测序结果正确, 表明PSilencer2.1(+)survivin真核表达载体构建成功. 借助阳离子脂质体转染方法, 将重组体PSilencer2.1(+)survivin和PSilencer2.1(-)survivin分别转染至HepG2细胞中, 经G418筛选得到阳性克隆, 再利用重组体中均含有neo标志基因这一特性, 借助RT-PCR的方法检测阳性转染细胞克隆中neo基因的表达, 以检验基因转染是否成功. 电泳结果从两种转染细胞克隆中扩增出的neo基因片段, 在237 bp处可见电泳条带, 其大小与预期结果一致(图1), 证实PSilencer2.1阳性质粒和阴性对照质粒已分别成功转染至HepG2细胞中, 分别称为HepG2/Silence(+)和HepG2/Silence(-)细胞.
转染Psilencer2.1(+)survivin后, HepG2/Silence(+)的PCR产物电泳带较对照组明显减弱, 而不影响-actin的表达, 而转染阴性对照质粒的HepG2细胞无明显变化(图2). 各组的吸光度A值的比值分别为: 1.432±0.012, 1.398±0.017, 0.386±0.013. 经方差分析t检验, HepG2/Silence(+)细胞与HepG2, HepG2/Silence(-)细胞mRNA表达量之间存在差异性(P<0.05).
流式细胞仪检测结果显示, HepG2/Silence(+)细胞survivin蛋白的阳性率为24.0%±0.3%, 对照组HepG2细胞、HepG2/Silence(-)细胞survivin蛋白的阳性率分别为95.4%±1.4%和94.8%±1.8%. 经方差分析t 检验, HepG2/Silence(+)细胞survivin蛋白的阳性率明显低于HepG2细胞和HepG2/Silence(-)细胞(P<0.01, 图3).
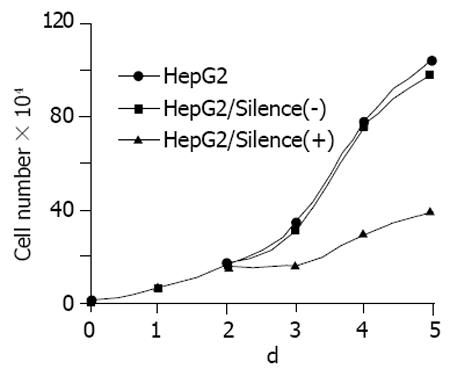
HepG2/Silence(-)细胞的增生和HepG2细胞相比无显著性差异, 而HepG2/Silence(+)细胞的增生从第3 d起明显受抑制, 这种抑制作用随时间延长而增加, 与对照组相比具有统计学差异(P<0.01, 表1, 图4).
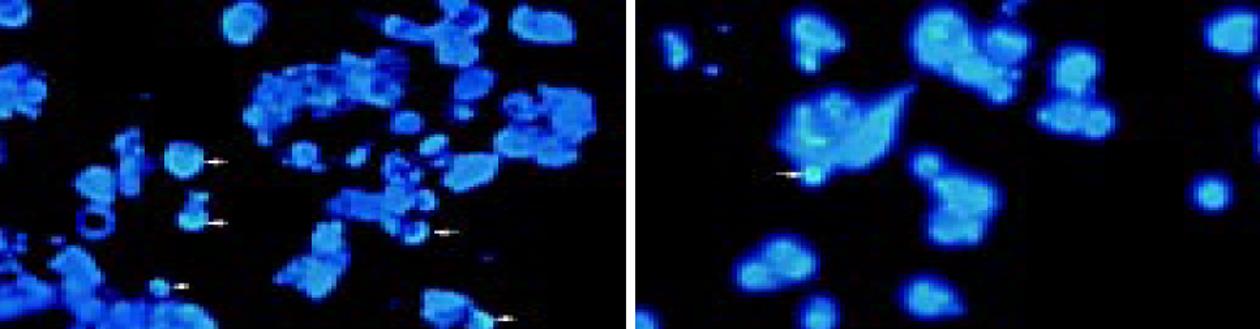
荧光显微镜下观察可见HepG2/Silence(+)细胞体积大小不等, 形态不一, 其中可见4-5个癌细胞体积缩小, 染色质浓缩, 边集(图5A, 箭头所示). 尚可见一癌细胞体积明显缩小, 呈固缩状(典型凋亡小体, 图5B, 箭头所示). 流式细胞仪检测结果表明, 转染survivin SiRNA后, HepG2/Silence(+)细胞凋亡率(7.28%±0.02%)明显增加, 与未转染组(0.48%±0.01%)和转染阴性对照组(0.73%±0.03%)相比, 差异具有显著性(P<0.01, 图6).
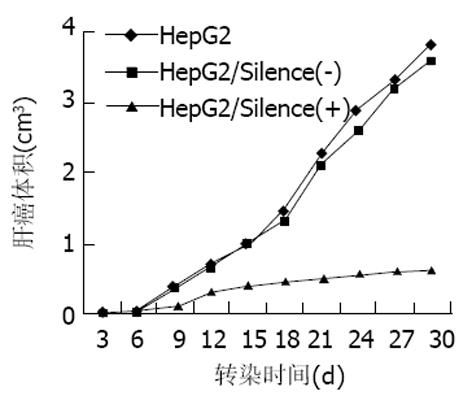
将HepG2、HepG2/silence(-)、HepG2/silence(+)细胞接种于裸鼠背部皮下, 经过5-10 d的成瘤潜伏期后, 在其接种部位形成肉眼可见的瘤块, 其成瘤率分别为100%、100%和25%, HepG2/silence(+)细胞体内成瘤率降低75%, t检验显示HepG2、HepG2/silence(-)、HepG2/silence(+)细胞成瘤率具有极显著差异(P<0.01). 另外持续观察其肿瘤的生长情况并绘制出各组裸小鼠肿瘤的生长曲线(图7). 结果显示: HepG2细胞和HepG2/silence(-)细胞 两组肿瘤的生长趋势较为一致, 而HepG2/silence(+)组肿瘤的生长明显受到抑制.
肿瘤的形成是由多因素造成的, 其中肿瘤细胞中凋亡抑制基因的高表达能明显抑制细胞凋亡, 逃避机体免疫系统对肿瘤细胞的识别和清除[11-12]. 另外凋亡抑制基因还可能在肿瘤细胞产生耐药的过程中发挥重要作用[13-14]. survivin基因具有高度保守性和抑制细胞凋亡的作用, 同时他仅在肿瘤细胞中表达的特性为肿瘤的治疗提供了新的研究方向[15-18]. 目前研究者多采用反义技术进行抑制survivin基因表达的研究, 但由于反义技术在抑制目的基因表达的同时可激活干扰素反应途径而导致非特异性基因mRNA的降解, 因此其应用受到限制[19]. 寻求新的基因干涉手段一直是近年来国内外研究的热点. RNAi是由作用于同源序列mRNA的双链RNA所介导的序列特异性、转录后基因沉默机制[20-24]. 由于执行RNAi 的效应分子siRNA(small interference RNA)在不影响非特异性干扰素途径的情况下可抑制靶基因的表达, 因此RNAi技术不仅成为了除反义技术以外又一种新的分析功能缺失表型的工具, 而且将其作为基因治疗的手段可能具有更广阔的前景[25]. 与传统的反义技术相比较, RNA干扰的作用更为强大[26]. 我们构建了siRNA真核表达载体, 其转录产物可在体内形成由一个茎环所分离的具有反向重复序列的发卡样RNA(shRNA), 此shRNA随后被加工成siRNA而降解目的基因mRNA表达. 这种方法克服了人工合成的siRNA成本高、费用大以及在体内作用时间短的缺点[27]. 通过构建靶向survivin siRNA真核表达载体, 经稳定转染肝癌细胞并进行细胞效应的研究发现, 其在mRNA及蛋白质水平对survivin的抑制率分别达73%和75%而不影响-actin的表达, 而对照组siRNA对的表达无明显影响, 这说明我们构建的siRNA不仅作用强大而且具有特异性.
肿瘤细胞的异常增生是其生物学行为的一项重要表现. 已有研究表明survivin基因通过与cdk4的相互作用释放野生型p21促进肝癌细胞的增生[28-30]. 我们运用RNAi技术抑制survivin基因的表达后观察肝癌细胞的生长情况时发现: 与对照组相比, HepG2/Silence(+)细胞的增值明显受抑制, 而且这种抑制作用随时间延长而增加. 说明survivin基因的表达被有效抑制后可通过诱导肿瘤细胞的凋亡而抑制肿瘤细胞的生长. 我们在运用Hoechst核染色观察肿瘤细胞的凋亡情况时也发现经稳定转染siRNA表达载体的肝癌细胞出现了明显的凋亡现象. 这也从形态上证实了survivin基因的表达抑制可诱导肿瘤细胞凋亡的发生. 在对肝癌细胞进行凋亡率的分析中我们发现经稳定转染siRNA表达载体的肝癌细胞的凋亡率明显高于对照组, 这进一步说明抑制survivin基因的表达可有效诱导肝癌细胞的凋亡. 为了验证survivin siRNA真核表达载体的体内效应, 我们还观察了survivin siRNA真核表达载体转染前后的各组细胞在裸鼠体内成瘤率和肿瘤生长曲线的变化, 发现survivin siRNA真核表达载体转染组肿瘤的生长明显受到抑制, 其成瘤率也明显降低, t检验显示实验组与对照组之间差异具有极显著性. 本实验结果表明, 靶向survivin的siRNA载体不仅可以诱导肝癌细胞的凋亡, 还可以抑制肝癌细胞体内肿瘤的形成, 这为RNAi技术的临床应用提供了有力的实验依据.
总之, 我们构建的siRNA真核表达载体不仅有效抑制了目的基因的表达, 还能在体内体外通过诱导肝癌细胞的凋亡而抑制肿瘤的生长. 这为下一步诱导其对化疗药物的敏感性提供了实验基础及理论.
编辑: N/A
| 1. | He C, Lao WF, Hu XT, Xu XM, Xu J, Fang BL. Anti-liver cancer activity of TNF-related apoptosis-inducing ligand gene and its bystander effects. World J Gastroenterol. 2004;10:654-659. [PubMed] [DOI] |
| 2. | Green DR, Evan GI. A matter of life and death. Cancer Cell. 2002;1:19-30. [PubMed] [DOI] |
| 3. | Fennell DA, Corbo MV, Dean NM, Monia BP, Cotter FE. In vivo suppression of Bcl-XL expression facilitates chemotherapy-induced leukaemia cell death in a SCID/NOD-Hu model. Br J Haematol. 2001;112:706-713. [PubMed] [DOI] |
| 4. | Altieri DC. Survivin, versatile modulation of cell division and apoptosis in cancer. Oncogene. 2003;22:8581-8589. [PubMed] [DOI] |
| 5. | Altieri DC. Survivin and apoptosis control. Adv Cancer Res. 2003;88:31-52. [PubMed] [DOI] |
| 6. | Chang Q, Liu ZR, Wang DY, Kumar M, Chen YB, Qin RY. Survivin expression induced by doxorubicin in cholangiocarcinoma. World J Gastroenterol. 2004;10:415-418. [PubMed] |
| 7. | Wall NR, Shi Y. Small RNA: can RNA interference be exploited for therapy? Lancet. 2003;362:1401-1403. [PubMed] [DOI] |
| 8. | Chi JT, Chang HY, Wang NN, Chang DS, Dunphy N, Brown PO. Genomewide view of gene silencing by small interfering RNAs. Proc Natl Acad Sci U S A. 2003;100:6343-6346. [PubMed] [DOI] |
| 9. | Ait-Si-Ali S, Guasconi V, Harel-Bellan A. [RNA interference and its possible use in cancer therapy]. Bull Cancer. 2004;91:15-18. [PubMed] |
| 10. | Song E, Lee SK, Wang J, Ince N, Ouyang N, Min J, Chen J, Shankar P, Lieberman J. RNA interference targeting Fas protects mice from fulminant hepatitis. Nat Med. 2003;9:347-351. [PubMed] [DOI] |
| 11. | Jäättelä M. Multiple cell death pathways as regulators of tumour initiation and progression. Oncogene. 2004;23:2746-2756. [PubMed] [DOI] |
| 12. | Kirkin V, Joos S, Zörnig M. The role of Bcl-2 family members in tumorigenesis. Biochim Biophys Acta. 2004;1644:229-249. [PubMed] [DOI] |
| 13. | Chandele A, Prasad V, Jagtap JC, Shukla R, Shastry PR. Upregulation of survivin in G2/M cells and inhibition of caspase 9 activity enhances resistance in staurosporine-induced apoptosis. Neoplasia. 2004;6:29-40. [PubMed] [DOI] |
| 14. | Vaziri SA, Grabowski DR, Tabata M, Holmes KA, Sterk J, Takigawa N, Bukowski RM, Ganapathi MK, Ganapathi R. c-IAP1 is overexpressed in HL-60 cells selected for doxorubicin resistance: effects on etoposide-induced apoptosis. Anticancer Res. 2003;23:3657-3661. [PubMed] |
| 15. | Chiou SK, Jones MK, Tarnawski AS. Survivin - an anti-apoptosis protein: its biological roles and implications for cancer and beyond. Med Sci Monit. 2003;9:PI25-PI29. [PubMed] |
| 16. | Zhang W, Chen X, Qiu F. An antisense plasmid targeting survivin expression induces apoptosis and sensitizes hepatocarcinoma cells to chemotherapy. J Huazhong Univ Sci Technolog Med Sci. 2003;23:387-391. [PubMed] [DOI] |
| 17. | Pennati M, Binda M, Colella G, Zoppe' M, Folini M, Vignati S, Valentini A, Citti L, De Cesare M, Pratesi G. Ribozyme-mediated inhibition of survivin expression increases spontaneous and drug-induced apoptosis and decreases the tumorigenic potential of human prostate cancer cells. Oncogene. 2004;23:386-394. [PubMed] [DOI] |
| 18. | Yamamoto T, Manome Y, Miyamoto A, Tanigawa N. [Development of a novel gene therapy using survivin antisense expressing adenoviral vectors]. Gan To Kagaku Ryoho. 2003;30:1805-1808. [PubMed] |
| 19. | Brantl S. Antisense-RNA regulation and RNA interference. Biochim Biophys Acta. 2002;1575:15-25. [PubMed] [DOI] |
| 20. | Agrawal N, Dasaradhi PV, Mohmmed A, Malhotra P, Bhatnagar RK, Mukherjee SK. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 2003;67:657-685. [PubMed] [DOI] |
| 22. | Carette JE, Overmeer RM, Schagen FH, Alemany R, Barski OA, Gerritsen WR, Van Beusechem VW. Conditionally replicating adenoviruses expressing short hairpin RNAs silence the expression of a target gene in cancer cells. Cancer Res. 2004;64:2663-2667. [PubMed] [DOI] |
| 23. | Wang QC, Nie QH, Feng ZH. RNA interference: antiviral weapon and beyond. World J Gastroenterol. 2003;9:1657-1661. [PubMed] [DOI] |
| 24. | Morita T, Yoshida K. [RNAi provides a new tool for functional analyses of the mammalian genes]. Tanpakushitsu Kakusan Koso. 2002;47:1839-1845. [PubMed] |
| 25. | Scherr M, Morgan MA, Eder M. Gene silencing mediated by small interfering RNAs in mammalian cells. Curr Med Chem. 2003;10:245-256. [PubMed] [DOI] |
| 26. | Caplen NJ. RNAi as a gene therapy approach. Expert Opin Biol Ther. 2003;3:575-586. [PubMed] [DOI] |
| 27. | Sui G, Soohoo C, Affar el B, Gay F, Shi Y, Forrester WC, Shi Y. A DNA vector-based RNAi technology to suppress gene expression in mammalian cells. Proc Natl Acad Sci U S A. 2002;99:5515-5520. [PubMed] [DOI] |
| 28. | Ito T, Shiraki K, Sugimoto K, Yamanaka T, Fujikawa K, Ito M, Takase K, Moriyama M, Kawano H, Hayashida M. Survivin promotes cell proliferation in human hepatocellular carcinoma. Hepatology. 2000;31:1080-1085. [PubMed] [DOI] |
| 29. | Suzuki A, Ito T, Kawano H, Hayashida M, Hayasaki Y, Tsutomi Y, Akahane K, Nakano T, Miura M, Shiraki K. Survivin initiates procaspase 3/p21 complex formation as a result of interaction with Cdk4 to resist Fas-mediated cell death. Oncogene. 2000;19:1346-1353. [PubMed] [DOI] |
| 30. | Moon WS, Tarnawski AS. Nuclear translocation of survivin in hepatocellular carcinoma: a key to cancer cell growth? Hum Pathol. 2003;34:1119-1126. [PubMed] [DOI] |