修回日期: 2004-02-25
接受日期: 2004-03-06
在线出版日期: 2004-06-15
目的: 结合本实验室研究结果, 充分利用NCBI的核酸和蛋白数据库中检索到的CagA基因碱基和蛋白质氨基酸序列, 分析其序列特征以及与地域和疾病的相关性.
方法: 利用Vector NTI Suite 9.0, ClastalX(version 1.8), Phylip (version 3.5)和Treeview (version 1.61)软件对检索到的幽门螺杆菌CagA基因碱基和蛋白质氨基酸序列进行多序列比较和系统发育分析.
结果: 检索到可以利用的全序列44条, 部分序列560条. 通过44条全序列比对, 相似性分析和构建系统进化树, 发现CagA基因碱基和蛋白质氨基酸序列可分为具有明显地域特征的东西方两类. 通过44条CagA全序列和266条C端部分序列分析, 发现C端相对多变区的重复序列可分为两类: 第一类为菌株共有的不连续重复序列, 第二类为个别菌株特有的连续重复序列, 此类重复序列包含EPIYA模体的重复. 序列特征与疾病的关系分析表明, 非胃癌株中包含第二类重复序列的菌株占13% (9/71), 胃癌株中包含第二类重复序列的菌株占31% (12/39), χ2检验P = 0.021<0.05, 故可以认为胃癌株中包含第二类重复序列出现率高于非胃癌株, 可以认为包含第二类重复序列的菌株与胃癌有关.
结论: 幽门螺杆菌CagA基因及蛋白序列多态性明显, 具有东西方两个地域聚类特征, 重复序列可分为两类.第二类重复序列中包含EPIYA模体的重复可能导致菌株致病性增强.
引文著录: 周军, 曾浔, 尹焱, 郭欣, 张建中. 幽门螺杆菌CagA基因及蛋白序列多态性分析. 世界华人消化杂志 2004; 12(6): 1307-1312
Revised: February 25, 2004
Accepted: March 6, 2004
Published online: June 15, 2004
AIM: To find the structural characteristics and correlation with region and the clinical outcomes by analyzing nucleotide and amino acid sequences of CagA, our research results and data of CagA sequences deposited in NCBI database.
METHODS: The characteristics of CagA sequences were analyzed, and a phylogenetic tree was constructed by using Vector NTI Suite 9.0, ClastalX (version 1.8), Phylip (version 3.5) and Treeview (version 1.61).
RESULTS: Through searching NCBI nucleotide and protein database, we obtained 44 complete and 560 partial CagA sequences. Aligning these sequences, analyzing similarity and constructing phylogenetic tree, we found CagA seqenceses could be classified into two types: western type and eastern type. Analyzing the CagA C terminal of 44 complete and 266 partial sequences, we classified the repeat sequence of CagA C terminal into two types: type I was discontinuous repeat sequence, common among all the strains, and type II was continuous repeat sequence, found in some individual strains. The type II repeat sequence was found in 31% (12/39) of strains from gastric cancer patients, compared with 13% (9/71) of strains from non-cancer patients (P = 0.021 < 0.05). Strains with the type II repeat sequence were associated with gastric cancer.
CONCLUSION: There is a high diversity of CagA nucleotide and amino acid sequences among H. pylori strains, showing a distinct geographic character as western and eastern region collection. The repeat sequence of CagA C terminal can be classified into two types, and strains with the type II repeat sequence containing EPIYA motif exhibits a stronger pathogenecity.
- Citation: Zhou J, Zeng X, Yin Y, Guo X, Zhang JZ. Sequence diversity analysis of CagA gene and corresponding protein in Helicobacter pylori. Shijie Huaren Xiaohua Zazhi 2004; 12(6): 1307-1312
- URL: https://www.wjgnet.com/1009-3079/full/v12/i6/1307.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i6.1307
幽门螺杆菌(Helicobacter pylori, 简称H. pylori)是慢性胃炎及消化性溃疡的重要致病因子[1-5], 与胃腺癌和MALT淋巴瘤的发生密切相关[6-17], 目前世界范围内成人Hp感染率达50-80%, 但Hp感染人群症状多种多样, 主要是由于感染菌株的毒力、宿主基因型和环境因素不同所致. 细胞毒素相关基因A(the cytotoxin-associated gene A, 简称CagA)编码的细胞毒相关蛋白(the cytotoxin-associated gene A Protein, 简称CagA)作为H. pylori毒力因子之一, 具有强免疫原性, CagA可通过Ⅳ型分泌系统进入胃上皮细胞, 并且发生磷酸化, CagA 蛋白可能与胃炎和胃癌的发生有关[18-20]. 欧美国家的研究表明[21-22], CagA基因存在于约50-70%的H. pylori菌株, 并认为CagA基因阳性菌株比CagA基因阴性菌株可引起更严重的组织炎症和损伤, 因而增加消化性溃疡和胃腺癌形成的危险性.但是中国、日本和韩国的研究发现[23-24], CagA阳性菌株在这些地区的检出率很高, 达90%以上, 并认为CagA不能作为这些地区H. pylori高毒力的标志. 不同菌株的CagA间又存在一些差异, 如其内部存在数个碱基重复序列被认为是产生CagA基因多样性的原因之一, 但其实际差异要复杂的多. 基因序列多态性往往与功能及其致病性相关, 对于CagA全长基因(包括重复序列)多样性的分析, 对深入研究H. pylori致病机制有重要意义. 我们曾对4株幽门螺杆菌, 动物实验用菌株悉尼株(Sydney strain, SS1)、胃癌分离株CAPM N62和CAPM N111, 以及十二指肠溃疡分离株CAPM N93的CagA基因全序列测定, 并已提交GenBank. GenBank中收录了大量序列信息, 本研究的目的是结合我们的测序结果, 充分利用可检索到的全部CagA基因序列和蛋白质氨基酸序列, 试图分析其可能存在的结构特征, 以及是否存在与地域分布和疾病发生的相关性等重要问题.
序列的来源:以"Helicobacter pylori AND CagA[Gene Name]"为关键词, 检索NCBI的核酸和蛋白数据库(http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Nucleotide和http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Protein).
序列的选择标准是重复提交菌株序列经比对后选择其中一条, 排除重复提交和存在提交错误的序列. 利用Vector NTI Suite 9.0软件对GenBank检索到的CagA核苷酸序列和CagA氨基酸序列进行序列比较和相似性分析. 44条全序列用ClastalX(version 1.8)进行序列比对, 并保存为Phylip格式文件, 然后用J. Felsenstein 的Phylip (http://evolution.genetics.washington.edu/phylip.html)程序构建系统进化树, 最后采用Treeview (version 1.61) (http://www.taxonomy.zool-ogy.gla.ac.uk/rod/rod.html)软件对系统进化树进行编辑和比较分析. 560条部分序列通过序列比对后, 分为266条CagA3'端序列和294条CagA 5'端序列, 分别进行序列特征分析.
统计学处理 对含第二类重复序列菌株以及不含第二类重复序列菌株与胃癌的关系进行χ2检验, P<0.05 认为具有统计学意义.
共检索到全序列49条其中J99提交了3次(GenBank接受号AE001439, AE001483, NC_000921).ATCC 43526(GenBank接受号AB015413, AF0-01357, AB003397)其中AF001357和AB003397序列完全一样, 为同一作者重复提交, AB015413为另一作者提交, 序列与另外两条有26个碱基不同, NCTC11637检索到三条序列(GenBank接受号AB015416和AF202973)其中AB015416序列与NCTC11638序列完全相同, 可能是提交时菌株号写错. 排除重复提交和错误序列, 共有44株全序列用于分析(其中4株菌株的CagA基因全序列为本实验室提交). 检索到部分序列560株, 其中3'端序列266株, 5'端序列294株. 核酸序列GenBank接受号: AF043487-AF043490 AJ239683-AJ239737 AJ252963-AJ252986 AF367251 U80065 AJ269852-AJ269897 AF222807-AF222809 AF202219-AF202225 AB090129 U80066 AF198468-AF198486 AF289399-AF289464 AJ315050 AJ313504-AJ313505 AF479032 AB056898-AB057105 AF427098-AF427100 AB089318 AB090090 AB090099 AB090102 AB090106 AB090114 AB090091-AB090098 AB090100-AB090101 AB090103-AB090105 AY176129 AB090107-AB090113 AB090115 AB090117-AB090127 AB090132-AB090154 AY169292-AY169299 AB110957-AB110974 AY162446-AY162453 AB090116 AB090128 AB116733-AB116768 AB003397 AF001357 AF083352 AF202972 AF202973 AF247651 AF249275 AF367250 AB015404-AB015416 AB017921-AB017923 AY121840 AB090140 AB090076-AB090078 AB090082-AB090084 AB090086 AB090073-AB090075 AB090079-AB090081 AB090085 AB090087-AB090089 AB090130 AB090131.
蛋白序列GenBank接受号: AAC02082-AAC02085 AAD06073 AAD27691 CAB37794-CAB37829 CAB55439-CAB55462 CAB56425-CAB56446 CAB56485 CAB56487-CAB56489 CAB56495 CAB56447-CAB56449 CAB56496 CAB56450 CAB56490 CAB56497 CAB56451 CAB56486 CAB56494 CAB56493 CAB56452-CAB56456 AAF17598 AAF63759 AAF35340-AAF35342 AAF42821-AAF42827 AAF43608-AAF43626 AAF82477 AAC44706 AAG09848-AAG09913 AAK54719 AAK54720 BAB20914-BAB20929 BAB87265-BAB87369 BAB87370-BAB87472 BAC10431 Q9ZLT1 AAL28091-AAL28093 AAM88613 BAC07548 BAC10433-BAC10435 BAC10437-BAC10444 BAC10446 BAC10447 BAC10449-BAC10451 BAC10453-BAC10459 BAC10461 P55746 BAC10463-BAC10473 BAC10475 BAC10478-BAC10485 BAC10487-BAC10500 P80200 AAO11686-AAO11693 BAC77000-BAC77017 AAN86014-AAN86021 AAF17597 P55980 BAC81787-BAC81822 BAA19853 AAB58747 AAB81560 AAB81561 AAC32880 CAB56492 CAB56491 CAB56457 AAL86902 CAC42220 CAC44458 CAC44467 BAC10432 BAC10436 BAC10445 BAC10448 BAC10452 BAC10460 BAC10462 BAC10474 BAC10476 BAC10477 BAC10486 BAC10425 BAC10422-BAC10424 BAC10428-BAC10430 BAC10419-BAC10421 AAO39035 BAC10427 NP_223213.
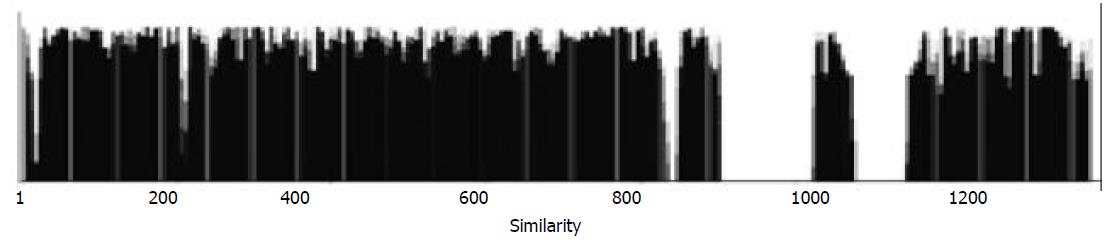
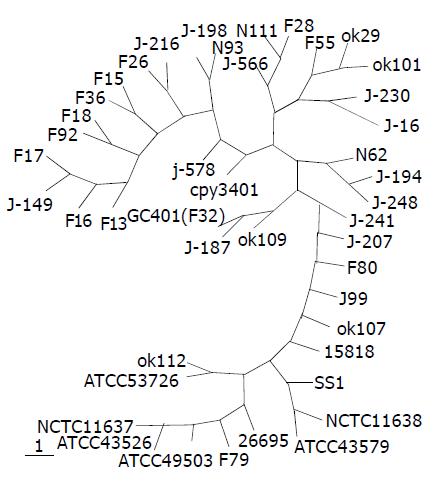
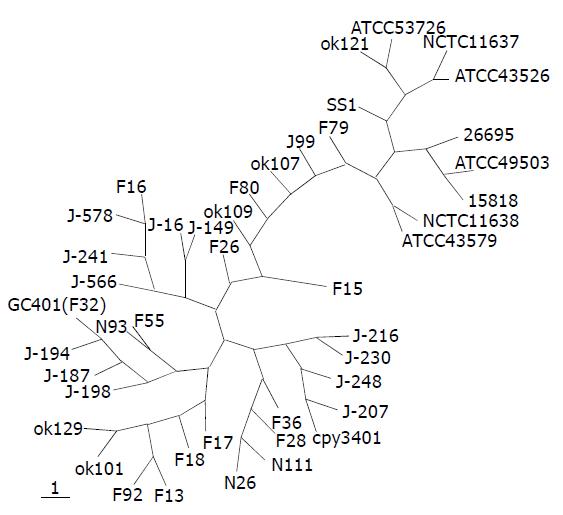
从CagA44条全长序列、294条5'端序列和266条3'端部分序列比对结果(因图形太大未附)来看, 序列内存在大量的碱基的插入、缺失、替换和重复. 5'端序列相对保守, 差别是由于替换、缺失和小的插入造成. 3'端序列由于个别菌株存在连续的大片段重复序列, 序列差异更加明显. 44株CagA全长序列碱基范围在3 444-3 828 bp之间, 最长与最短之间相差376 bp. 相应氨基酸残基范围在1 147-1 275 aa, 最长与最短之间相差128 aa, 分子质量范围在128-142 ku之间. 3'端多变区位于2 610-3 090 bp之间; 从蛋白质氨基酸序列分析来看, 多变区位于870-1 030 aa之间. 从长度上来分大多数氨基酸序列在1 147-1 186 aa之间, 其差别是由于替换、缺失和小的插入造成, 大于1 186 aa是由于个别菌株内部连续的重复序列造成的. 从44株CagA全长序列推测的氨基酸序列的相似性比较结果 (图1), 可见, 序列相似性比值大多数部位在0.5-1.0之间, 是由于序列之间存在大量的碱基的插入, 缺失和替换. 有五个区间相似形小于0.2. 序列比对结果可见, 前三个是由个别菌株短序列插入或缺失造成的, 后两个是由个别菌株的重复序列造成的. 依据44株CagA全长核苷酸序列和推定氨基酸序列比对结果, 绘制出的上述44株菌CagA基因及CagA蛋白质的系统进化树(图2, 3). 可见, 无论从CagA基因核苷酸序列, 还是推定CagA氨基酸序列, 东方菌株包括中国、韩国、日本、香港台湾菌株聚类关系较近, 而西方菌株包括澳大利亚、英国、美国等聚类关系较近. 从序列的相似性比较及进化分析来看, 菌株可分为东西方两类, 其中西方菌株中包含3株日本菌株(F79, F80和OK107), 但其序列与西方菌株非常相似, 菌株F80含有部分东方菌株序列替换.
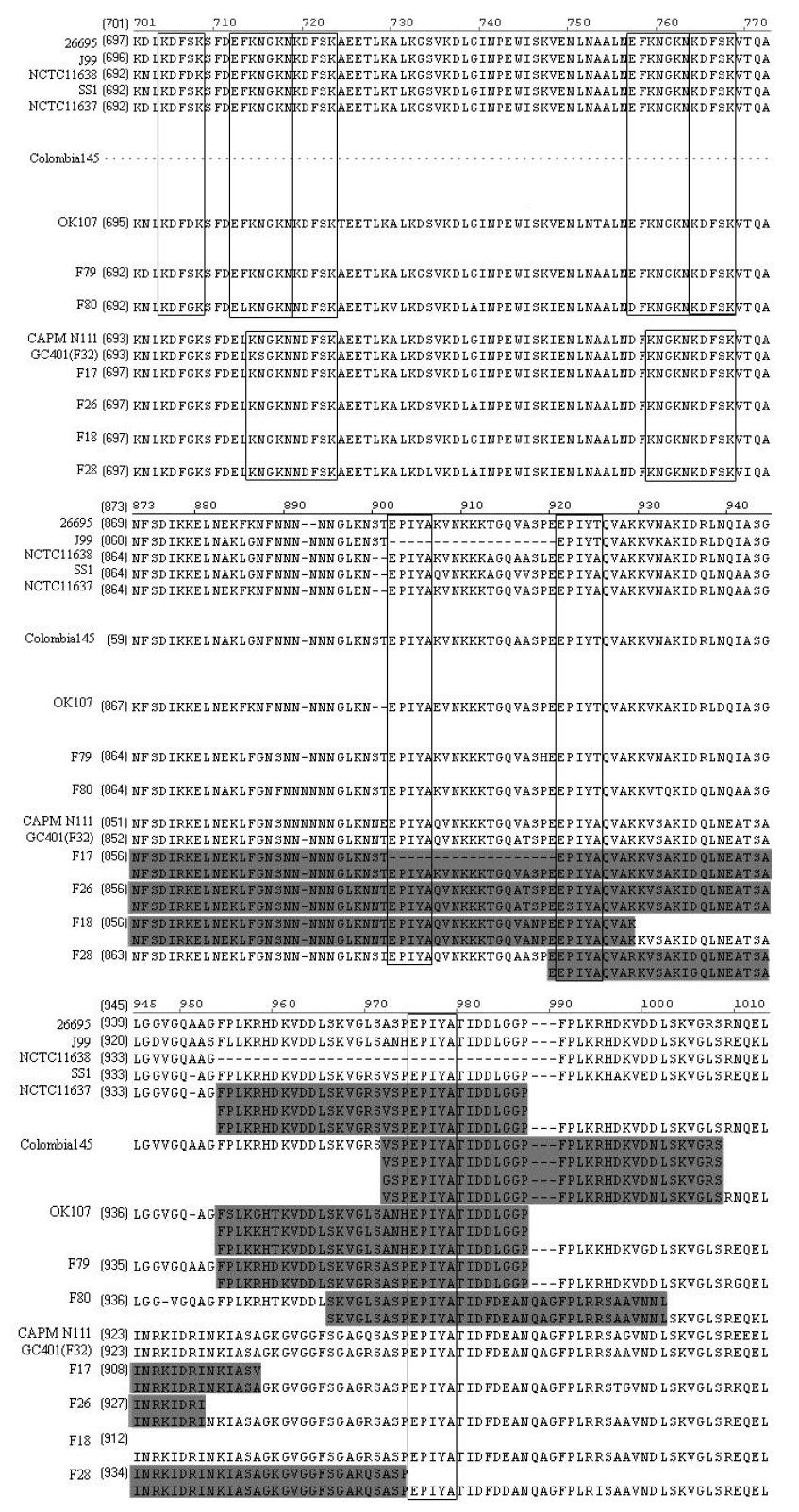
根据44条CagA全长序列和266条部分序列C端多变区重复序列代表菌株比对结果(图4), 重复序列可分两类: 第1类为菌株间共有不连续的重复序列. 所有菌株共有重复序列EPIYA重复3次, 西方菌株共有的重复序列EFKNGKNKDFSK重复2次, 发现了一个新重复序列KDFSK重复3次, 其中两个拷贝在重复序列EFKNGKNKDFS内部, FPLKRHDKVDDLSKVR/LS重复两次, 东方菌株共有的重复序列KNGKNKDFSK重复两次. 第2类为个别菌株特有的连续的重复序列, 这种重复区起使位点和长度不一, 但都位于EPIYA重复序列附近, 重复序列内大部分都包含EPIYA序列, 东方菌株重复区多在前两个EPIYA序列附近, 并且包含前两个EPIYA序列中的1个或2个. 西方菌株重复序列多在第3个EPIYA序列附近, 并且包含第3个EPIYA序列. 个别菌株重复序列包含多个EPIYA序列(最多可达6个). 在44条CagA全长序列和266条部分序列中已知疾病背景菌株共110株, 其中胃癌株39株, 非胃癌株71株, 序列特征与疾病的关系见(表1), 非胃癌株中包含第2类重复序列的菌株占13% (9/71), 胃癌株中包含第2类重复序列的菌株占31% (12/39), χ2检验χ2 = 5.335, P = 0.021<0.05, 故可以认为胃癌株中包含第2类重复序列出现率高于非胃癌株, OR = 3.062, 95%CI (1.180-7.947), 即与非胃癌株相比, 感染含第2类重复序列的菌株的人患胃癌的危险性高于不包含第2类重复序列的菌株的人, 可以认为包含第2类重复序列的菌株与胃癌有关.
| 序列特征 | 疾病分类 | 合计 (n = 100) | |
| 非胃癌株(n = 71) | 胃癌株(n = 39) | ||
| 含第2类重复序列 | 9 (13) | 12 (31) | 21 (19) |
| 不含第2类重复序列 | 62 (87) | 27 (69) | 89 (81) |
现在, 许多公用序列数据库收录了大量世界各地的序列信息, 为进行不同地区和疾病背景菌株序列分析提供了便利的条件. 我们检索到H. pylori CagA全序列49条, 部分序列560条, 但序列中存在一定的错误率, 有许多序列疾病背景不清楚, 在今后利用和提交序列时应注意. 通过分析44株CagA全长序列和560株部分序列发现幽门螺杆菌CagA基因及蛋白序列多态性明显, 序列之间存在大量的碱基插入、缺失、替换和重复, 尤其是个别菌株3'端多变区存在大片段连续重复序列, 这种个别菌株内部的重复序列是导致不同菌株CagA分子质量差异的主要原因.结果表明CagA序列间存在高度的多态性, 远比以往认识的差别要复杂的多.
有研究发现H. pylori CagA序列有明显的地域聚类关系[25-27], 我们通过分析和比较多个地区的H. pylori CagA序列特征和系统进化关系, 发现H. pylori CagA序列可明显的分为东西方两类, 有3株日本菌株序列与西方菌株一致, 并且有一株序列包含一小段东方菌株序列的插入. Yamaoka et al[28]也发现了一株韩国菌株有一小段西方菌株序列插入. 可能是由于随着人类交往的频繁, 存在着东西方菌株的交叉和混合感染, 并且发生菌株间序列交换.
CagA蛋白的多态性主要由CagA的C端多变区的重复序列造成, Covacci et al[29]通过CCUG17874和G39菌株测序分析比较, 发现CCUG17874包含EFKNGKN-KDFSK和EPIYA重复序列, G39包含2个拷贝的102 bp重复序列, 相应氨基酸序列为D1(PEPIYA)、D2(DDL)、D3(FPLKRHDKVDDLSKV). Dong et al[30]测序分析12株爱尔兰菌株, 提出了一个新的重复序列b(EPIYTQVAKKVNAKIDRLNQASGLLGGVGQAAG). Evans et al[31]测序分析了13株菌包括CagA3'端可变区在内的1059NT核苷酸序列, 发现除102 bp重复序列外, 还含有12-99 bp不等的重复序列, 相应氨基酸序列为D1a(EPIYA),D1b(PEEPIYA),D2(TIDDLGG), D3(FPLKRHDKVDDLSKV), D4(GRSVSP), D5(KIDQ/RLN-Q or KIDNLN/SQ), D6(D6 = D3-D4-D1a-D2). Yamaoka et al[28]分析7株日本菌株和4株西方菌株提出了3个重复序列R1(EPIYA), R2(QVNKKKTGQ-ATSPE), R3(QVARKVSAKIDQLNEATSAINRKIDRINKIASAGKGVGGFSGAGRSANP). 我们的分析结果表明重复序列可分为两类, 这两类重复序列是截然不同的, 第2类重复序列(个别菌株特有的连续重复序列)是导致不同菌株CagA序列多态性的主要原因, 但其起始位点和长度不一, 无法用共有序列来概括. 以前的分析因其选用的菌株少, 没有分清两类菌株和两类重复序列的差异, 虽然提出了很多重复序列, 但不能很好的描述重复序列的特点. 我们根据序列特点对菌株和重复序列进行了分类, 更好的描述了重复序列的特点. 对于理解CagA序列多态性特点和序列重复的机制有很大帮助. 第2类重复序列与疾病的关系分析发现, 胃癌株中包含第2类重复序列出现率高于非胃癌株, 统计分析表明含第2类重复序列的菌株与胃癌有关.而在第2类重复中都包含EPIYA模体的重复, 许多研究通过质谱和突变证实CagA的磷酸化位点都是EPIYA模体内的酪氨酸[32-34], 是否第2类重复序列包含更多酪氨酸磷酸化模体, 导致菌株致病性增强, 还有待于实验证明.
H. pylori菌株间的多态性表明, H. pylori是目前多态性最高的细菌菌株, 充分认识H. pylori多态性与地域和疾病相关性, 有助于了解菌株与宿主的相互作用, 以及何种菌株具有致病性, 何种菌株导致胃癌等严重胃肠道疾病, 对于H. pylori感染的防治有重要意义. 我们分析了44株CagA全长序列和560株部分序列特征以及多变区重复序列特征与疾病的关系, 根据序列特点对菌株和重复序列进行了分类, 发现第2类重复序列中包含EPIYA模体的重复可能导致菌株致病性增强, 但是, CagA蛋白的磷酸化以及功能的实现, 除了蛋白质的结构以外, 还有赖于CagA蛋白质的表达和完整的IV型分泌系统对CagA蛋白的转运. Mimurod et al[35]最近研究发现H. pylori感染引起的细胞蜂鸟样改变并不需要CagA蛋白的磷酸化. 另外, H. pylori感染是胃癌发生多因素、多步骤过程中的一个因素, 影响其中一个步骤或多个步骤, 还有其他许多因素影响H. pylori感染后致病的最终结果. 尽管CagA蛋白的多态性与疾病的关系还有待于进一步的明确, 但可以肯定的说, 利用国际范围内的共享资源进行全面分析, 将越来越受到重视, 其分析结果对H. pylori及其相关疾病的研究也将会起到重要推动作用.
| 1. | Hobsley M, Tovey FI. Helicobacter pylori: the primary cause of duodenal ulceration or a secondary infection? World J Gastroenterol. 2001;7:149-151. [PubMed] [DOI] |
| 5. | Vandenplas Y. Helicobacter pylori infection. World J Gastroenterol. 2000;6:20-31. [PubMed] [DOI] |
| 15. | Gao HJ, Yu LZ, Bai JF, Peng YS, Sun G, Zhao HL, Miu K, L XZ, Zhang XY, Zhao ZQ. Multiple genetic alterations and behavior of cellular biology in gastric cancer and other gastric mucosal lesions:H.pylori infection, histological types and staging. World J Gastroenterol. 2000;6:848-854. [PubMed] [DOI] |
| 18. | Odenbreit S, Püls J, Sedlmaier B, Gerland E, Fischer W, Haas R. Translocation of Helicobacter pylori CagA into gastric epithelial cells by type IV secretion. Science. 2000;287:1497-1500. [PubMed] [DOI] |
| 19. | Asahi M, Azuma T, Ito S, Ito Y, Suto H, Nagai Y, Tsubokawa M, Tohyama Y, Maeda S, Omata M. Helicobacter pylori CagA protein can be tyrosine phosphorylated in gastric epithelial cells. J Exp Med. 2000;191:593-602. [PubMed] [DOI] |
| 20. | Higashi H, Tsutsumi R, Muto S, Sugiyama T, Azuma T, Asaka M, Hatakeyama M. SHP-2 tyrosine phosphatase as an intracellular target of Helicobacter pylori CagA protein. Science. 2002;295:683-686. [PubMed] |
| 21. | Nomura A, Stemmermann GN, Chyou PH, Perez-Perez GI, Blaser MJ. Helicobacter pylori infection and the risk for duodenal and gastric ulceration. Ann Intern Med. 1994;120:977-981. [PubMed] [DOI] |
| 22. | Nomura A, Stemmermann GN, Chyou PH, Kato I, Perez-Perez GI, Blaser MJ. Helicobacter pylori infection and gastric carcinoma among Japanese Americans in Hawaii. N Engl J Med. 1991;325:1132-1136. [PubMed] [DOI] |
| 23. | Pan ZJ, van der Hulst RW, Feller M, Xiao SD, Tytgat GN, Dankert J, van der Ende A. Equally high prevalences of infection with cagA-positive Helicobacter pylori in Chinese patients with peptic ulcer disease and those with chronic gastritis-associated dyspepsia. J Clin Microbiol. 1997;35:1344-1347. [PubMed] |
| 24. | Shimoyama T, Fukuda S, Tanaka M, Mikami T, Saito Y, Munakata A. High prevalence of the CagA-positive Helicobacter pylori strains in Japanese asymptomatic patients and gastric cancer patients. Scand J Gastroenterol. 1997;32:465-468. [PubMed] [DOI] |
| 25. | Rahman M, Mukhopadhyay AK, Nahar S, Datta S, Ahmad MM, Sarker S, Masud IM, Engstrand L, Albert MJ, Nair GB. DNA-level characterization of Helicobacter pylori strains from patients with overt disease and with benign infections in Bangladesh. J Clin Microbiol. 2003;41:2008-2014. [PubMed] [DOI] |
| 26. | van der Ende A, Pan ZJ, Bart A, van der Hulst RW, Feller M, Xiao SD, Tytgat GN, Dankert J. cagA-positive Helicobacter pylori populations in China and The Netherlands are distinct. Infect Immun. 1998;66:1822-1826. [PubMed] |
| 27. | Hoshino FB, Katayama K, Watanabe K, Takahashi S, Uchimura H, Ando T. Heterogeneity found in the cagA gene of Helicobacter pylori from Japanese and non-Japanese isolates. J Gastroenterol. 2000;35:890-897. [PubMed] [DOI] |
| 28. | Yamaoka Y, El-Zimaity HM, Gutierrez O, Figura N, Kim JG, Kodama T, Kashima K, Graham DY. Relationship between the cagA 3' repeat region of Helicobacter pylori, gastric histology, and susceptibility to low pH. Gastroenterology. 1999;117:342-349. [PubMed] [DOI] |
| 29. | Covacci A, Censini S, Bugnoli M, Petracca R, Burroni D, Macchia G, Massone A, Papini E, Xiang Z, Figura N. Molecular characterization of the 128-kDa immunodominant antigen of Helicobacter pylori associated with cytotoxicity and duodenal ulcer. Proc Natl Acad Sci USA. 1993;90:5791-5795. [PubMed] [DOI] |
| 30. | Dong Q, O'Sullivan M, Hall W, Herra C, Kean C, O'Morain C, Buckley M. Identification of a new segment involved in cagA 3' region variation of Helicobacter pylori. FEMS Immunol Med Microbiol. 2002;33:51-55. [PubMed] [DOI] |
| 31. | Evans DJ Jr, Queiroz DM, Mendes EN, Evans DG. Diversity in the variable region of Helicobacter pylori cagA gene involves more than simple repetition of a 102-nucleotide sequence. Biochem Biophys Res Commun. 1998;245:780-784. [PubMed] [DOI] |
| 32. | Stein M, Bagnoli F, Halenbeck R, Rappuoli R, Fantl WJ, Covacci A. c-Src/Lyn kinases activate Helicobacter pylori CagA through tyrosine phosphorylation of the EPIYA motifs. Mol Microbiol. 2002;43:971-980. [PubMed] [DOI] |
| 33. | Asahi M, Tanaka Y, Izumi T, Ito Y, Naiki H, Kersulyte D, Tsujikawa K, Saito M, Sada K, Yanagi S. Helicobacter pylori CagA containing ITAM-like sequences localized to lipid rafts negatively regulates VacA-induced signaling in vivo. Helicobacter. 2003;8:1-14. [PubMed] [DOI] |
| 34. | Backert S, Moese S, Selbach M, Brinkmann V, Meyer TF. Phosphorylation of tyrosine 972 of the Helicobacter pylori CagA protein is essential for induction of a scattering phenotype in gastric epithelial cells. Mol Microbiol. 2001;42:631-644. [PubMed] [DOI] |