修回日期: 2003-07-25
接受日期: 2003-11-13
在线出版日期: 2004-03-15
目的: 报告SEN病毒核苷酸的异质性和准种特点.
方法: 取深圳地区SEN病毒流行病学调查得到的SENV D型或H型阳性血清各3份进行巢式PCR扩增, 阳性PCR产物克隆至T载体, 每份随机挑选1-11份阳性克隆进行分别测序, 并与GeneBank上的北京株、美国株、意大利株和日本株进行比较分析.
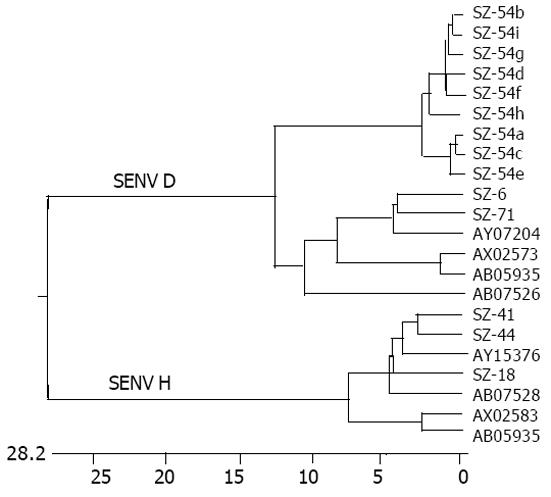
结果: 挑选的16株克隆中D型和H型分别为13株和3株, 均属于SENV序列. 来自SZ-54的11株SENV D型克隆中准种占81.8%(9/11). 本地3例患者的11株SENV D型阳性克隆与GeneBank的4株异地株, 共15株间的同源性为77.7%-99.5%. 同一宿主9个克隆间的碱基变异个数为8.5±5.2, 与同一地区不同宿主的11个克隆间的28.9±3.2和不同地区不同宿主的15个克隆间的29.6±7.8比较, 有显著性差异(P均<0.001). 3株SENV H型阳性克隆与GeneBank的4株异地株共7株间的同源性为74.6%-95.0%. 进化分析表明, 来自同一宿主的分离株遗传关系最近, 同地区不同宿主的分离株其次, 与4株异地分离株的遗传距离较远.
结论: SENV核酸的变异性高, 个体间存在较大差异, 且在同一个感染者体内也存在大量SENV准种. 在检测SENV的方法选择、比较SENV的序列差异和分析SENV的临床意义时应充分考虑到SENV的这种特性.
引文著录: 唐蔚, 彭晓谋, 杨山麦, 王召钦, 王辉, 蒋晓玲, 周伯平. SEN病毒核酸异质性及准种现象的观察. 世界华人消化杂志 2004; 12(3): 604-606
Revised: July 25, 2003
Accepted: November 13, 2003
Published online: March 15, 2004
AIM: To describe the gene heterogeneity and quasispecies of SEN virus.
METHODS: Three SEN virus subtype D or H positive sera from a SEN virus prevalence investigation in Shenzhen District were randomly selected and a nested-PCR were performed. 3 positive PCR products of each subtype were ligated into pMD18 T-vectors. 1-11 clones from every PCR product were randomly selected to be sequenced and aligned with SENV sequences retrieved from GeneBank representing strains from 4 different countries and district. Homology of nucleotide sequences were analysed and a phylogenic tree was conducted.
RESULTS: 13 clones of SENV D and 3 of SENV H were sequenced and demonstrated to belong to SENV. 81.8% (9/11) clones from a single host SZ-54 were quasispecies. The nucleotide sequence homology of 15 SENV D clones (11 of Shenzhen and 4 retrieved from GeneBank representing 4 other countries and district) were 77.7%-99.5%. The amount of mutated bases between each two of 9 clones from same host were 8.5±5.2, which was significantly different from that between each two of 11 clones from 3 hosts within Shenzhen district (28.9±3.2) and that between each two of 15 clones from different hosts and districts (29.6±7.8) (P < 0.001). The nucleotide sequence homology of 7 SENV H clones (3 of Shenzhen and 4 retrieved from GeneBank as described above) were 74.6%-95.0%. The phylogenic tree showed that clones from same patient were more related to each other than those from other patients and much closer than strains representing other countries and district.
CONCLUSION: There exist SENV quasispecies in SENV carriers. The heterogeneity of nucleic acid of SENV may be influnced by differences between hosts and regions. This unique characteristic should be taken in consideration when detecting the virus, determining sequence mutation and even discussing its clinical implication.
- Citation: Tang W, Peng XM, Yang SM, Wang ZQ, Wang H, Jiang XL, Zhou BP. Demonstration of gene heterogeneity and quasispecies of SEN virus. Shijie Huaren Xiaohua Zazhi 2004; 12(3): 604-606
- URL: https://www.wjgnet.com/1009-3079/full/v12/i3/604.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i3.604
自1997年日本学者Nishizawa et al[1]首先从未知病原的输血后肝炎患者血清中分离得到一种被称为TTV的病毒以来, 世界各地的学者做了大量的研究. 这是一组单链DNA病毒, 陆续报道有新的型别和种群如SANBAN病毒[2]、YONBAN病毒[3]、SEN病毒(SEN virus, SENV)[4-5]、PM病毒(PM virus, PMV)[6]等被分离. SENV有8个亚型[7], 且感染率有显著的地区差异[8], 本课题组对深圳地区进行SENV的流行病学调查[9]时, 还显示该病毒核酸存在显著的个体差异. 为此, 本研究进一步探讨了SENV感染者体内的准种情况和意义.
检测SENV D型或H型阳性的患者各3例均从流行情况调查[9]中随机选出. SENV D 型阳性的SZ-6、SZ-71和SZ-54均为慢性乙肝患者, 男2例, 女1例, 年龄23-47岁. SENV H型阳性的3例中SZ-18为慢性丙肝患者, SZ-41为慢性乙肝患者, SZ-44为非甲-戊型肝炎患者, 均为男性, 30-41岁. 诊断参照《病毒性肝炎防治方案》[10].
按照文献报道的步骤, 采用巢式PCR方法对6份血清进行PCR扩增, 产物经纯化后克隆至pMD-18 T 载体中, 编号为SZ-54的标本挑选11个克隆外, 其余5份分别挑选1个克隆, 共16株, 完成阳性克隆鉴定、抽提纯化和测序, 获得DNA序列. 用Lasergene99和MegAlign软件先将SZ-54标本的11株克隆进行序列比对, 剔除相同的序列, 计算准种株的比例. 将剩余的SENV D型深圳株的克隆序列与GeneBank上公布的北京株、美国株、意大利株和日本株(Accession Number见表1)一起排序, 并列表进行同源性比较. 分别计算所有核苷酸序列之间碱基变异的个数, 列表并将数据分为同宿主组、同地区不同宿主组和不同地区不同宿主组, 组间数据用方差分析作统计学处理. 将3株SENV H型深圳株克隆序列与GeneBank上公布的北京株、美国株、意大利株和日本株(Accession Number见表1)一起排序, 并列图表进行同源性比较. 用MegAlign软件将所有SENV克隆序列14株, 连同GeneBank公布各国家地区SENV D型和H型代表株8株一起进行种系进化分析, 并作进化树图.
| SENV D | SENV H | |
| 北京株 | AY072045 | AY153769 |
| 美国株 | AB075263 | AB075283 |
| 意大利株 | AX025730 | AX025838 |
| 日本株 | AB059352 | AB059353 |
SENV D型11株克隆与GeneBank来自北京、美国、意大利和日本的序列的排序图、同源性分析表和碱基变异个数统计表分别见图1, 表2. SZ-54的11株克隆中, 有3株序列相同, 准种株9株占81.8%(9/11). 这9株克隆间的核苷酸同源性介于91.4-99.5%, SZ-6、SZ-54和SZ-71的11株克隆间的同源性为82.7-91.4%, 与北京、美国、意大利和日本的4株克隆间的同源性介于77.7-90.4%. 突变无明显热点.
| AY153769 | AB075283 | AX025838 | AB059353 | SZ-18 | SZ-41 | SZ-44 | |
| AY153769 | *** | 93.9 | 84.0 | 80.7 | 89.5 | 95.0 | 90. 6 |
| AB075283 | *** | 89.0 | 85.6 | 85.6 | 90.1 | 84.5 | |
| AX025383 | *** | 95.0 | 78.5 | 80.1 | 78.5 | ||
| AB059353 | *** | 74.6 | 77.3 | 77.3 | |||
| SZ-18 | *** | 95.0 | 89.5 | ||||
| SZ-41 | *** | 95.0 | |||||
| SZ-44 | *** |
如表2所示, 将克隆序列的碱基变异个数按宿主和地区再进行分组:黄色区域代表同宿主组, 绿色区域为同地区不同宿主组, 兰色区域为不同地区不同宿主组, 各组碱基变异个数的均值(个)分别为8.5±5.2、28.9±3.2和29.6±7.8. 经方差分析, 同宿主组的碱基变异个数均数与同地区不同宿主组和不同地区不同宿主组相比, P值<0.001, 后两组相比, P>0.05. 表明SENV D型序列间有明显的个体差异.
SENV H型3株克隆与GeneBank来自北京、美国、意大利和日本的序列的排序图和同源性分析表. 深圳地区3个感染者的3株克隆间的同源性为89.5-95.0%, 来自深圳及北京、美国、意大利和日本的7株克隆间的同源性为74.6-90.6%. 突变无明显热点.
所有16株克隆序列与GeneBank来自北京、美国、意大利和日本的序列的进化分析树. 进化树分析进一步证实16株克隆分属SENV D型和H型, 同一宿主的分离株遗传关系最近, 同一地区不同宿主的分离株其次, 与4株异地分离株的遗传距离最远.
DNA病毒的碱基突变率与基因组长度成反比, 环状病毒是最小的DNA病毒, 所以SENV碱基突变的速度也快. 日本学者Takeji比较了2例慢性SENV-D感染患者间隔12 a的系列血清SENV核酸序列[11], 发现(1)在SENV的高突变区域(HVRs)的同义替代率远小于非同义替代率(dS/dN<1), 反映了出于免疫压力, 持续感染的变异株被选择出来, 以逃避宿主的免疫监控, 形成持续感染. (2) SENV D ORF1和ORF2的同义替代率是7.32×10-4/位点/年, 与RNA病毒相似, 比DNA病毒高, 表明SENV在复制时基本无或仅有很低的校对功能。以上研究合理地解释了SENV序列存在高度变异性的原因. 本研究显示, 同一感染者体内检测到不同的SENV序列属准种群, 与HCV[12-13]和HIV[14]等易形成准种的情况高度类似, 但准种的数量更多, 变异更大.SENV D型和H型的核苷酸序列存在明显的个体差异. 本研究虽然只运用了普通的Taq DNA聚合酶, 但其错配率为103-4, 而目的片段长度仅约200 bp, 因此可排除由于PCR扩增的错配而导致的突变.
SENV基因组核苷酸序列的高异质性特点, 是环状单链DNA病毒的共同特性[5]. Takeji估计, SENV是通过宿主的DNA聚合酶系统进行无校正复制的, 因为在SENV基因组中缺少编码聚合酶的基因. 那么, SENV的基因组可能是由功能相对不重要的基因如假基因等组成的, 这些基因比功能相关基因进化得快[15]. 对SENV核苷酸异质性和准种现象的观察的意义在于我们选择检测SENV的方法、比较SENV的序列差异和分析SENV的临床意义时应充分考虑到这种特性. 如选择检测方法时, 选择检测方时, 既要检测SENV总感染率, 也要对其进行型、株和准种的鉴定. 虽然从流行病学上分析, SENV可能不具备致病性, 但目前尚不能排除个别型或株, 甚至部分准种存在致病性的可能性.
编辑: N/A
| 1. | Nishizawa T, Okamoto H, Konishi K, Yoshizawa H, Miyakawa Y, Mayumi M. A novel DNA virus (TTV) associated with elevated transaminase levels in posttransfusion hepatitis of unknown etiology. Biochem Biophys Res Commun. 1997;241:92-97. [PubMed] [DOI] |
| 2. | Hijikata M, Takahashi K, Mishiro S. Complete circular DNA genome of a TT virus variant (isolate name SANBAN) and 44 partial ORF2 sequences implicating a great degree of diversity beyond genotypes. Virology. 1999;260:17-22. [PubMed] [DOI] |
| 3. | Takahashi K, Hijikata M, Samokhvalov EI, Mishiro S. Full or near full length nucleotide sequences of TT virus variants (Types SANBAN and YONBAN) and the TT virus-like mini virus. Intervirology. 2000;43:119-123. [PubMed] [DOI] |
| 4. | Primi D, Fiordalisi G. International patent number WO0028039. Avaliable from: http://ep.espacenet.com/. |
| 5. | Tanaka Y, Primi D, Wang RY, Umemura T, Yeo AE, Mizokami M, Alter HJ, Shih JW. Genomic and molecular evolutionary analysis of a newly identified infectious agent (SEN virus) and its relationship to the TT virus family. J Infect Dis. 2001;183:359-367. [PubMed] [DOI] |
| 6. | Hallett RL, Clewley JP, Bobet F, McKiernan PJ, Teo CG. Characterization of a highly divergent TT virus genome. J Gen Virol. 2000;81:2273-2279. [PubMed] [DOI] |
| 7. | Sottini A, Mattioli S, Fiordalisi G, Mantero G, Imberti L, Moratto D, Primi D. Molecular and biological characterization of SEN virusese: a family of viruses remotely related to the original TTV isolation. Proceedings of the 10th International Symposium on Viral Hepatitis and Liver Disease. H. Margolis (Ed), Meditech Media, Atlanta. 2001;. |
| 8. | Kao JH, Chen W, Chen PJ, Lai MY, Chen DS. Prevalence and implication of a newly identified infectious agent (SEN virus) in Taiwan. J Infect Dis. 2002;185:389-392. [PubMed] [DOI] |
| 9. | 唐 蔚, 彭 晓谋, 张 瑛, 王 辉, 蒋 晓玲, 周 伯平. 聚合酶链反应检测SEN病毒D型和H型方法的建立及初步应用. 世界华人消化杂志. 2003;11:1540-1543. [DOI] |
| 11. | Umemura T, Tanaka Y, Kiyosawa K, Alter HJ, Shih JW. Observation of positive selection within hypervariable regions of a newly identified DNA virus (SEN virus)(1). FEBS Lett. 2002;510:171-174. [PubMed] [DOI] |
| 12. | McAllister J, Casino C, Davidson F, Power J, Lawlor E, Yap PL, Simmonds P, Smith DB. Long-term evolution of the hypervariable region of hepatitis C virus in a common-source-infected cohort. J Virol. 1998;72:4893-4905. [PubMed] |
| 13. | Smith DB, Simmonds P. Characteristics of nucleotide substitution in the hepatitis C virus genome: constraints on sequence change in coding regions at both ends of the genome. J Mol Evol. 1997;45:238-246. [PubMed] [DOI] |
| 14. | Yamaguchi Y, Gojobori T. Evolutionary mechanisms and population dynamics of the third variable envelope region of HIV within single hosts. Proc Natl Acad Sci USA. 1997;94:1264-1269. [PubMed] [DOI] |