修回日期: 2003-08-10
接受日期: 2003-08-16
在线出版日期: 2004-02-15
目的: 筛选与克隆TAHCCP2的反式激活基因, 了解其可能存在的调节功能线索.
方法: 应用抑制性消减杂交(SSH)技术及生物信息学(bioin-formatics)技术筛选并克隆TAHCCP2反式激活的新型靶基因. 以TAHCCP2表达质粒pcDNA3.1(-)-TAHCCP2转染HepG2细胞, 以空载体pcDNA3.1(-)为平行对照, 制备转染后的细胞裂解液, 提取mRNA并逆转录为cDNA, 经Rsa I酶切后, 将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制性聚合酶链反应(PCR), 将产物与T/A载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR扩增后进行测序及同源性分析.
结果: 成功构建人TAHCCP2反式激活基因差异表达的cDNA消减文库. 文库扩增后得到70个阳性克隆, 进行菌落PCR分析, 均得到200-1 000 bp插入片段. 对插入片段测序, 并通过生物信息学分析获得其全长基因序列, 结果共获得15种编码基因.
结论: 筛选到的cDNA全长序列, 包括一些与细胞生长调节、物质代谢、免疫及细胞凋亡密切相关的蛋白编码基因, 推测了TAHCCP2可能存在的调控机制的线索, 尚需进一步的实验证明.
引文著录: 王建军, 刘妍, 成军, 杨倩, 纪冬, 党晓燕, 徐志强, 王春花. 应用抑制性消减杂交技术筛选TAHCCP2的反式调节基因. 世界华人消化杂志 2004; 12(2): 302-305
Revised: August 10, 2003
Accepted: August 16, 2003
Published online: February 15, 2004
AIM: To clone and identify human genes transactivated by TAHCCP2 by constructing a cDNA subtractive library with suppression subtractive hybridization technique.
METHODS: Suppression subtractive hybridization (SSH) and bioinformatics techniques were used for screening and cloning of the target genes transactivated by TAHCCP2 protein. The mRNA was isolated from HepG2 cells transfected with pcDNA3.1(-)-TAHCCP2 and pcDNA3.1(-) empty vector, respectively, and SSH method was employed to analyze the differentially expressed DNA sequence between the two groups. After restriction enzyme Rsa I digestion, small sizes cDNAs were obtained. Then tester cDNA was divided into two groups and ligated to the specific adaptor 1 and adaptor 2, respectively. After tester cDNA was hybridized with driver cDNA twice and underwent two times of nested PCR and then was subcloned into T/A plasmid vectors to set up the subtractive library. Amplification of the library was carried out with E. coli strain JM109. The cDNA was sequenced and analyzed in GenBank with Blast search after PCR.
RESULTS: The subtractive library of genes transactivated by TAHCCP2 was constructed successfully. The amplified library contained 70 positive clones. Colony PCR showed that these clones contained 200-1 000 bp inserts. Sequence analysis was performed in 32 clones, and the full length sequences were obtained with bioinformatics method. Altogether 15 coding sequences were achieved.
CONCLUSION: The obtained sequences may be target genes transactivated by TAHCCP2 among which some genes coding proteins involve in cell cycle regulation, metabolism, immunity and cell apoptosis. Advanced experiments need to be done to prove this finding.
- Citation: Wang JJ, Liu Y, Cheng J, Yang Q, Ji D, Dang XY, Xu ZQ, Wang CH. Screening and cloning of the target genes transactivated by TAHCCP2 using suppression subtractive hybridization technique. Shijie Huaren Xiaohua Zazhi 2004; 12(2): 302-305
- URL: https://www.wjgnet.com/1009-3079/full/v12/i2/302.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i2.302
丙型肝炎病毒(HCV)基因组含有单一的开放读码框架, 编码3 010-3 033个氨基酸残基(aa)的多蛋白前体, 两侧是5'-非翻译区及3'-非翻译区. 多蛋白前体至少被加工为10种结构蛋白和非结构蛋白, 其中核心区(1-573 nt)编码的21 kD HCV核衣壳蛋白(191 aa)是一种多功能蛋白质, 能够影响多种细胞信号转导途径, 激活多种病毒及细胞基因启动子, 具有广泛的反式激活作用[1-6], 最近研究还发现其与HCV感染后脂肪肝的形成也有一定关系[7-9], 在HCV致病过程中可能起着重要的作用. 本室利用抑制性消减杂交技术(suppression subtractive hybridization, SSH), 对于表达HCV 核心蛋白载体转染的HepG2细胞进行研究, 结合生物信息学技术(bioinformatics)克隆了HCV 核心蛋白反式激活作用的新靶基因, 命名为HCV核心蛋白反式激活基因2(TAHCCP2). 继续应用抑制性消减杂交技术筛选与克隆TAHCCP2反式激活的基因, 推测其在体内可能存在功能的线索, 为研究HCV的致病机制及探索未知基因的功能提供了新的方向.
HepG2细胞及感受态大肠杆菌JM109(本室保存), pcDNA3.1(-)真核表达载体(Invitrogen公司); FuGENE6 转染试剂(Roche公司), mRNA Purification试剂盒(Amersham Pharmacia Biotech公司), PCR-Select cDNA Subtraction试剂盒, 50碢CR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech公司), High Pure PCR Product Purification试剂盒(Boehringer Mannheim公司), T7、SP6通用引物及pGEM-Teasy载体(Promega公司).
1.2.1 消减杂交文库的建立及克隆分析: 分别将pcDNA3.1(-)-core及pcDNA3.1(-)空载体转染HepG2细胞, 48 h后提取mRNA, 按PCR-Select cDNA Subtraction试剂盒扩增差异表达的CDNA. 对于所获基因片段序列分析表明, 其中之一为新型基因片段, 与GenBank中注册的已知功能基因序列没有同源性, 利用表达序列标签(EST)序列的搜索和比对, 进行电子拼接, 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 确定新型基因序列. 从HepG2细胞提取总RNA, 以逆转录多聚酶链反应(RT-PCR)技术扩增获得该新基因的全长序列, 并测序证实, 命名为HCV核心蛋白反式激活基因2(TAHCCP2), 在GenBank中注册, 注册号为AY039043. TAHCCP2基因的编码序列全长为429个核苷酸(nt), 编码产物由142个氨基酸残基(aa)组成.
1.2.2 真核表达载体及细胞转染: TAHCCP2真核表达质粒pcDNA3.1(-)-TAHCCP2 由本室构建. 用FuGENE6 转染试剂将2 μg pcDNA3.1(-)-TAHCCP2及pcDNA3.1(-)空载体分别转染35 mm平皿HepG2 细胞, 48 h后收获细胞.
1.2.3 细胞mRNA提取: 使用mRNA Purification试剂盒, 直接提取转染了核心表达质粒及空载体的HepG2细胞mRNA, 经琼脂糖凝胶电泳及分光光度计进行定性定量分析.
1.2.4 双链cDNA(dscDNA)合成: 用Clontech公司的PCR-SelectTM cDNA Subtraction Kit中的试剂, 以获得的mRNA为模板逆转录合成cDNA.
1.2.5 消减杂交文库的建立: 采用Clontech公司的PCR-SelectTM cDNA Subtraction Kit[3], 常规SSH方法按说明书进行: 转染了核心表达质粒及空载体的HepG2 细胞cDNA分别标记为Tester和Driver, 经RsaⅠ(一种识别4碱基序列的内切酶)消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的cDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver cDNA进行杂交; 合并两种杂交产物后再与Driver cDNA作第2次杂交; 然后将杂交产物做选择性PCR扩增, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增.
1.2.6 克隆鉴定分析: 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(100-1 000 bp), 测序(上海申友公司). 应用生物信息学将测得序列GenBank数据库进行同源性分析.
TAHCCP2真核表达质粒pcDNA3.1(-)-TAHCCP2经测序及酶切鉴定均正确.
使用高质量的mRNA是保证cDNA高产量的前提. 紫外分光检测显示, 转染了pcDNA3.1(-)-TAHCCP2质粒及空载体的HepG2 细胞mRNA分别为4.64 μg和4.38 μg, A260/A280 = 1.87. 1.0%琼脂糖凝胶电泳见 mRNA为大于0.5 kb清晰慧尾片状条带.
dscDNA与接头连接效率的高低是决定抑制性消减杂交成败的最关键步骤. 将连接有adaptor l和adaptor 2的两组dscDNA分别用不同的特异性引物(看家基因甘油三磷酸脱氢酶G3PDH引物)进行28个循环扩增, 产物用2.0%琼脂糖凝胶电泳鉴定. 结果显示两组dscDNA扩增产物浓度相当, 说明dscDNA已与接头高效率连接.
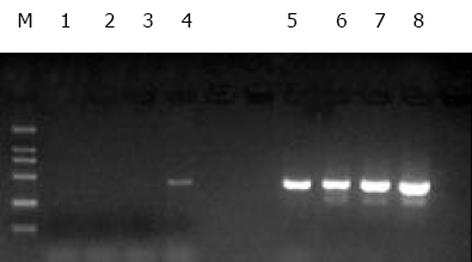
分别取l μL抑制性及非抑制性PCR产物为模板, 用看家基因G3PDH引物进行PCR扩增, 分别在18、23、28、33次循环结束时从体系中吸取5 μL进行电泳鉴定. 结果显示: 与非抑制性PCR产物相比, 抑制性PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图1).
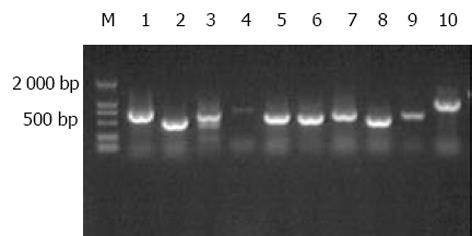
杂交产物经两轮PCR扩增后, 菌落PCR扩增结果显示为200-1 000 bp大小不等的插入片段, 所获得的70个克隆中几乎均含有插入片段, 这些条带可能代表差异表达的基因片段(图2).
挑选其中32个克隆测序, 与GenBank数据库进行初步比较. 有32个与已知基因的部分序列高度同源(98%-100%), 详细结果见表1.
| 同源蛋白名称 | 同源菌株 | 同源性 |
| 1 核糖体蛋白 | 5 | 98-100% |
| 2 线粒体单倍型基因 | 4 | 99-100% |
| 3 二氢乙醇脱氢酶 | 3 | 99% |
| 4 血栓粘合素1 | 1 | 98% |
| 5 纤维连接蛋白1 | 4 | 99-100% |
| 6 白蛋白 | 4 | 99-100% |
| 7 肿瘤排斥抗原1 | 1 | 99% |
| 8 结缔组织生长因子3'非翻译区 | 1 | 100% |
| 9 前列腺结合蛋白 | 1 | 100% |
| 10 硬脂酰辅酶A脱氢酶 | 1 | 99% |
| 11 苯丙氨酸羟化酶 | 1 | 100% |
| 12 载脂蛋白H | 2 | 100% |
| 13 丝氨酸/苏氨酸激酶19 | 1 | 100% |
| 14 真核翻译延长因子1 | 2 | 100% |
| 15 包涵素重链多肽 | 1 | 99% |
HCV核心蛋白是一种多功能蛋白质, 除了作为核壳蛋白具有病毒颗粒组装功能外, 还具有多种调控细胞、病毒基因表达、细胞生长以及免疫调节等功能. 研究证实核心蛋白也是一种反式激活蛋白, 甚至其作用还要超过X蛋白[10-11]. 核心蛋白对细胞信号转导途径, 尤其是核因子-κB (NF-κB)、激活因子1(AP-1)和血浆反应因子(SRE)相关途径具有明显的增强作用[12]; 在HepG2细胞中, 核心蛋白激活人c-myc基因、劳氏肉瘤病毒长末端重复序列(RSV-LTR)和SV40早期启动子[13]; 核心蛋白还能抑制或增强p53基因启动子功能[14-15], 这些证据表明核心蛋白具有潜在的致癌作用. 近来研究还发现HCV核心蛋白可与细胞内的一些细胞凋亡因子作用, 促进细胞凋亡[16-20]. 也有人发现HCV核心蛋白启动CAP-Rf反式激活能力可能是通过复合体形式和cDNA编码的DEAD盒家族中推定的RNA解旋酶(CAP-Rf ATP酶)活性调节, 表明HCV已经进化了一种特殊机制, 通过其核壳蛋白涉及RNA代谢[21]. Aoki et al[22]发现HCV核心蛋白通过和14-3-3蛋白相互作用表现出一种新型的Raf-1激酶激活蛋白特性, 可能对肝细胞的生长起调节作用. HCV核心蛋白的反式激活功能在HCV致病中发挥重要的作用, 研究其作用分子生物学机制有助于理解HCV感染的慢性化和致癌作用机制. 本室利用SSH, 对于表达HCV核心蛋白载体转染的HepG2细胞进行研究, 结合生物信息学技术(bioinformatics)克隆了HCV核心蛋白反式激活作用的新靶基因, 命名为HCV核心蛋白反式激活基因2(TAHCCP2), 并构建其真核表达载体TAHCCP2-cDNA3.1(-). 继续应用SSH技术筛选与克隆TAHCCP2反式激活的基因, 推测其在体内可能存在功能的线索.
SSH方法是近年发展起来的一项新的基因克隆技术, 与传统的方法比较, 具有实验周期短、易操作、可靠性高、假阳性率低等特点, 能有效地分离扩增低丰度特异表达的基因, 可以在较短的时间内获得较理想的实验结果[23]. 我们将真核表达载体pcDNA3.1(-)-TAHCCP2, 转染肝母细胞瘤细胞系HepG2, 并以转染空白载体的相同细胞系作为对照, 以2种转染的细胞系中提取的mRNA为起始材料, 应用SSH方法成功地构建了TAHCCP2反式激活相关基因差异表达的cDNA消减文库, 挑选32个克隆测序分析, 结果主要包括 (1)细胞内结构与细胞生长相关蛋白, 如线粒体蛋白、核糖体蛋白、包涵素重链多肽为细胞内结构蛋白、血栓粘合素1(THBS1)、纤维连接蛋白1(FN1)真核翻译延长因子在细胞生长、分化、黏附中起重要作用. (2)参与细胞内物质代谢的蛋白基因, 如白蛋白在各种物质代谢中起运输作用, 并且是维持血浆胶体渗透压的重要物质. 硬脂酰辅酶A脱氢酶(SCD), 参与体内脂肪的合成过程, 其升高可能会增加体内不饱和脂肪酸的合成. 苯丙氨酸羟化酶(PAH), 催化苯丙氨酸转化成酪氨酸的酶, 他调控苯丙氨酸在体内的分解代谢. 二氢乙醇脱氢酶是肝内多环芳香烃代谢重要的酶, 最近研究发现, 大约40%的HCC患者中表达显著降低. 然而, 在肝肿瘤的边缘区域表达增高, 并且增高的水平与肿瘤的大小和病情的进展相关[24]. 还有人发现其在可检测到的非小细胞肺癌可能是早期而非进展期生化指标[25]. 在卵巢肿瘤中其表达可诱导对顺铂药物治疗肿瘤的抵抗作用[26]. 载脂蛋白H在减少甘油三酯, 增加脂蛋白脂肪酶方面有一定作用[27], 对低密度脂蛋白的氧化有类似抗氧化剂的作用, 可以抑制胆固醇从细胞外易位到巨噬细胞内[28]. 研究还发现其在止血和产生抗磷酯抗体方面有作用[29], 体外可影响巨噬细胞对表面包含磷脂酰丝氨酸颗粒的摄取, 如凋亡的胸腺细胞和单板层囊泡一样. 介导体内衰老细胞的祛除作用, 所以对细胞凋亡可能有一定作用. (3)与机体免疫功能相关的蛋白基因, 如肿瘤排斥抗原1与体内的抗肿瘤免疫相关. 前列腺结合蛋白包括C1、C2、C3三种成分, 形成两种不同的二聚体(C1-C3H和C2-C3), 最近研究表明前列腺类固醇结合蛋白他是一种免疫抑制因子, 可以保护精子免受女性生殖道免疫损伤. 丝氨酸/苏氨酸激酶被认为可调节C-fos和p27基因的表达, 并且参与对细胞调亡的调节, 可以抑制前调亡蛋白的活性, 抑制T细胞的活性. (4)与肝损伤后纤维化作用密切相关的基因, 如结缔组织生长因子, 在纤维化损伤时表达增加的前纤维化基因分子, 可刺激结缔组织细胞增生, 引起组织纤维化[1], 与肝纤维化相关. 他的表达增强可能与丙型肝炎患者肝硬化发病机制有一定关系.
通过对TAHCCP2的上述反式激活基因的分析, 我们发现他与体内物质代谢尤其是脂肪代谢关系较密切, 并且对细胞分化生长与纤维化也有一定关系, 能在病毒感染后肝纤维化方面有一定的作用. 另外, 他也可能负性调节体内的某些免疫反应. TAHCCP2是正常人体存在的基因, 在病毒蛋白NS5A的存在下其功能被进一步激活. 关于其在体内与各种活性因子的具体调节机制, 仍需进一步实验来验证.
编辑: N/A
| 2. | Cheng J. Molecular pathogenesis of viral hepatitis. J Gastroenterol Hepatol. 2002;16:A185. |
| 5. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 洪 源, 夏 小兵. 酵母双杂交技术筛选克隆HCV核心蛋白结合蛋白基因1. 世界华人消化杂志. 2001;9:1379-1383. [DOI] |
| 6. | Sato C. [Effects of hepatitis C virus proteins on the interferon-stimulated signal transduction]. Nihon Rinsho. 2001;59:1271-1276. [PubMed] |
| 7. | 王 琳, 李 克, 成 军, 陆 荫英, 张 健, 洪 源, 刘 妍, 王 刚, 钟 彦伟, 段 惠娟. 丙型肝炎病毒核心蛋白与载脂蛋白AI结合的研究. 世界华人消化杂志. 2002;10:1018-1021. [DOI] |
| 8. | 成 军, 任 进余, 李 莉, 陆 志檬, 李 克, 洪 源, 陆 荫英, 王 刚, 刘 妍, 张 玲霞. 丙型肝炎病毒结构基因转基因小鼠引起肝脏脂肪变. 世界华人消化杂志. 2002;10:1022-1026. [DOI] |
| 11. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 李 莉, 张 玲霞, 陈 菊梅. HCV核心蛋白与截短型HBV表面抗原中蛋白的协同反式激活功能. 中华肝脏病杂志. 2002;10:354-357. |
| 12. | Kato N, Yoshida H, Ono-Nita SK, Kato J, Goto T, Otsuka M, Lan K, Matsushima K, Shiratori Y, Omata M. Activation of intracellular signaling by hepatitis B and C viruses: C-viral core is the most potent signal inducer. Hepatology. 2000;32:405-412. [PubMed] [DOI] |
| 13. | Chang J, Yang SH, Cho YG, Hwang SB, Hahn YS, Sung YC. Hepatitis C virus core from two different genotypes has an oncogenic potential but is not sufficient for transforming primary rat embryo fibroblasts in cooperation with the H-ras oncogene. J Virol. 1998;72:3060-3065. [PubMed] |
| 14. | Ray RB, Steele R, Meyer K, Ray R. Transcriptional repression of p53 promoter by hepatitis C virus core protein. J Biol Chem. 1997;272:10983-10986. [DOI] |
| 15. | Lu W, Lo SY, Chen M, Wu Kj, Fung YK, Ou JH. Activation of p53 tumor suppressor by hepatitis C virus core protein. Virology. 1999;264:134-141. [PubMed] [DOI] |
| 16. | Hahn CS, Cho YG, Kang BS, Lester IM, Hahn YS. The HCV core protein acts as a positive regulator of fas-mediated apoptosis in a human lymphoblastoid T cell line. Virology. 2000;276:127-137. [PubMed] [DOI] |
| 17. | Honda A, Hatano M, Kohara M, Arai Y, Hartatik T, Moriyama T, Imawari M, Koike K, Yokosuka O, Shimotohno K. HCV-core protein accelerates recovery from the insensitivity of liver cells to Fas-mediated apoptosis induced by an injection of anti-Fas antibody in mice. J Hepatol. 2000;33:440-447. [PubMed] [DOI] |
| 18. | Rubbia-Brandt L, Taylor S, Gindre P, Quadri R, Abid K, Spahr L, Negro F. Lack of in vivo blockade of Fas- and TNFR1-mediated hepatocyte apoptosis by the hepatitis C virus. J Pathol. 2002;197:617-623. [PubMed] [DOI] |
| 19. | Farooq M, Kim Y, Im S, Chung E, Hwang S, Sohn M, Kim M, Kim J. Cloning of BNIP3h, a member of proapoptotic BNIP3 family genes. Exp Mol Med. 2001;33:169-173. [PubMed] [DOI] |
| 20. | Gross A, McDonnell JM, Korsmeyer SJ. BCL-2 family members and the mitochondria in apoptosis. Genes Dev. 1999;13:1899-1911. [PubMed] [DOI] |
| 21. | You LR, Chen CM, Yeh TS, Tsai TY, Mai RT, Lin CH, Lee YH. Hepatitis C virus core protein interacts with cellular putative RNA helicase. J Virol. 1999;73:2841-2853. [PubMed] |
| 22. | Aoki H, Hayashi J, Moriyama M, Arakawa Y, Hino O. Hepatitis C virus core protein interacts with 14-3-3 protein and activates the kinase Raf-1. J Virol. 2000;74:1736-1741. [PubMed] [DOI] |
| 23. | Kuang WW, Thompson DA, Hoch RV, Weigel RJ. Differential screening and suppression subtractive hybridization identified genes differentially expressed in an estrogen receptor-positive breast carcinoma cell line. Nucleic Acids Res. 1998;26:1116-1123. [PubMed] [DOI] |
| 24. | Yang MD, Wu CC, Chiou SH, Chiu CF, Lin TY, Chiang IP, Chow KC. Reduction of dihydrodiol dehydrogenase expression in resected hepatocellular carcinoma. Oncol Rep. 2003;10:271-276. [PubMed] [DOI] |
| 25. | Chen CY, Hsu CP, Hsu NY, Shih CS, Lin TY, Chow KC. Expression of dihydrodiol dehydrogenase in the resected stage I non-small cell lung cancer. Oncol Rep. 2002;9:515-519. [PubMed] [DOI] |
| 26. | Deng HB, Parekh HK, Chow KC, Simpkins H. Increased expression of dihydrodiol dehydrogenase induces resistance to cisplatin in human ovarian carcinoma cells. J Biol Chem. 2002;277:15035-15043. [PubMed] [DOI] |
| 27. | Takada D, Ezura Y, Ono S, Iino Y, Katayama Y, Xin Y, Wu LL, Larringa-Shum S, Stephenson SH, Hunt SC. Apolipoprotein H variant modifies plasma triglyceride phenotype in familial hypercholesterolemia: a molecular study in an eight-generation hyperlipidemic family. J Atheroscler Thromb. 2003;10:79-84. [PubMed] [DOI] |
| 28. | Lin KY, Pan JP, Yang DL, Huang KT, Chang MS, Ding PY, Chiang AN. Evidence for inhibition of low density lipoprotein oxidation and cholesterol accumulation by apolipoprotein H (beta2-glycoprotein I). Life Sci. 2001;69:707-719. [PubMed] [DOI] |
| 29. | Mehdi H, Manzi S, Desai P, Chen Q, Nestlerode C, Bontempo F, Strom SC, Zarnegar R, Kamboh MI. A functional polymorphism at the transcriptional initiation site in beta2-glycoprotein I (apolipoprotein H) associated with reduced gene expression and lower plasma levels of beta2-glycoprotein I. Eur J Biochem. 2003;270:230-238. [PubMed] [DOI] |