修回日期: 2004-09-13
接受日期: 2004-09-19
在线出版日期: 2004-11-15
目的: 筛选与大肠癌相关的差异表达基因.
方法: 用包含8 000个cDNA的基因芯片法检测4例大肠高分化腺癌组织及其癌旁正常肠黏膜组织标本RNA表达谱, 通过对比分析寻找与大肠癌相关的基因.
结果: 大肠癌差异表达基因中有1 807个基因发生了显著性变化, 其中上调表达的基因有936个, 最有显著上调的有36个; 下调表达的基因有871个, 最有显著下调的有30个.
结论: 大肠癌的发生是一个多基因表达失调的过程, 大肠癌与癌旁正常肠黏膜组织间存在差异表达的基因.
引文著录: 南清振, 张振书, 杨希山, 肖冰, 高蕾, 叶方鹏, 武金保. 应用基因芯片技术筛查大肠癌相关基因. 世界华人消化杂志 2004; 12(11): 2742-2744
Revised: September 13, 2004
Accepted: September 19, 2004
Published online: November 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(11): 2742-2744
- URL: https://www.wjgnet.com/1009-3079/full/v12/i11/2742.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i11.2742
随着人类基因组计划的开发研究和分子生物学、分子遗传学的迅速发展成熟, 人类对肿瘤的认识已进入分子水平[1]. 寻找大肠癌相关基因, 深入了解该相关基因在肿瘤发生及转移过程中的作用已成为可能. 基因芯片技术适用于大规模的基因检测和基因功能的研究、疾病发生机制的研究及临床诊断等方面[2-4]. 本研究系选用有8000条人类基因的cDNA表达谱芯片, 以临床切除的大肠癌及癌旁正常肠黏膜组织标本为研究对象, 筛查大肠癌发生、发展过程中与肿瘤相关的基因, 为大肠癌的早期诊断与治疗提供理论依据.
我院2003年收治的4例大肠高分化腺癌患者组织标本和其对应癌旁5 cm以上的正常大肠黏膜组织. 标本切除后立即置于液氮, 冻存备用. TRIzol试剂购自Life Technologies公司, dNTP购自Promega公司; Taq酶购自上海生物工程服务公司; 逆转录酶及缓冲液购自Gibco公司; 荧光素3标记的脱氧尿嘧啶三磷酸(Cy3 dUTP)、荧光素标记的5脱氧尿嘧啶三磷酸(Cy5 dUTP)购自美国Amersham Pharmacia公司; 总RNA提取试剂盒购自德国Qiagen公司. 芯片类型(微芯公司基因表达谱芯片CSC-GE-80), Gen III Microarray Spotter芯片点样仪购自Amersham Pharmacia公司; 恒温杂交箱购自Shellab公司; 芯片扫描仪购自Amersham Pharmacia公司; 聚合酶链反应(PCR)仪购自MJ Research公司; 扫描图像显示用软件为ImageQuant (Amersham Pharmacia Biotech, Ltd); 扫描图像分析用软件为ArrayVision(Imaging Research, Ltd). 芯片实验数据分析软件为MIDAS(Chipscreen Biosciences, Ltd).
按一步法抽提、纯化大肠癌及正常大肠组织的总RNA. 用无RNA酶的三蒸水溶解沉淀, 紫外分光分析及电泳检测显示, 获得高质量的总RNA. 参照Draghici et al[5]方法行逆转录标记cDNA探针并纯化. 在链合成中掺入荧光素标记的dUTP, 用Cy3 dUTP标记正常组织mRNA, 用Cy5 dUTP标记大肠癌组织mRNA. 检测标记的荧光含量. 用分光光度计检测260 nm, 550 nm, 650 nm时的A值, 然后计算出荧光含量. 将标记好的探针(Cy3/Cy5)按等量混匀, 溶解在30 μL的杂交缓冲液中. 将芯片和杂交混合探针分别置于95 ℃双蒸水浴中变性5 min, 立即将混合探针加在芯片上, 封片(不能留有气泡), 置杂交舱, 密闭, 42 ℃孵箱恒温杂交16 h, 洗片, 晾干. 玻片完全干燥后, 放入扫描片夹中, 利用扫描仪进行扫描; 扫描后将图像转化为基于荧光强度的数字信号, 随后进行数据分析和处理. 用以下3个条件作为判定基因差异表达的标准: (1)Cy3和Cy5信号的比值相差2倍(上调Cy3/Cy5>2.0或下调<0.5)以上; (2)Cy3或Cy5, Cy3和Cy5信号值其中之一必须>600; (3)聚合酶链反应实验结果良好. 同时要求筛选出的内对照基因表达数据在所有标本中重复[6-9].
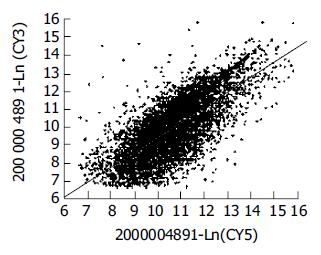
大肠癌组织A260/A280值达1.85, 而癌旁正常肠黏膜组织的A260/A280值1.96, 电泳结果证实高纯化的总RNA. 部分基因芯片杂交结果及所有基因点CY3/CY5信号强度散点图(图1, 2). 用微矩阵基因芯片筛选4例大肠癌和癌旁正常肠黏膜组织的差异表达的基因, 按照我们的数据筛选标准, 芯片结果中有1 807个基因发生了显著性变化, 其中上调表达的基因有936个, 最有显著性上调的有36个; 下调表达的基因有871个, 最有显著性下调的有30个. 筛选出的有显著差异性的基因按照功能进行分类可以发现他们涉及到细胞生长的各个方面, 包括细胞增生, 细胞凋亡, 细胞代谢以及细胞骨架结构, 同时还涉及到各种原癌基因和肿瘤抑制基因的表达变化. 这反映了肿瘤生长过程涉及到各方面的基因表达变化.
我们采用的微矩阵基因芯片是以微芯基因科技公司的8 000条基因为靶基因, 制备成cDNA芯片, 以临床切除的大肠癌及正常大肠黏膜标本为研究对象, 对这两种组织总RNA逆转录产生的cDNA进行杂交检测, 寻找和筛查大肠癌相关基因, 探讨大肠癌在发生、发展过程中各种基因的表达变化, 为大肠癌的早期诊治提供理论依据. 软件统计分析微矩阵扫描结果提示, 大肠癌发生中有众多基因发生了分子生物学改变(个体差异存在不同), 其中有1 807个基因发生了显著性变化, 上调表达的基因有936个, 最有显著性上调的有36个; 下调表达的基因有871个, 最有显著性下调的有30个. 而且发现在大肠癌的发生、发展中有多类基因的参与, 他们涉及到细胞生长的各个方面, 包括细胞增生, 细胞凋亡, 细胞代谢以及细胞骨架结构, 同时还涉及到各种原癌基因和肿瘤抑制基因的表达变化. 证实大肠癌中基因改变的多样性, 进一步推断肿瘤的发生、发展是一多基因、多阶段过程[10-12].
由于肿瘤部位所包含的细胞类型的多样性, 使得人们在解释这些相关基因的表达变化与肿瘤中特定细胞类型的表型变化的相关性时遇到一定的困难. 但是对于那些有非常明显变化的基因和主要的细胞类型间的相关性仍然可以在进一步的研究中得到明确[13-15]. 我们按照相关的功能挑选了部分基因可以看出一些与肿瘤相关的基因表达变化, 如细胞凋亡相关基因的下调表达, 但是同时也注意到一些细胞凋亡启动基因的表达上调, 这其中可能反映了肿瘤对抗细胞凋亡的内在机制[16-17]; 而在原癌基因方面, 大部分发生了表达上调, 但是也有部分表达下调, 这可能与不同原癌基因在不同肿瘤发生中的作用不同有关; 其他如细胞骨架蛋白相关基因的表达变化则与肿瘤细胞的生长和转移特性密切相关[18].
编辑: N/A
| 4. | Man XB, Tang L, Qiu XH, Yang LQ, Cao HF, Wu MC, Wang HY. Expression of cytochrome P4502E1 gene in hepatocellular carcinoma. World J Gastroenterol. 2004;10:1565-1568. [PubMed] [DOI] |
| 5. | Draghici S, Kulaeva O, Hoff B, Petrov A, Shams S, Tainsky MA. Noise sampling method: an ANOVA approach allowing robust selection of differentially regulated genes measured by DNA microarrays. Bioinformatics. 2003;19:1348-1359. [DOI] |
| 6. | Yang IV, Chen E, Hasseman JP, Liang W, Frank BC, Wang S, Sharov V, Saeed AI, White J, Li J. Within the fold: assessing differential expression measures and reproducibility in microarray assays. Genome Biol. 2002;3:research0062. [PubMed] |
| 8. | 刘 妍, 成 军, 王 春花, 杨 倩, 王 建军, 纪 冬. 基因表达谱芯片技术筛选XTP4基因转染细胞差异表达基因. 胃肠病学和肝病学杂志. 2004;13:209-213. |
| 9. | Li SR, Dorudi S, Bustin SA. Identification of differentially expressed genes associated with colorectal cancer liver metastasis. Eur Surg Res. 2003;35:327-336. [PubMed] [DOI] |
| 11. | 潘 胜武, 孙 安乐, 刘 变英, 崔 大祥, 闫 小君, 栗 彤, 雷 宇锋, 王 胜. 大肠癌相关基因表达的早期诊断意义. 世界华人消化杂志. 2000;8:1431-1432. [DOI] |
| 12. | Frederiksen CM, Knudsen S, Laurberg S, Ørntoft TF. Classification of Dukes' B and C colorectal cancers using expression arrays. J Cancer Res Clin Oncol. 2003;129:263-271. [PubMed] |
| 14. | Gupta V, Cherkassky A, Chatis P, Joseph R, Johnson AL, Broadbent J, Erickson T, DiMeo J. Directly labeled mRNA produces highly precise and unbiased differential gene expression data. Nucleic Acids Res. 2003;31:e13. [PubMed] [DOI] |
| 15. | Skubitz KM, Skubitz AP. Gene expression in aggressive fibromatosis. J Lab Clin Med. 2004;143:89-98. [PubMed] [DOI] |
| 16. | Kluger HM, Kluger Y, Gilmore-Hebert M, DiVito K, Chang JT, Rodov S, Mironenko O, Kacinski BM, Perkins AS, Sapi E. cDNA microarray analysis of invasive and tumorigenic phenotypes in a breast cancer model. Lab Invest. 2004;84:320-331. [PubMed] [DOI] |
| 17. | Williams NS, Gaynor RB, Scoggin S, Verma U, Gokaslan T, Simmang C, Fleming J, Tavana D, Frenkel E, Becerra C. Identification and validation of genes involved in the pathogenesis of colorectal cancer using cDNA microarrays and RNA interference. Clin Cancer Res. 2003;9:931-946. [PubMed] |
| 18. | Ognjanovic S, Tashima LS, Bryant-Greenwood GD. The effects of pre-B-cell colony-enhancing factor on the human fetal membranes by microarray analysis. Am J Obstet Gynecol. 2003;189:1187-1195. [PubMed] [DOI] |