修回日期: 2004-08-09
接受日期: 2004-08-16
在线出版日期: 2004-10-15
目的: 筛选丙型肝炎病毒(HCV)非结构蛋白4B(NS4B)转染细胞差异表达基因, 进一步阐明NS4B蛋白在丙型肝炎慢性化及致肝细胞癌发生发展过程中的分子生物学机制.
方法: 根据HCV-H病毒株序列设计、合成HCV NS4B基因序列特异性的引物, 以含有HCV-H全基因组cDNA的质粒pBRTM3011作为模板, 应用聚合酶链反应(PCR)技术扩增NS4B蛋白编码基因片段, 以常规的分子生物学技术将获得的HCV NS4B编码基因片段克隆到TA载体中进行核苷酸序列的测定, 构建真核表达载体pcDNA3.1(-)-NS4B. 以脂质体转染肝母细胞瘤细胞系HepG2, 提取mRNA, 逆转录为cDNA, 与转染空白表达载体pcDNA3.1(-)的HepG2细胞进行DNA芯片分析.
结果: 构建的表达载体经过限制性内切酶分析和DNA序列测定, 证实准确无误. 以单链可变区抗体的Western blot杂交技术证实构建的表达载体转染HepG2细胞之后有NS4B蛋白的表达, 提取高质量的mRNA并逆转录成为cDNA, 进行DNA芯片技术分析. 在1 152个基因表达谱的筛选中, 发现有22个基因表达水平显著上调, 34个基因表达水平显著下调.
结论: 应用基因表达谱芯片成功筛选了HCV NS4B转染细胞后差异表达基因, 为进一步阐明NS4B蛋白致病的分子生物学机制提供依据.
引文著录: 刘妍, 王建军, 成军, 杨倩, 纪冬, 王春花, 党小艳, 徐志强. 筛选丙型肝炎病毒非结构蛋白4B转染细胞差异表达基因. 世界华人消化杂志 2004; 12(10): 2316-2320
Revised: August 9, 2004
Accepted: August 16, 2004
Published online: October 15, 2004
AIM: To screen genes differently expressed in human hepatoblastoma cell line HepG2 transfected with non-structural protein 4B (NS4B) of hepatitis C virus (HCV) , and to further elucidate the molecular biological mechanism of NS4B in chronic hepatitis C and carcinogenesis, and progression of hepatoma.
METHODS: Sequence-specific primers of HCV NS4B were designed and synthesized. The plasmid pBRTM3011, in which the full length of HCV-H cDNA genome was contained, was treated as the template to amplify the NS4B-coded DNA fragment with polymerase chain reaction (PCR) technique. The expressive vector of pcDNA3.1(-)-NS4B was constructed by routine molecular biological methods. The technology of cDNA microarray was adopted to detect the mRNA extracted from the HepG2 cells transfected with pcDNA3.1(-)-NS4B and pcDNA3.1(-) using lipofectamine, respectively. The expression of NS4B protein in the transfected vector was confirmed by Western blot with single chain variable region antibody.
RESULTS: The expressive vector was constructed and confirmed after restriction enzyme digestion and DNA sequencing analysis. The expression of NS4B protein in the transfected vector was confirmed by Western blot with single chain variable region antibody. High quality mRNA and cDNA were prepared. Among 1 152 genes of the DNA microarray, we found 56 genes were differently expressed in HepG2 cells transfected with NS4B, in which 22 genes were significantly up-regulated and 34 were significantly down-regulated.
CONCLUSION: Differently expressed genes are successfully screened in HepG2 cells transfected with NS4B by cDNA microarray, which may help to further elucidate the molecular mechanism of NS4B in HCV infection and development of hepatocellular carcinoma.
- Citation: Liu Y, Wang JJ, Cheng J, Yang Q, Ji D, Wang CH, Dang XY, Xu ZQ. Screening of genes differentially expressed in HepG2 cells transfected with non-structural protein 4B ofhepatitis C virus. Shijie Huaren Xiaohua Zazhi 2004; 12(10): 2316-2320
- URL: https://www.wjgnet.com/1009-3079/full/v12/i10/2316.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i10.2316
丙型肝炎病毒(HCV)非结构蛋白4B(NS4B)基因由783个核苷酸组成, 编码由261个氨基酸残基组成的疏水蛋白(分子量为27 kD)[1].近年研究表明HCV NS4B蛋白定位于细胞内质网膜, 与细胞内膜结构的功能有关[2]. 在体外实验中, 他和NS4A蛋白一起可以抑制宿主细胞的翻译, 在一定程度上可以减弱机体对抗病毒药物干扰素-a(INF-a)的反应, 同时使病毒逃避宿主的免疫作用. NS4B蛋白还可以与HCV 其他非结构蛋白NS3、NS4A、NS5A、NS5B等相互作用, 对这些非结构蛋白的功能起协同、促进甚或是下调作用[3-5]. 可见HCV NS4B蛋白是一种多功能的蛋白质, 在HCV病毒感染及致肿瘤发生过程中起着一定的作用. 基因芯片技术(cDNA microarray)是由大量目的基因片段有序、密集地固定于玻片或尼龙膜上而制成芯片, 将两组组织或细胞的mRNA逆转录成cDNA, 掺入荧光标记, 同时与芯片杂交, 通过扫描分析每一位置的荧光信息可以快速有效地检测到二者间差异表达的基因[6-8].
本研究应用基因芯片技术, 筛选HCV NS4B基因转染细胞后差异表达的基因, 检测NS4B蛋白的表达对肝细胞基因表达谱的影响, 为深入了解HCV NS4B蛋白在HCV病毒慢性感染及致肿瘤发生过程中的作用机制提供理论依据.
肝母细胞瘤细胞系HepG2细胞及含有HCV-H全基因组cDNA的质粒pBRTM3011(美国Rockfeller大学的Rice CM教授惠赠)由本室保存, 细胞培养相关试剂、总RNA提取试剂Trizol及真核表达载体pcDNA3.1(-)均购自Invitrogen公司. 人类基因组分类I芯片包括原癌基因和抑癌基因、免疫调节相关基因、细胞凋亡和应激反应蛋白相关基因、细胞信号转导相关基因等1 152个cDNA, 由上海联合基因有限公司提供. mRNA纯化试剂Oligotex mRNA Midi Kit 购自Qiagen公司.
1.2.1 真核表达载体构建及细胞转染: 根据HCV-H病毒株的基因序列设计、合成HCV NS4B基因序列特异性引物, 上下游引物序列分别为: 5'-GAA TTC ATG TCT CAG CAC TTA CCG TAC-3', 5'-GGA TCC TCA GCA TGG AGT GGT ACA CTC-3', 下划线部分为引物两端的酶切位点, EcoRI和BamHI. 以含有全长HCV-H株的cDNA质粒pBRTM3011作为模板, 应用聚合酶链反应(PCR)技术扩增NS4B的全长编码基因. 先克隆到TA载体中进行序列测定, 然后再亚克隆到真核表达载体pcDNA3.1(-)中, 构建真核表达载体pcDNA3.1(-)-NS4B. 在35 mm培养皿中常规培养HepG2细胞, 细胞生长至对数期时, 分别以脂质体转染试剂Lipofectamine PLUS将2 mg pcDNA3.1(-)-NS4B和空载体pcDNA3.1(-) 转染HepG2细胞, 48 h后收获细胞, 每5×106个细胞加入1 mL Trizol试剂, 立即于液氮中保存.
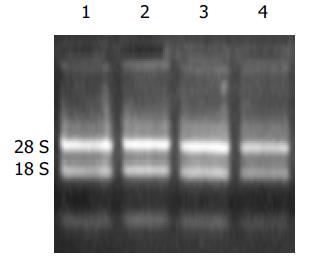
1.2.2 总RNA提取及mRNA纯化: 使用Trizol试剂一步法提取转染pcDNA3.1(-)-NS4B和空载体pcDNA3.1(-)的细胞HepG2总RNA(分别标记为实验组和对照组), 样品经分光光度计检测吸光度A值, 并行热稳定实验, 于-20 ℃和70 ℃保温1 h后, 经琼脂糖凝胶电泳检测28 S、18 S条带变化. 纯化mRNA并行电泳检测.
1.2.3 探针标记及芯片制备: 常规方法逆转录标记cDNA探针并纯化. Cy3-dUTP标记对照组细胞mRNA(5 mg), Cy5-dUTP标记实验组细胞mRNA(5 mg). 乙醇沉淀后溶解在20 mL 5×SSC+2 g/L SDS杂交液中. 芯片包含的1 152个cDNA以通用引物进行PCR扩增, PCR产物长度为1 000-3 000 bp. 靶基因以0.5 mg / mL溶解于3×SSC溶液中, 用Cartesian公司的Cartesian 7 500 点样仪及TeleChem公司的硅烷化玻片进行点样. 玻片经水合(2 h)、室温干燥(30 min), UV交联, 再分别用0.2% SDS、水及硼氢化钠溶液处理10 min, 晾干备用.
1.2.4 杂交及洗涤: 将基因芯片和杂交探针在95 ℃水浴变性5 min, 将混合探针加在基因芯片上, 置于60 ℃杂交15-17 h. 依次以2×SSC+2 g/L SDS、1 mL/L×SSC +2 g/L SDS、1 mL/L×SSC洗涤10 min, 室温晾干.
1.2.5 检测与分析: 用General Scanning公司的ScanArray 3 000扫描芯片. 用预先选定的内参照基因(24条管家基因, 每个基因点2个点, 共48个点)对Cy3和Cy5的原始提取信号进行均衡和修正. 用ImaGene3.0软件分析Cy3、Cy5两种荧光信号的强度, 计算Cy5/Cy3比值. 阳性结果判断: Cy5/Cy3>1.9, 红色荧光, 显示表达增强; Cy5/Cy3<0.6, 为绿色荧光, 显示表达减弱.
真核表达载体pcDNA3.1(-)-NS4B经过限制性内切酶作图分析和核苷酸序列的测定, 证实含有完整的开放读码框架, 序列准确无误. 以单链可变区抗体的Western blot杂交技术证实构建的表达载体转染HepG2细胞之后有NS4B蛋白的表达.
实验组和对照组总RNA的吸光度比值A260/A280分别为2.015和2.009, 热稳定实验70 ℃保温1 h与-20 ℃ 1 h电泳条带比较, 显示28 S条带无明显降解, 10g/L琼脂糖凝胶电泳结果证实已抽提高纯度的总RNA(图1). mRNA主要集中于0.9-4.0 kb的连续条带.
在芯片上共有1 152个cDNA. 为了监控芯片杂交技术体系的整个过程, 在芯片上设置了阴性对照(8条水稻基因, 共8个点), 这些点的杂交信号均很低, 证实了数据的可靠性. 由于实验组探针标记Cy5荧光素(呈红色), 对照组探针标记Cy3荧光素(呈绿色), 红绿颜色的差异就显示该基因在实验组和对照组中基因表达水平上的差异, 黄色代表表达水平无差异. 按阳性标准, 从1 152个基因中筛选出差异表达基因共56条, 占4.86%, 其中22条基因表达增强, 34条基因表达降低.
表达增强的基因主要有: 细胞凋亡相关基因, 如凋亡相关的RNA结合蛋白, 含有死亡效应域蛋白DEDD;细胞增生分化相关基因, 如转化生长因子b受体1, 肿瘤坏死因子受体超家族成员1b, 肿瘤坏死因子配体超家族成员10; 细胞信号转导相关蛋白, 如cAMP依赖的蛋白激酶调节亚单位2b, cAMP应答元件结合蛋白, 钾离子通道KQT样超家族成员3; 细胞转录翻译相关蛋白, 如核糖体蛋白S26, 真核翻译延伸因子2. 部分表达显著增强的基因(见表1). 表达降低的基因主要有: DEAD/H盒多肽21 DDχ21, 凋亡相关的半胱氨酸蛋白酶胱冬肽酶(cysteine-aspartic acid protease, caspases)4, 核糖核苷酸还原酶M2多肽等. 部分表达显著降低的基因(表2).
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_004661 | 细胞分裂周期蛋白CDC23 | 1.918 |
| 2 | NM_032998 | 含有死亡效应域蛋白DEDD | 1.924 |
| 3 | NM_004612 | 转化生长因子β受体1 | 1.928 |
| 4 | NM_004519 | 钾离子通道KQT样亚家族成员3 KCNQ3 | 1.935 |
| 5 | NM_001066 | 肿瘤坏死因子受体超家族成员1β | 1.946 |
| 6 | NM_002736 | cAMP依赖的蛋白激酶调节亚单位2β | 1.984 |
| 7 | NM_003810 | 肿瘤坏死因子配体超家族成员10 | 1.987 |
| 8 | AF090693 | 凋亡相关的RNA结合蛋白 | 2.004 |
| 9 | NM_004904 | CAMP应答元件结合蛋白CRE-BPa | 2.028 |
| 10 | NM_053274 | FKBP相关蛋白FAP48 | 2.114 |
| 11 | NM_001029 | 核糖体蛋白S26 | 2.165 |
| 12 | NM_00196 | 真核翻译延伸因子2 EEF2 | 3.379 |
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_004728 | DEAD/H盒多肽21-DDχ21 | 0.463 |
| 2 | NM_006754 | 神经囊泡蛋白Synaptophysin相关蛋白SYPL | 0.470 |
| 3 | NM_006415 | 丝氨酸辅酶A转移酶SPTLC1 | 0.472 |
| 4 | NM_006437 | ADP核糖转移酶 | 0.481 |
| 5 | NM_002624 | 预折叠蛋白PFDN5 | 0.513 |
| 6 | NM_002064 | 巯基转移酶GLRX | 0.522 |
| 7 | M73547 | 家族性息肉基因DP1 | 0.535 |
| 8 | NM_033306 | 凋亡相关的半胱氨酸蛋白酶caspase 4 | 0.537 |
| 10 | NM_001034 | 核糖核苷酸还原酶M2多肽RRM2 | 0.549 |
HCV基因组含有单一的开放读码框架, 编码3 010-3 033个氨基酸残基的多肽前体, 两侧是5'-非翻译区及3'-非翻译区, 多肽前体至少被加工为10种结构蛋白和非结构蛋白[9]. 临床和实验研究显示, HCV 核心蛋白, 非结构蛋白3(NS3)、非结构蛋白5A(NS5A)具有多种调控细胞、病毒基因表达、细胞生长以及免疫调节等功能, 分别在HCV感染及致肝细胞癌(HCC)发生发展过程中扮演重要的角色[10-12]. 而HCV 非结构蛋白4B(NS4B)的功能至今为止尚不明确, 研究发现其定位于宿主细胞内质网, 通过"翻译阻断"的方式抑制宿主细胞蛋白的合成, 反之病毒本身的蛋白大量翻译, 进而有助于HCV病毒在宿主细胞内的感染及存活. 研究发现, HCV NS4B蛋白仅存在于细胞质中, 特别是在细胞核周围区域[13-16]. 令人感兴趣的是, HCV NS4B蛋白与Ha-ras癌基因协同作用能够使NIH 3T3细胞发生恶性转化, 表现为细胞间缺乏接触抑制, 有形态学改变和独立生长, 表明HCV NS4B蛋白在肿瘤形成中具有一定功能[17]. NS4B蛋白还可与HCV其他非结构蛋白一起构成复制复合体, 并且与NS5A、NS5B、NS4A等有相互作用, 调节HCV在体内的复制[18-21]. 可见HCV NS4B蛋白是一种多功能的蛋白质, 研究其基因转染细胞后表达的NS4B蛋白对肝细胞基因表达谱的影响, 对于深入分析该蛋白在肝细胞中的生物行为及在慢性HCV感染、肝细胞癌发生过程中的作用具有重要意义.
传统的Northern blot杂交或点杂交方法是以电泳为基础的方法, 一次只能研究一条或几条基因的表达, 其研究效率, 尤其是研究基因群之间相互作用和调控关系等方面受到极大的限制. 基因表达谱芯片技术是将大量的基因特异的探针或其cDNA片段固定在一块基因芯片上, 对来源不同的个体、不同组织、不同细胞周期、不同发育阶段、不同分化阶段、不同病变、不同刺激(包括不同诱导、不同治疗手段)下的细胞内的mRNA或逆转录产物cDNA进行检测, 从而大规模对这些基因表达的个体特异性、组织特异性、发育阶段特异性、分化阶段特异性、病变特异性、刺激特异性进行综合分析和判断[22]. 本研究构建HCV NS4B真核细胞表达载体pcDNA3.1(-)-NS4B, 应用基因表达谱芯片对pcDNA3.1(-)-NS4B转染的HepG2细胞和以空载体转染的相同细胞系的mRNA进行检测, 通过分析二者之间基因表达的差异信息, 寻找NS4B蛋白表达对肝细胞基因表达谱的影响. 在1 152个基因中筛选出56个差异表达的基因, 从表1、2列出的部分表达变化显著的基因中, 看出这些基因涉及细胞信号传导、细胞凋亡、肿瘤发生、能量代谢等生物过程, 说明HCV NS4B蛋白在肝细胞中的生物行为与细胞增生分化生长调节等密切相关.
分析表达增强的基因, 细胞分裂周期蛋白CDC23与酿酒酵母的CDC23高度同源, 是促进细胞分裂后期复合物(anaphase-promoting complex, APC)的成员, 对于细胞周期由G2期向M期过渡是必不可少的, APC能够催化细胞周期蛋白B与泛素(cyclin B-ubiquitin)形成共轭复合物, 在泛素介导的细胞周期蛋白B的蛋白水解过程中起重要作用, HCV NS4B通过上调CDC23的表达对细胞周期有调控作用[23]. 含有死亡效应域蛋白DEDD是细胞程序性死亡通路中共享的蛋白-蛋白相互作用功能域, 该基因过表达预示诱导微弱的凋亡作用, 刺激后, DEDD蛋白从胞质转位至细胞核中, 与细胞核中RNA聚合酶I转录必需的基本转录因子UBTF共同存在.此外, 凋亡相关的RNA结合蛋白也是诱导细胞凋亡的蛋白, HCV NS4B上调两种蛋白的表达, 诱导肝细胞凋亡[24-27]. 转化生长因子b受体I( TGF-bRI)是丝氨酸/苏氨酸激酶膜受体家族的成员, TGF-bRI激酶过表达, 能促进多种信号传导途径的激活, 对于肿瘤细胞的迁移, 以及重要的致瘤事件如Smad2/Smad3的磷酸化是必需的[28-29]. 肿瘤坏死因子(TNF)受体超家族成员1b可与TNF受体1形成杂合物, 介导两种抗凋亡蛋白c-IAP1和c-IAP2的功能, 其中c-IAP1通过TNF受体相关蛋白2 (TNF-receptor-associated factor 2,TRAF2)的泛素化和降解作用而增强TNF诱导的凋亡, 小鼠基因敲除实验也证实该蛋白能够通过刺激抗氧化途径而保护细胞, 防止细胞发生凋亡. 肿瘤坏死因子配体超家族成员10可与其受体结合, 引发MAPK8/JNK、胱冬肽酶8、胱冬肽酶3信号传导通路的激活, 具有选择性的优先诱导转化的细胞和肿瘤细胞凋亡, 而并不杀死正常细胞的特性. HCV NS4B蛋白对上述基因表达有上调作用, 推测其在调控细胞凋亡的过程中处于动态平衡过程[30-32]. 已知cAMP应答元件结合蛋白CRE-BPa是CRE-BP1家族的新成员, 含有推定的金属指状结构和由碱性氨基酸簇和亮氨酸拉链组成的DNA结合功能域, CRE-BPa能特异性的与CRE结合形成同源二聚体, 与转录因子c-JUN或CRE-BP1结合成异源二聚体, 是CRE依赖的转录反式激活因子[33]. 钾离子通道KQT样亚家族成员3 KCNQ3是细胞电信号转导途径中重要的调节因子[34], HCV NS4B蛋白对CRE-BPa及KCNQ3有上调作用, 暗示NS4B蛋白通过正调控细胞信号传导途径, 而促进某些与细胞转录调节密切相关的基因, 如癌基因的转录, 在HCV感染及肿瘤发生中发挥作用. 此外, 核糖体蛋白S26和真核翻译延伸因子2是与细胞转录翻译相关的蛋白, 可见, HCV NS4B对细胞生长调节有一定作用.
分析表达降低的基因, DEAD/H盒多肽DDχ21是推定的重要的RNA解旋酶, 参与众多细胞的RNA二级结构加工过程, 如翻译起始、核糖体RNA合成及加工过程, DDχ21在肿瘤组织中低表达, 而在正常组织中有显著的高水平表达, HCV NS4B对DDχ21的表达有下调作用, 提示, NS4B蛋白在一定程度上可能下调细胞核糖体RNA加工与合成[35]. 预折叠蛋白是辅助其他蛋白正确折叠和有效组装的伴侣蛋白, 该蛋白的第五亚单位蛋白前折叠素5(prefoldin)可与原癌基因c-myc相互结合, 抑制c-myc的转录活性, 被公认为是候选的肿瘤抑制基因. 可见, HCV NS4B蛋白下调肿瘤抑制基因前折叠素5的表达, 间接激活原癌基因c-myc的转录, 推测这可能是NS4B蛋白参与肝细胞恶性转化的重要机制之一[36]. 凋亡相关的半胱氨酸蛋白酶胱冬肽酶4是半胱氨酸-精氨酸蛋白酶家族成员, 胱冬肽酶家族酶的依次活化, 在细胞凋亡的各时相起着至关重要的作用. ADP核糖转移酶蛋白含有催化功能域, 能够催化裂解多聚ADP核糖化反应, 在DNA修复和细胞凋亡过程中起关键作用, HCV NS4B下调胱冬肽酶4和ADP核糖转移酶蛋白的表达, 发挥其对抗细胞凋亡的作用, 这可能是NS4B蛋白参与HCV慢性感染迁延不愈的主要机制之一. 结合表1和表2的结果, 发现HCV NS4B蛋白对细胞凋亡的调节是双向的.
总之, 利用基因表达谱芯片分析了HCV NS4B蛋白对肝细胞基因表达谱的影响, NS4B蛋白能够上调或下调HepG2细胞中许多不同基因的表达, 这些基因与细胞信号传导、细胞增生与分化、细胞凋亡等生物过程密切相关. 本实验结果对于阐明HCV NS4B蛋白在慢性HCV感染及致肝细胞肿瘤发生中的作用具有重要意义.
| 1. | Lee JC, Shih YF, Hsu SP, Chang TY, Chen LH, Hsu JT. Development of a cell-based assay for monitoring specific hepatitis C virus NS3/4A protease activity in mammalian cells. Anal Biochem. 2003;316:162-170. [PubMed] [DOI] |
| 2. | Lundin M, Monné M, Widell A, Von Heijne G, Persson MA. Topology of the membrane-associated hepatitis C virus protein NS4B. J Virol. 2003;77:5428-5438. [PubMed] [DOI] |
| 3. | Florese RH, Nagano-Fujii M, Iwanaga Y, Hidajat R, Hotta H. Inhibition of protein synthesis by the nonstructural proteins NS4A and NS4B of hepatitis C virus. Virus Res. 2002;90:119-131. [PubMed] [DOI] |
| 4. | Piccininni S, Varaklioti A, Nardelli M, Dave B, Raney KD, McCarthy JE. Modulation of the hepatitis C virus RNA-dependent RNA polymerase activity by the non-structural (NS) 3 helicase and the NS4B membrane protein. J Biol Chem. 2002;277:45670-45679. [PubMed] [DOI] |
| 5. | Egger D, Wölk B, Gosert R, Bianchi L, Blum HE, Moradpour D, Bienz K. Expression of hepatitis C virus proteins induces distinct membrane alterations including a candidate viral replication complex. J Virol. 2002;76:5974-5984. [PubMed] [DOI] |
| 6. | Kamiński S. DNA microarrays - a methodological breakthrough in genetics. J Appl Genet. 2002;43:123-130. [PubMed] |
| 7. | Lakhani SR, Ashworth A. Microarray and histopathological analysis of tumours: the future and the past? Nat Rev Cancer. 2001;1:151-157. [PubMed] [DOI] |
| 8. | Borrebaeck CA, Ekström S, Hager AC, Nilsson J, Laurell T, Marko-Varga G. Protein chips based on recombinant antibody fragments: a highly sensitive approach as detected by mass spectrometry. Biotechniques. 2001;30:1126-1130, 1132. [PubMed] |
| 9. | Rosenberg S. Recent advances in the molecular biology of hepatitis C virus. J Mol Biol. 2001;313:451-464. [PubMed] [DOI] |
| 10. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |
| 11. | 刘 妍, 成 军, 牟 劲松, 陆 荫英, 王 建军, 李 克, 王 琳, 张 玲霞. 丙型肝炎病毒非结构蛋白NS3反式激活SV40病毒早期启动子的研究. 解放军医学杂志. 2003;28:44-46. |
| 13. | Kato J, Kato N, Yoshida H, Ono-Nita SK, Shiratori Y, Omata M. Hepatitis C virus NS4A and NS4B proteins suppress translation in vivo. J Med Virol. 2002;66:187-199. [PubMed] [DOI] |
| 14. | Kato N. Molecular virology of hepatitis C virus. Acta Med Okayama. 2001;55:133-159. [PubMed] |
| 15. | Hügle T, Fehrmann F, Bieck E, Kohara M, Kräusslich HG, Rice CM, Blum HE, Moradpour D. The hepatitis C virus nonstructural protein 4B is an integral endoplasmic reticulum membrane protein. Virology. 2001;284:70-81. [PubMed] [DOI] |
| 16. | Kato N, Yoshida H, Ono-Nita SK, Kato J, Goto T, Otsuka M, Lan K, Matsushima K, Shiratori Y, Omata M. Activation of intracellular signaling by hepatitis B and C viruses: C-viral core is the most potent signal inducer. Hepatology. 2000;32:405-412. [PubMed] [DOI] |
| 17. | Park JS, Yang JM, Min MK. Hepatitis C virus nonstructural protein NS4B transforms NIH3T3 cells in cooperation with the Ha-ras oncogene. Biochem Biophys Res Commun. 2000;267:581-587. [PubMed] [DOI] |
| 18. | De Francesco R. Molecular virology of the hepatitis C virus. J Hepatol. 1999;31 Suppl 1:47-53. [PubMed] [DOI] |
| 19. | Neddermann P, Clementi A, De Francesco R. Hyperphosphorylation of the hepatitis C virus NS5A protein requires an active NS3 protease, NS4A, NS4B, and NS5A encoded on the same polyprotein. J Virol. 1999;73:9984-9991. [PubMed] |
| 20. | Kim JE, Song WK, Chung KM, Back SH, Jang SK. Subcellular localization of hepatitis C viral proteins in mammalian cells. Arch Virol. 1999;144:329-343. [PubMed] [DOI] |
| 21. | Koch JO, Bartenschlager R. Modulation of hepatitis C virus NS5A hyperphosphorylation by nonstructural proteins NS3, NS4A, and NS4B. J Virol. 1999;73:7138-7146. [PubMed] |
| 23. | Zhao N, Lai F, Fernald AA, Eisenbart JD, Espinosa R, Wang PW, Le Beau MM. Human CDC23: cDNA cloning, mapping to 5q31, genomic structure, and evaluation as a candidate tumor suppressor gene in myeloid leukemias. Genomics. 1998;53:184-190. [PubMed] [DOI] |
| 24. | Stegh AH, Schickling O, Ehret A, Scaffidi C, Peterhänsel C, Hofmann TG, Grummt I, Krammer PH, Peter ME. DEDD, a novel death effector domain-containing protein, targeted to the nucleolus. EMBO J. 1998;17:5974-5986. [PubMed] [DOI] |
| 25. | Leo CP, Hsu SY, McGee EA, Salanova M, Hsueh AJ. DEFT, a novel death effector domain-containing molecule predominantly expressed in testicular germ cells. Endocrinology. 1998;139:4839-4848. [PubMed] [DOI] |
| 26. | Zhan Y, Hegde R, Srinivasula SM, Fernandes-Alnemri T, Alnemri ES. Death effector domain-containing proteins DEDD and FLAME-3 form nuclear complexes with the TFIIIC102 subunit of human transcription factor IIIC. Cell Death Differ. 2002;9:439-447. [PubMed] [DOI] |
| 27. | Alcivar A, Hu S, Tang J, Yang X. DEDD and DEDD2 associate with caspase-8/10 and signal cell death. Oncogene. 2003;22:291-297. [PubMed] [DOI] |
| 28. | Itoh S, Thorikay M, Kowanetz M, Moustakas A, Itoh F, Heldin CH, ten Dijke P. Elucidation of Smad requirement in transforming growth factor-beta type I receptor-induced responses. J Biol Chem. 2003;278:3751-3761. [PubMed] [DOI] |
| 29. | Bourguignon LY, Singleton PA, Zhu H, Zhou B. Hyaluronan promotes signaling interaction between CD44 and the transforming growth factor beta receptor I in metastatic breast tumor cells. J Biol Chem. 2002;277:39703-39712. [PubMed] [DOI] |
| 30. | Rothe M, Sarma V, Dixit VM, Goeddel DV. TRAF2-mediated activation of NF-kappa B by TNF receptor 2 and CD40. Science. 1995;269:1424-1427. [PubMed] [DOI] |
| 31. | Renshaw SA, Parmar JS, Singleton V, Rowe SJ, Dockrell DH, Dower SK, Bingle CD, Chilvers ER, Whyte MK. Acceleration of human neutrophil apoptosis by TRAIL. J Immunol. 2003;170:1027-1033. [PubMed] [DOI] |
| 32. | Kim YS, Schwabe RF, Qian T, Lemasters JJ, Brenner DA. TRAIL-mediated apoptosis requires NF-kappaB inhibition and the mitochondrial permeability transition in human hepatoma cells. Hepatology. 2002;36:1498-1508. [PubMed] |
| 33. | Nomura N, Zu YL, Maekawa T, Tabata S, Akiyama T, Ishii S. Isolation and characterization of a novel member of the gene family encoding the cAMP response element-binding protein CRE-BP1. J Biol Chem. 1993;268:4259-4266. [PubMed] |
| 34. | Schroeder BC, Kubisch C, Stein V, Jentsch TJ. Moderate loss of function of cyclic-AMP-modulated KCNQ2/KCNQ3 K+ channels causes epilepsy. Nature. 1998;396:687-690. [PubMed] [DOI] |