修回日期: 2003-07-01
接受日期: 2003-07-16
在线出版日期: 2004-01-15
目的: 应用抑制性消减杂交技术(SSH)及生物信息学技术(bioinformatics)筛选并克隆丙型肝炎病毒(HCV)非结构蛋白NS5A反式激活新型靶基因, 进一步阐明HCV感染相关疾病的发病机制.
方法: 以HCV NS5A蛋白表达质粒pcDNA3.1(-)-NS5A转染HepG2细胞, 以空载体pcDNA3.1(-)为平行对照, 提取mRNA并进行抑制性消减杂交分析.应用分子生物学技术, 结合生物信息学技术, 分析并克隆HCV NS5A反式激活作用的新的靶基因.
结果: 对于所获基因片段序列分析表明, 其中之一为新型基因片段, 从转染pcDNA3.1(-)-NS5A的HepG2细胞提取总RNA, 以逆转录多聚酶链反应(RT-PCR)技术扩增获得该新基因的全长序列, 并测序证实, 命名为NS5ATP1.NS5ATP1基因的编码序列全长为1011个核苷酸(nt), 编码产物由336个氨基酸残基(aa)组成.
结论: HCV NS5ATP1是一种典型的病毒基因组编码的具有反式激活作用的蛋白, 而SSH是一种鉴定、分离组织细胞中选择性表达基因的技术.通过这种技术, 发现了HCV NS5ATP1反式激活作用的新的靶基因, 这一发现, 为进一步研究HCV NS5ATP1蛋白反式激活作用的分子生物学机制和探索新型治疗技术奠定了基础.
引文著录: 刘敏, 成军, 王琳, 张树林, 邵清, 张健, 梁耀东. 丙型肝炎病毒非结构蛋白NS5A反式激活基因1的克隆. 世界华人消化杂志 2004; 12(1): 78-81
Revised: July 1, 2003
Accepted: July 16, 2003
Published online: January 15, 2004
AIM: To screen and clone the target genes transactivated by hepatitis C virus (HCV) nonstructural protein 5A (NS5A) and to pave the way for elucidating the pathogenesis of HCV infection.
METHODS: Suppression subtractive hybridization (SSH) technique and bioinformatics were used. mRNA from HepG2 cells transfected pcDNA3.1(-)-NS5A and pcDNA3.1(-) empty vector, respectively, was isolated, and SSH method was employed to analyze the differentially expressed DNA sequence between the two groups. The coding gene transactivated by HCV NS5A was cloned by bioinformatics methods.
RESULTS: The obtained sequences were searched for homologous DNA sequence from GenBank, one of which was a new gene with unknown function. The new gene with no homology with known genes in this database was confirmed and electric polymerase chain reaction was conducted for the cloning of the full-length DNA for the new gene and in conjunction with Kozak role and the exist of polyadenyl signal sequence. The reverse transcription PCR (RT-PCR) was used to amplify the new gene, named as NS5ATP1, from the mRNA of HepG2 cells transfected.
CONCLUSION: HCV NS5A is a potential transactivator. Suppression subtractive hybridization is an efficient and convenient method for identification and separation of expressed genes in hepatocytes. These results pave the way for the study of the molecular mechanism of the transactivating effects of HCV NS5A protein and the development of new therapy for chronic hepatitis C.
- Citation: Liu M, Cheng J, Wang L, Zhang SL, Shao Q, Zhang J, Liang YD. Cloning and identification of human gene 1 transactivated by nonstructural protein 5A of hepatitis C virus. Shijie Huaren Xiaohua Zazhi 2004; 12(1): 78-81
- URL: https://www.wjgnet.com/1009-3079/full/v12/i1/78.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i1.78
丙型肝炎病毒(HCV)是一种单股正链RNA病毒, 基因组含有惟一的开放读框(ORF), 可以分成结构基因区和非结构基因区[1-4]. NS5A是非结构蛋白的一种, 哺乳动物细胞表达的NS5A蛋白以两种形式存在, 分子量分别为56 KD和58 KD. HCV NS5A是HCV基因组编码的一种重要的具有多种生物学活性的非结构蛋白质, 在HCV多蛋白的成熟和RNA的复制过程中具有十分重要的作用[5-7]. NS5A还是一种作用很强转录激活因子, 调控着细胞内许多病毒及细胞基因的转录, 与感染HCV细胞发生恶性转化的过程有关[8,9]. 我们利用抑制性消减杂交技术(suppression subtractive hybridization, SSH)及生物信息学技术(bioinformatics)筛选并克隆了NS5A蛋白反式激活作用的新靶基因, 命名为丙型肝炎病毒NS5A蛋白反式激活基因1(NS5ATP1), 为HCV NS5A蛋白反式激活作用及HCV相关疾病发病机制的研究奠定了基础.
HepG2细胞及感受态大肠杆菌JM109为本室保存, pcDNA3.1(-)真核表达载体购自Invitrogen公司; Lipofectamine PLUS 转染试剂购自Gibco公司, mRNA Purification试剂盒购自Amersham Pharmacia Biotech, PCR-Select cDNA Subtraction试剂盒, 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒购自Clontech, High Pure PCR Product Purification试剂盒购自Boehringer Mannheim公司, T7、SP6通用引物及pGEM-Teasy载体购自Promega公司.丙型肝炎病毒非结构蛋白真核表达质粒pcDNA3.1(-)-NS5A由本室构建.DNA测序由上海博亚公司完成.
1.2.1 消减杂交文库的建立及克隆分析: 分别将pcDNA3.1(-)-NS5A及pcDNA3.1 (-)空载体转染HepG2细胞, 48 h后提取mRNA, 以此mRNA为模板逆转录合成双链cDNA(dscDNA), 并分别标记为Tester和Driver, 经RsaI(一种识别4碱基序列的内切酶)消化后将Tester cDNA分为两份, 分别连接特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 与过量的Driver cDNA进行2次消减杂交和2次抑制性PCR, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增. 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(200-1 000 bp), 测序. 对于每一条基因片段的序列提交GenBank进行同源基因序列的搜索, 确认所得基因片段的独特性, 然后依据GenBank数据库中的同源基因片段进行电子拼接, 根据Kozak规则和终止密码子下游的多聚腺苷酸信号序列, 确定完整的编码基因序列.
1.2.2 新基因的PCR扩增与序列分析: 根据电子拼接的新基因序列(1 011 bp), 利用生物软件Vector NTI设计新基因的序列特异性的含有特异性核酸内切酶(EcoRⅠ/BamHⅠ)的引物, 利用转染了pcDNA3.1 (-)-NS5A的HepG2细胞来源的mRNA, 经过RT-PCR扩增, 并与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含有氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 使用碱裂解法提质粒后进行双酶切(EcoRⅠ/BamHⅠ)鉴定, 证明目的基因约1 011 bp后测序, 进一步鉴定正确后获得阳性克隆.
纯化高质量的mRNA是获得 cDNA高产量的前提. 紫外分光检测显示, 转染了NS5A表达质粒及空载体的HepG2 细胞mRNA分别为 4.64 g和4.38 g, A260/A280= 1.97. 10 g/L琼脂糖凝胶电泳见 mRNA为大于0.5 kb清晰慧尾片状条带, 证实获得高质量mRNA.
dscDNA两端连接效率检测结果显示两组dscDNA已与接头高效率连接. 消减效率的鉴定结果显示: 与未消减组PCR产物相比, 消减组PCR产物中看家基因甘油三磷酸脱氢酶(G3PDH)基因产物大大减少, 说明所构建的消减文库具有很高的消减效率. 对差异表达基因片段进行菌落PCR扩增, 结果显示为200-1 000 bp大小不等的插入片段. 挑选克隆测序并与GenBank数据库进行初步比较.
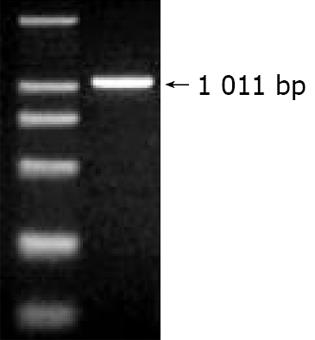
利用美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)及其同源基因序列的搜索(BLASTN), 发现其中的1个克隆序列与GenBank中注册的已知功能基因序列没有同源性. 电子拼接推定该基因的开放读码框架, 设计引物, 从表达HCV NS3蛋白的HepG2细胞提取总RNA后, 进行RT-PCR, 扩增出约1 011 bp片段(图1), 经过测序克隆出全长基因片段, 获得HCV NS5A反式激活作用的新型靶基因, 命名为NS5ATP1. 新基因的开放读码框架(ORF)长度为1 011个核苷酸(nt), 编码产物由336个氨基酸残基(aa)组成(图2).
HCV感染是引起慢性肝炎的主要原因, 与肝纤维化及肝细胞癌(HCC)的发生密切相关, 其中病毒编码的NS5A蛋白起着重要的作用. NS5A是反式转录激活因子, 其羧基末端富含酸性氨基酸及脯氨酸, 这是真核细胞转录因子特有的结构特征, 但是其参与细胞转录调节的机制仍不十分清楚[9-11]. 一般来说, 反式转录激活因子是在细胞核中起作用的. 尽管已有明确证据显示NS5A蛋白全长定位于细胞内质网, 但在细胞核内仍可以检测到NS5A的N-末端定位信号[12-16], 提示NS5A的部分结构可进入细胞核内并成为转录激活因子的一部分. 缺失N-末端146 aa和DNA结合区域在GAL4(一种酵母细胞转录激活因子)的HCV NS5A蛋白可强烈的激活酵母和哺乳动物细胞的转录活性. 结合到GAL4 DNA结合区域的全长HCV NS5A蛋白并没有激活转录的作用, NS5A片段转录激活作用最强的位点定位于2 135和2 331 aa之间. NS5A的转录激活区域包括两个酸性氨基酸区(2 143-2 184 aa, 2 220-2 273 aa)和一个脯氨酸富集区(2282-2327 aa).酸性氨基酸区的酸性氨基酸残基的数量与NS5A的反式激活活性相关, 2个酸性氨基酸区是NS5A的转录激活作用的核心区域, 因此酸性氨基酸区2的一些氨基酸的突变可明显影响NS5A的转录激活作用, 但是这些突变更多的是引起蛋白二级结构的改变. 脯氨酸富集区被认为对NS5A的转录激活作用只有一定的加强作用, 相对而言并不特别重要.
Gong et al[17]研究发现, NS5A能够反式激活核转录因子NF-B及STAT3, 在细胞炎症反应、肿瘤发生及转移过程中起重要作用. NS5A在细胞质内通过氧化应激作用激活NF-B和STAT-3转录因子. NS5A引起细胞内钙离子的紊乱.Ca2+作为第二信使激发线粒体内的活性氧簇水平, 使NF-B和STAT-3易位入细胞核内. 有证据表明STAT-3的激活部分有NS5A的作用. 在抗氧化剂N-乙酰-L-半胱氨酸(NAC)或者Ca2+拮抗剂(EGTA-AM, TMB-8)作用下, NS5A诱导的NF-B和STAT-3的反式激活作用消失. 这些结果说明NS5A可引起细胞内与病毒感染相关的病理改变的发病机制. Ghosh et al[18]研究发现, NS5A蛋白能够抑制细胞周期调节基因p21WAF1, 激活人肝癌细胞中增生细胞核抗原(PCNA)基因, 从而调节细胞凋亡, 促进细胞增生. NS5A cDNA能够使转染的小鼠成纤维细胞NIH 3T3具有转化特性, 且转化细胞移植入裸鼠体内可形成纤维肉瘤灶, 直接证明了HCV NS5A蛋白的恶性转化潜能.由于NS5A表现出对抗干扰素α(IFNα)的治疗效应而引起人们广泛的关注, NS5A能够与肝细胞中的IFN刺激蛋白-双链RNA依赖的激酶(PKR)相互作用, 抑制PKR的功能, 从而下调IFNα刺激的抗病毒效应[19].
本项研究中发现了HCV NS5A蛋白反式激活的新的靶基因, 为研究HCV NS5A蛋白的反式激活作用及其后果提供了新的研究方向和思路. 由于目前对于新克隆的NS5A蛋白反式激活的靶基因的结构与功能、表达与调控, 以及新基因的生物学作用和在丙型肝炎致病机制中的作用和地位不明, 需要进一步研究, 以阐明新基因的研究意义.
编辑: N/A
| 4. | Cheng J. Molecular pathogenesis of viral hepatitis. J Gastroenterol Hepatol. 2002;16:A185. |
| 5. | 成 军. 丙型肝炎病毒干扰素敏感决定区的研究进展. 国外医学. 流行病学传染病学分册. 2000;27:55-58. |
| 6. | 钟 彦伟, 成 军, 陈 新华, 王 刚, 洪 源, 王 琳, 李 莉, 张 玲霞, 陈 菊梅. 应用噬菌体表面展示技术筛选丙型肝炎病毒NS5A抗原模拟表位. 世界华人消化杂志. 2002;10:133-136. [DOI] |
| 7. | 成 军, 陈 菊梅, 钟 彦伟, 施 双双, 倪 勤, 刘 妍, 王 刚, 董 菁, 夏 小兵, 刘 友昭. HCV非结构蛋白NS5A人源单链可变区抗体基因的筛选与鉴定. 中华实验与临床病毒学杂志. 2001;15:216-218. [DOI] |
| 9. | Kato N, Lan KH, Ono-Nita SK, Shiratori Y, Omata M. Hepatitis C virus nonstructural region 5A protein is a potent transcriptional activator. J Virol. 1997;71:8856-8859. [PubMed] |
| 10. | Kato N, Yoshida H, Kioko Ono-Nita S, Kato J, Goto T, Otsuka M, Lan K, Matsushima K, Shiratori Y, Omata M. Activation of intracellular signaling by hepatitis B and C viruses: C-viral core is the most potent signal inducer. Hepatology. 2000;32:405-412. [PubMed] [DOI] |
| 11. | Chung KM, Song OK, Jang SK. Hepatitis C virus nonstructural protein 5A contains potential transcriptional activator domains. Mol Cells. 1997;7:661-667. [PubMed] |
| 12. | Ghosh AK, Steele R, Meyer K, Ray R, Ray RB. Hepatitis C virus NS5A protein modulates cell cycle regulatory genes and promotes cell growth. J Gen Virol. 1999;80:1179-1183. [DOI] |
| 13. | No authors listed. Post-transfusion hepatitis leading to chronic hepatitis. Gastroenterology. 1978;75:732-741. |
| 14. | Polyak SJ, Khabar KS, Paschal DM, Ezelle HJ, Duverlie G, Barber GN, Levy DE, Mukaida N, Gretch DR. Hepatitis C virus nonstructural 5A protein induces interleukin-8, leading to partial inhibition of the interferon-induced antiviral response. J Virol. 2001;75:6095-6106. [PubMed] [DOI] |
| 15. | Tan SL, Katze MG. How hepatitis C virus counteracts the interferon response: the jury is still out on NS5A. Virology. 2001;284:1-12. [PubMed] [DOI] |
| 16. | Sato C. Effects of hepatitis C virus proteins on the interferon-stimulated signal transduction. Nippon Rinsho. 2001;59:1271-1276. [PubMed] |
| 17. | Gong G, Waris G, Tanveer R, Siddiqui A. Human hepatitis C virus NS5A protein alters intracellular calcium levels, induces oxidative stress, and activates STAT-3 and NF-B. Proc Natl Acad Sci USA. 2001;98:9599-9604. [PubMed] [DOI] |
| 18. | Ghosh AK, Steele R, Meyer K, Ray R, Ray RB. Hepatitis C virus NS5A protein modulates cell cycle regulatory genes and promotes cell growth. J Gen Virol. 1999;80:1179-1183. [PubMed] [DOI] |