修回日期: 2002-10-10
接受日期: 2002-10-17
在线出版日期: 2003-05-15
研究食管、贲门癌细胞染色体异常及意义.
16例食管癌, 3例贲门癌采用比较基因组杂交技术, 以不同荧光染料分别标记正常人胎盘DNA和癌细胞DNA, 与人中期染色体杂交, 用荧光显微镜观察摄取图像, 通过专用分析软件, 确定染色体DNA的扩增和缺失.
食管、贲门癌同一患者可出现多条染色体扩增和缺失, 可出现整条染色体或长臂或短臂或某一染色体上DNA位点的扩增或缺失, 常见扩增有3q, 17q, 20q, 8q, 9q, 11q, 5p, 17p; 缺失为4q, 4p, 9q, 18q; 6例有8q扩增者, 病变均侵犯食管壁全层、邻近组织且伴淋巴结转移.染色体DNA扩增数目最多的见于Ⅱ期患者, Ⅰ期最少.
食管、贲门癌细胞染色体DNA扩增和缺失与病变的发生、发展、转移和分期有关.
引文著录: 武珊珊, 刘吉福, 王明荣. 食管、贲门癌染色体异常分析及意义. 世界华人消化杂志 2003; 11(5): 681-684
Revised: October 10, 2002
Accepted: October 17, 2002
Published online: May 15, 2003
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2003; 11(5): 681-684
- URL: https://www.wjgnet.com/1009-3079/full/v11/i5/681.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v11.i5.681
食管癌是常见恶性肿瘤, 是癌症死亡的常见原因. 研究其发生、发展和影响治疗的因素是提高远期生存率的重要环节. 从染色体和基因方面进行研究是获取上述资料的关键途径. 1999-08/2000-05收集了19例食管、贲门标本, 用比较基因组杂交技术研究食管、贲门癌细胞染色体异常及意义, 报道如下.
手术切除食管癌16例、贲门癌3例; 其中男10例, 女9例. 避开切除肿瘤的坏死区, 取部分肿瘤组织, 立即保存于-170 ℃液氮, 备用. 标本全部取自北京军区总医院胸外科. 食管、贲门癌标本的临床资料见(表1).
| 序号 | 性别 | 年龄(岁) | 病变部位 | 病理类型 | 分化程度 | TNM分类 | 分期 |
| 1 | 女 | 63 | 食管中段癌 | 鳞癌 | 中分化 | T2N1M0 | Ⅱb |
| 2 | 男 | 63 | 食管中段癌 | 鳞癌 | 中高分化 | T3N1M0 | Ⅲ |
| 3 | 女 | 51 | 食管 | 鳞癌 | 中分化 | T4N1M1 | Ⅳ |
| 4 | 女 | 58 | 食管 | 鳞癌 | 高分化 | T2N0M0 | Ⅱa |
| 5 | 男 | 51 | 食管 | 鳞癌 | 高分化 | T2N0M0 | Ⅱa |
| 6 | 女 | 74 | 食管下段癌 | 鳞癌 | 低分化 | T3N0M0 | Ⅱb |
| 7 | 男 | 62 | 食管中下段癌 | 鳞癌 | 高分化 | T3N0M0 | Ⅱb |
| 8 | 女 | 73 | 食管癌 | 鳞癌 | 低分化 | T3N1M0 | Ⅲ |
| 9 | 男 | 64 | 食管癌 | 鳞癌 | 高分化 | T3N0M0 | Ⅱb |
| 10 | 男 | 51 | 食管癌 | 鳞癌 | 中高分化 | T3N1M0 | Ⅲ |
| 11 | 男 | 50 | 食管中段癌 | 鳞癌 | 低分化 | T4N1 M0 | Ⅳ |
| 12 | 女 | 64 | 食管癌 | 鳞癌 | 低分化 | T4N1 M0 | Ⅳ |
| 13 | 女 | 58 | 食管癌 | 鳞癌 | 中高分化 | T1 N0 M0 | Ⅰ |
| 14 | 女 | 55 | 食管上段癌 | 鳞癌 | 未分化 | T3N1M0 | Ⅲ |
| 15 | 男 | 55 | 食管中段癌 | 鳞癌 | 中高分化 | T3N1M0 | Ⅲ |
| 16 | 男 | 76 | 食管中段癌 | 鳞癌 | 中低分化 | T1NX M0 | Ⅰ |
| 17 | 男 | 77 | 贲门 | 腺鳞 | 中低分化 | T3N0M0 | Ⅱb |
| 18 | 男 | 50 | 贲门 | 腺癌 | 低分化 | T3N1M0 | Ⅲ |
| 19 | 女 | 68 | 贲门 | 腺癌 | 中低分化 | T3N1M0 | Ⅲ |
1.2.1 DNA提取 按分子生物学方法常规提取组织基因组DNA.
1.2.2 比较基因组杂交(CGH) (1)探针标记: 采用切口平移法, 用Biotin-16-dUTP标记肿瘤DNA, 用Digoxin-11-dUTP标记参照(胎盘组织)DNA, 标记探针最后产物大小在500-1200 bp. (2)探针沉淀: 等量的肿瘤探针和正常参照探针混合(500 ng/每管), 加Cot-1 DNA、醋酸钠、乙醇沉淀, 离心. 沉淀的DNA用5 μL杂交液溶解(去离子甲酰胺、40%硫酸葡聚糖和双蒸水), 室温放置20 min. (3)用标准方法制备正常外周血淋巴细胞中期染色体片. (4)选择中期染色体分布的最佳部位, 用RNase、胃蛋白酶预处理. 1%多聚甲酰胺固定, 梯度酒精脱水, 干燥. (5)杂交: (a)探针变性: 杂交混合液在75 ℃水浴变性8 min, 至37 ℃预复性20 min. (b)中期染色体片变性: 将已预处理的中期染色体片置70%去离子甲酰胺2XSSC中, 73 ℃变性2 min. 在预冷的2XSSC中洗涤2次, 每次5 min. 梯度酒精脱水. (6)杂交及封片: 将预复性的杂交混合液滴在处理好的中期染色体片标记的部位, 用盖玻片封片, 37 ℃, 60-72 h.杂交后用50% 甲酰胺2XSSC, 43 ℃, 15 min. 2XSSCX2洗涤. (7)杂交后标记检测: 肿瘤探针的杂交信号用FITC-avidin和抗- avidin测定; 正常参照(胎盘组织)探针用鼠源性antidigoxin-TRITC 和抗-鼠抗体测定; 染色体用4, 6-二氨基-2-苯吲哚(DAPI)复染.
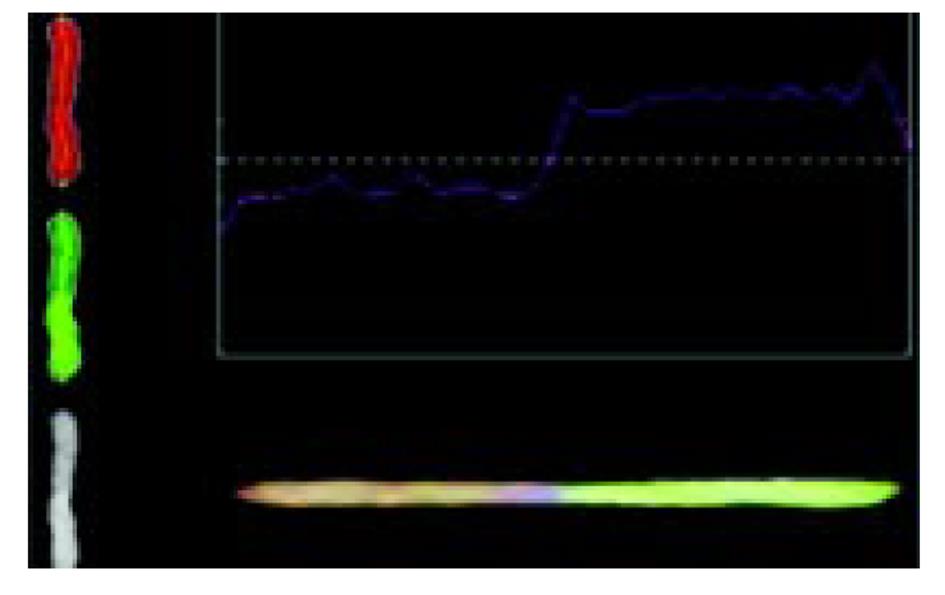
1.2.3 CGH图像的获取和分析 (1)图像通过荧光显微镜, 63倍物镜观察, 荧光信号通过冷电荷藕荷设备(CCD)相机(prinston inc)摄取, 连入计算机; 分别摄取对应DAPI、TRITC、FITC3种荧光染料的 3个黑白数字图像, 这3种染料分别分配蓝、红、绿三原色, 由此合成了彩图. (2)CGH图像分析: 应用CGH analyzer软件(由清华大学编制), 分别计算每条染色体的中轴荧光信号比值, 红、绿荧光的比值相等处定为零, 阈值分别定为1.2(绿)和0.8(红), 超过阈值处分别定义为扩增或缺失(图1).
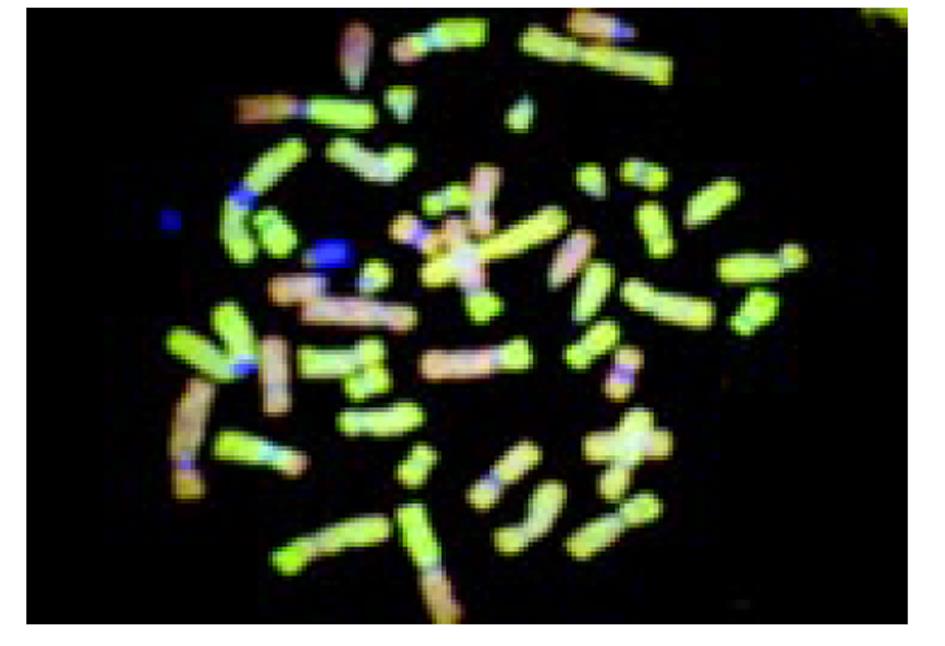
16例食管癌和3例贲门癌均被发现有染色体的扩增; 染色体缺失在3例贲门癌均存在, 食管癌有13例(表2); 常见食管癌染色体异常频率(表3). CCD拍摄的原始中期染色体照片(图2); 染色体的红绿两色荧光的比例剖面图(图3).
| 序号 | 期、级 | DNA扩增位点 | DNA缺失位点 |
| 1 | Ⅱ | 3q27, 5p15, 7p1-13, 9q21, 11, 17, 20q12, 21q21-222, xp21 | 1p13-31, 4q35, 18q2, xp22 |
| 2 | Ⅲ | 1q, 1p1-21, 2q, 3q | 1p32-pter |
| 3 | Ⅳ | 1q, 2q | |
| 4 | Ⅱ | 5p, 8q, 11q13, 20q | 5q13-qter, 6q, 8p2, 9, 18q2, xp |
| 5 | Ⅱ | 5, 8q12-13, 9q, 11q, 14q, 19q11-12, 20q11 | 4, 22, x |
| 6 | Ⅱ | 1q32-qter, 2q1, 3q27, 6p21-22, 6q15-22, 7q21, 11q13, 14q, 16, 20q12, xp21 | 3p3q1, 4q35, 5p, 6p23-pter, 6q27 |
| 7 | Ⅱ | 2p22-23, 5p13-15.2, 7p13-15, 7q11.2, 19, 21q, 17 | 4, x |
| 8 | Ⅲ | 2p16, 2q32, 3q27, 5p, 8q, 11q13, 13q, 15q22-qter, 17, 21, xp22 | 2p25, 8p, 15q12-21, 18q21-qter |
| 9 | Ⅱ | 1p31-pter, 3q, 4p, 9q34, 12, 13, 15q26, 17, 18p | x |
| 10 | Ⅲ | 3q21-27, 4p15-q32, 8q12-241 | 1p35, 7q3, 21 |
| 11 | Ⅳ | 1p2, 3q | |
| 12 | Ⅳ | 3q1, 3q24-qter, 5q31, 8q, 10q21, 15q2 19q11-12 | 1q32, 4p15-pter, 9, 10p, xp22 |
| 13 | Ⅰ | 1q, 3q, 13q, 7, 8 | 19 |
| 14 | Ⅲ | 1p, 3q, 5p31-pter, 7p15-21, 9p11-13, 9q, 11, 13, 17, 18, 19, 20, 21q | 4, 5q, 9p22-pter, 12, 14, 15, 16, x |
| 15 | Ⅲ | 3q, 13q, 17 | |
| 16 | Ⅰ | 1q22, 3q, 5p, 9 | |
| 17 | Ⅱ | 2q, 3, 16q, 20 | 5p, 12, 21, 19, xp22 |
| 18 | Ⅲ | 3q, 4q3-qter, 6, 8p25-pter, 11p, 18q, 20q1, 21q | 11 |
| 19 | Ⅲ | 1p, 9, 13, 15 | 1q |
| 区域 | 扩增频率 | 区域 | 缺失频率 |
| 频率(n = 16) | 频率(n = 16) | ||
| 3q | 12 | 4q | 5 |
| 3q27 | 12 | 4q35 | 5 |
| 5p | 7 | 4p | 4 |
| 5p15 | 6 | 9p | 2 |
| 8q | 5 | 9p22-pter | 3 |
| 8q12 | 6 | 18q | 3 |
| 9q | 5 | 18q2 | 3 |
| 11q | 6 | Xp22 | 7 |
| 11q13 | 6 | ||
| 17q | 7 | ||
| 17p | 6 | ||
| 20q | 5 | ||
| 20q12 | 5 |
DNA拷贝数出现明显扩增的常见位点为3q(12例, 75%), 17q, 5p(7例, 43.7%), 8q, 11q, 17p(6例, 37.5%), 9q, 20q(5/16例, 31.3%). DNA拷贝数减少的位点在4q, 4p, 9q, 18q, xp. DNA拷贝数的扩增和缺失可发生于整条染色体或染色体的一条长臂或短臂, 也可以发生于染色体DNA位点, 并且以后者更为多见.
食管癌染色体DNA拷贝数增加最多的发生在Ⅱ期的病例. 本组6例中有5例(83.8%), Ⅲ、Ⅳ期8例患者仅有3例(37.5%), 2例Ⅰ期患者染色体扩增数目均少. DNA缺失也以Ⅱ期最多见, 3/16例. 贲门癌DNA拷贝数扩增数多的仅有1例, 以整条染色体扩增或缺失最常见. 染色体扩增或缺失的位点分布在一窄区域内, 食管癌在3q27, 20q11-12, 8q12-13, 8q12-24.1, 5p15扩增; 缺失在18q2, 4q35, 3/16例, X染色体DNA缺失本组食管癌有7例, 贲门癌1例. 6例有8q扩增的患者, 其中有2例T3N1M0, 1例T4N1M0.
研究食管癌细胞染色体异常, 包括染色体DNA位点扩增或缺失, 是深入了解食管癌发生、发展、转移和对化疗反应的重要途径. 然而, 食管癌染色体异常改变的复杂性, 也增加了此研究的难度.用先进的比较基因组杂交技术(comparative genomic hybridization, CGH), 检测肿瘤细胞染色体扩增或/和缺失的部位及基因位点, 以分析其临床意义, 目前国内尚未见报道.
CGH是一项快速可靠的分子细胞遗传学的检测技术, 基本原理是用不同的荧光染料分别标记正常基因组DNA和肿瘤细胞DNA, 然后与正常人中期染色体杂交, 通过检测染色体上两种荧光信号的相对强度比率, 了解肿瘤细胞DNA拷贝数的改变, 并能同时进行染色体定位. 我们利用这一技术检测了19例食管、贲门癌患者, 结果表明食管癌患者染色体扩增和缺失涉及多条染色体, 多位点改变, 更说明了食管癌遗传学的发生、发展是极其复杂的, 与临床上观察到的肿瘤增长速度、转移和预后有较密切联系[1,2]. 食管癌染色体异常的复杂性可表现为同一患者有多条染色体DNA扩增和缺失, 3q扩增最多见. 本组与文献报道的多种肿瘤检测结果相似[3-5]. 目前已有研究表明3q区域有原癌基因包括上皮细胞转化序列2(epithelial cell transforming sequence2, ECT2); 截短的P63变形体和易突变基因甲基化CPG结合结构域蛋白4, 这些基因在肿瘤的发生过程中有重要作用, 这可能是多数肿瘤3q区域出现高频扩增的原因.
致癌基因的扩增是食管癌的重要特征: Tada et al[6]用比较基因组方法检查了36例食管鳞癌标本, 分析了鳞癌细胞扩增、过表达及缺失情况. 以3q(75%), 8q23-qter(50%), 11q13(44%)出现扩增频率最高. his1, cyclin-D(bcl-1)定位于11q13位点, 表明与癌细胞分化有关. 结果表明11q13-qter扩增预示淋巴结转移. 本组食管癌11q染色体扩增有6例占31.6%, 而分化好的仅有2例, 分化差的有4例; 临床观察到11q位点扩增多表现为分化差、恶性度高. myc基因定位于8q, 该基因在食管癌中常发现有扩增, 间质金属蛋白酶(MMPS)也定位于8q, 他能分解细胞外基质的多种内肽酶, 该基因与肿瘤侵袭过程有关. 本组8q扩增患者食管癌有6例(占37%), 其中有3例患者病变侵犯深, 伴淋巴结转移.
抑癌基因的缺失是食管癌的另一特征, APC是已知的抑癌基因[7-9], 定位于5q21位点; 而DCC为1个促进细胞凋亡的基因, 定位于18q染色体[10]. 食管切除标本中, Barrett食管增生, 间变到癌变化中, APC等位基因缺失或/和APC蛋白表达缺失较MCC基因发生更早, 在不同阶段发生的比率上升, 表明这2个基因在癌发生发展中的作用. 抑癌基因的缺失促进了癌的发生. 本组食管癌中18q缺失有3例(3/16例, 占18.8%).
食管癌染色体DNA拷贝数的增加与病理分期有关. 食管癌的DNA倍体数与癌的病理分期的关系已有报道. Watanabe et al[11]报道78例食管鳞癌中DNA高度单倍体(high grade aneuploid, HGA)多见于晚期癌(占55%), 而DNA二倍体则多见于早期癌或表浅癌. 食管鳞癌DNA单倍体基因改变率为73.3%, 而二倍体基因改变率仅20.0%[11]. 有关胃肠实体瘤DNA倍体数的变化已进行研究[12]. 最近Varis et al[13]用比较基因组杂交方法, 比较胃腺癌与食管鳞癌DNA倍体变化最常见的扩增均在20q(50%), 17q(39%); 本组患者染色体扩增20q占36.8%(11/17例), 17q为31.6%(6/17例). 另有报道[14,15]食管癌发生转移者, 6q, 9q杂合性缺失显著. 本组中缺失位于6q, 9q者, 在Ⅱ-Ⅳ期中占17.7%(3/17例), 表明这两个位点缺失与癌转移有关. 本组癌细胞染色体DNA扩增数目以Ⅱ期最多, 其次是Ⅲ, Ⅳ, Ⅰ期最少, 这种变化与癌细胞倍体变化应有一定联系.
癌细胞染色体DNA扩增的区域有相近之处. 有报道食管原发腺癌以4q, 14q缺失, 2p, 17p扩增常见, 而且在染色体窄区域内出现扩增. 本组在8q, 5p扩增, 18q, 4q缺失均出现, 均显示在染色体窄区域, 其意义有待进一步探讨. 癌细胞染色体DNA中癌基因扩增在肿瘤的发生和发展中具有重要作用, 多种致癌基因扩增, 表明食管、贲门癌是多种癌基因联合作用的结果; 同时抑癌基因的缺失促进肿瘤的发展. D'Cruz et al[16]研究发现, 乳腺癌当c-myc基因存在, 又通过激活ras基因突变, 促进肿瘤进展; 反之消除c-myc基因的诱导作用, 无ras基因突变, 则乳腺增生或癌病变消退, 这进一步证明癌的发生、发展是由多种癌基因协同作用的结果; 而在食管癌中, 各种癌基因协同作用的方式、途径及影响因素, 则有待进一步研究. X染色体DNA缺失与获得性抗药性有关, 但是与遗传因素的关系有待探讨.
| 1. | Takashi U, Akira T, Shigefumi Y, Toshihiro A, Hiroaki T, Tomoko F, Shigeto K, Atsunori O, Masaaki O, Kohsuke S. Gain of 5p15 detected by comparative genomic hybridization as an independent marker of poor prognosis in patients with esophageal squamous cell carcinoma. Clin Cancer Res. 2002;8:526-533. |
| 2. | Yen CC, Chen YJ, Chen JT, Hsia JY, Chen PM, Liu JH, Fan F S, Chiou TJ, Wang WS, Lin CH. Comparative genomic hybridization of esophageal squamous cell carcinoma: correlations between chromosomal aberrations and disease progression/prognosis. Cancer. 2001;92:2769-2777. [DOI] |
| 3. | Petersen I, Langreck H, Wolf G, Schwendel A, Psille R, Vogt P, Reichel MB, Ried T, Dietel M. Small-cell lung cancer is characterized by a high incidence of deletions on chromosomes 3q, 4q, 5q, 10q, 13q and17. Br J Cancer. 1997;75:79-86. [DOI] |
| 4. | El Rifai W, Frierson HF Jr, Moskaluk CA, Harper JC, Petroni GR, Bissonette EA, Jones DR, Knuutila S, Powell SM. Genetic differences between adenocarcinomas arising in Barrett esophagus and gastric mucosa. Gastroenterology. 2001;121:592-598. [PubMed] |
| 5. | Tada K, Oka M, Hayashi H, Tangoku A, Oga A, Sasaki K. Cytogenetic analysis of esophageal squamous cell carcinoma cell lines by comparative genomic hybridization: relationship of cytogenetic aberrations to in vitro cell growth. Cancer Genet Cytogenet. 2000;117:108-112. [DOI] |
| 6. | Tada K, Oka M, Tangoku A, Hayashi H, Oga A, Sasaki K. Gains of 8q23-qter and 20q and loss of 11q22-qter in esophageal squamous cell carcinoma associated with lymph node metastasis. Cancer. 2000;88:268-273. [DOI] |
| 7. | Bektas N, Donner A, Wirtz C, Heep H, Gabbert H E, Sarbia M. Allelic loss involving the tumor suppressor genes APC and MCC and expression of the APC protein in the development of dysplasia and carcinoma in Barrett, s esophagus. Am J Clin Pathol. 2000;114:890-895. [PubMed] [DOI] |
| 8. | Werner M, Walch A. Morphologie and biologie der invasion am beispiel des barrett-karzinoms. Morphology and biology of invasion with Barret's carcinoma as an example. Verh Dtsch Ges Pathol. 2000;84:62-68. [PubMed] |
| 9. | Dolan K, Garde J, Walker S J, Sutton R, Gosney J, Field J K. LOH at the sites of the DCC, APC, and TP53 tumor suppressor genes occurs in Barrett's metaplasia and dysplasia adjacent to adenocarcinoma of the esophagus. Hum Pathol. 1999;30:1508-1514. [DOI] |
| 10. | Mehlen P, Rabizadeh S, Snipas SJ, Assa-Munt N, Salvesen GS, Bredesen DE. The DCC gene product induces apoptosis by a mechanism requiring receptor proteolysis. Nature. 1998;395:801-804. [PubMed] [DOI] |
| 11. | Watanabe M, Kuwano H, Tanaka S, Toh Y, Sadanaga N, Sugimachi K. Flow cytometric DNA analysis is useful in detecting multiple genetic alterations in squamous cell carcinoma of the esophagus. Cancer. 1999;85:2322-2328. [DOI] |
| 12. | Sarlomo Rikala M, El Rifai W, Lahtinen T, Andersson LC, Miettinen M, Knuutila S. Different patterns of DNA copy number changes in gastrointestinal stromal tumors, leiomyomas, and schwannomas. Hum Pathol. 1998;29:476-481. [DOI] |
| 13. | Varis A, Puolakkainen P, Savolainen H, Kokkola A, Salo J, Nieminen O, Nordling S, Knuutila S. DNA copy number profiling in esophageal Barrett adenocarcinoma: comparison with gastric adenocarcinoma and esophageal squamous cell carcinoma. Cancer Genet Cytogenet. 2001;127:53-58. [DOI] |
| 14. | Hu N, Roth MJ, Emmert-Buck MR, Tang ZZ, Polymeropolous M, Wang QH, Goldstein AM, Han XY, Dawsey SM, Ding T. Allelic loss in esophageal squamous cell carcinoma patients with and without family history of upper gastrointestinal tract cancer. Clin Cancer Res. 1999;5:3476-3482. [PubMed] |
| 15. | van Dekken H, Geelen E, Dinjens WN, Wijnhoven BP, Tilanus H W, Tanke HJ, Rosenberg C. Comparative genomic hybridization of cancer of the gastroesophageal junction: deletion of 14Q31-32.1 discriminates between esophageal (Barrett's) and gastric cardia adenocarcinomas. Cancer Res. 1999;59:748-752. [PubMed] |